IDENTIFICACIÓN MOLECULAR DE POBLACIONES BACTERIANAS ASOCIADAS AL CARACOL PALA (Strombus gigas) DEL CARIBE COLOMBIANO
El caracol Pala, Strombus gigas (Strombidae), es de gran importancia ecológica y socioeconómica en el área caribeña colombiana. Sin embargo, es una especie catalogada como “vulnerable” y existe muy poca información referente a las especies bacterianas asociadas al caracol que puedan ser importantes para el desarrollo, manejo productivo y de seguridad acuícola de estos gastrópodos. En este trabajo, nosotros empleamos un estudio microbiológico y molecular de la región intergénica entre los genes 16S y 23S rDNA, análisis del gen rDNA 16S y secuenciación, para analizar las bacterias asociadas al caracol Pala (S. gigas). La composición de bacterias cultivables asociadas fue evaluada por su capacidad para crecer en agar marino y en medios de cultivos selectivos. De un total de 28 muestras analizadas encontramos que el número de bacterias cultivadas en condiciones aerobias fue de alrededor 106 ufc mL-1 donde las bacterias pertenecientes a la familia Vibrionacea fueron las más abundantes, cerca de >105 ufc mL-1 . El análisis molecular de la región intergénica entre los genes 16S y 23S rDNA de las diferentes muestras, reveló una gran complejidad bacteriana asociada a S. gigas. Las secuencias de los amplificados del gen rDNA 16S identificó Pseudoalteromonas sp., Halomonas sp., Psycrobacter sp., Cobetia sp., Pseudomonas sp. y Vibrios sp. Nuestros resultados podrían sugerir un rol importante de estas bacterias como componentes de la comunidad asociada al S. gigas. Esta información puede complementar los estudios que se están implementando en los procesos para la conservación y repoblamiento de las poblaciones de S. gigas en Colombia.
Palabras clave: Strombus gigas, Caracol pala, Bacteria, Región intergénica 16S-23S, rDNA 16S.
ABSTRACT
The Queen Conch, Strombus gigas (Strombidae), is a species of great ecological and socioeconomic importance in the Caribbean area of Colombia. However, it is currently catalogued as “vulnerable”; there is limited information concerning the bacterial species associated with conch and important in the management of hatcheries for higher productivity and safety of these gastropods. In this study, we used a microbiology and molecular approach using the 16S-23S intergenic region, the 16S rDNA analysis and sequencing to determine the bacterial populations associated with Queen Conch (S. gigas). Also, the capacity to grow in marine agar and selective culture media was used to evaluate the composition of bacteria associated. The 28 total samples analysed we found the number of bacteria recovered after aerobic culture about 106 cfu mL-1 and most belong to the Vibrionaceae family in the order of 105 ufc mL-1. The molecular results of the spacer regions between the 16 and 23S genes from the different analyzed samples indicated a great complexity in the bacterial population associated to S. gigas. The sequencing of the amplicons of 16S rDNA identifies Pseudoalteromonas sp, Halomonas sp., Psycrobacter sp., Cobetia sp., Pseudomonas sp. and Vibrios sp. This suggests these bacteria can play an important role as components of the bacterial community associated to S. gigas. This information can help to improve both the management of hatcheries for higher productivity and for the implementation for the conservation processes of Colombian S. gigas.
Key words: Strombus gigas, Queen Conch, Bacteria, 16S-23S intergenic region, 16S rDNA.
IDENTIFICACIÓN MOLECULAR DE POBLACIONES BACTERIANAS ASOCIADAS AL CARACOL PALA (Strombus gigas) DEL CARIBE COLOMBIANO
Molecular Identification Of Bacterial Populations Associated To Queen Conch (Strombus gigas) From Colombian Caribbe
EDINSON ANDRÉS ACOSTA1, Estudiante Ingeniería Biológica; ELIANA GÓMEZ1, Estudiante Ingeniería Biológica; MAGALLY ROMERO TABAREZ1, Ph. D.; GLORIA ESTER CADAVID RESTREPO1, Ph. D.; CLAUDIA XIMENA MORENO HERRERA1, Ph. D.
1Grupo de Microbiodiversidad y Bioprospección (Microbiop) Laboratorio de Microbiología Industrial y Biología Celular y Molecular. Departamento de Biociencias, Facultad de Ciencias, Universidad Nacional de Colombia, Sede Medellín, Colombia.
Correspondencia: Claudia Ximena Moreno Herrera, Universidad Nacional de Colombia, Sede Medellín. Bloque 21-332. cxmoreno@unalmed.edu.co.
Presentado 15 de diciembre de 2008, aceptado 18 de marzo de 2009, correcciones 2 de julio de 2009.
RESUMEN
El caracol Pala, Strombus gigas (Strombidae), es de gran importancia ecológica y socioeconómica en el área caribeña colombiana. Sin embargo, es una especie catalogada como “vulnerable” y existe muy poca información referente a las especies bacterianas asociadas al caracol que puedan ser importantes para el desarrollo, manejo productivo y de seguridad acuícola de estos gastrópodos. En este trabajo, nosotros empleamos un estudio microbiológico y molecular de la región intergénica entre los genes 16S y 23S rDNA, análisis del gen rDNA 16S y secuenciación, para analizar las bacterias asociadas al caracol Pala (S. gigas). La composición de bacterias cultivables asociadas fue evaluada por su capacidad para crecer en agar marino y en medios de cultivos selectivos. De un total de 28 muestras analizadas encontramos que el número de bacterias cultivadas en condiciones aerobias fue de alrededor 106 ufc mL-1 donde las bacterias pertenecientes a la familia Vibrionacea fueron las más abundantes, cerca de >105 ufc mL-1. El análisis molecular de la región intergénica entre los genes 16S y 23S rDNA de las diferentes muestras, reveló una gran complejidad bacteriana asociada a S. gigas. Las secuencias de los amplificados del gen rDNA 16S identificó Pseudoalteromonas sp., Halomonas sp., Psycrobactersp., Cobetia sp., Pseudomonas sp. y Vibrios sp. Nuestros resultados podrían sugerir un rol importante de estas bacterias como componentes de la comunidad asociada al S. gigas. Esta información puede complementar los estudios que se están implementando en los procesos para la conservación y repoblamiento de las poblaciones deS. gigas en Colombia.
Palabras clave: Strombus gigas, Caracol pala, Bacteria, Región intergénica 16S-23S, rDNA 16S.
ABSTRACT
The Queen Conch, Strombus gigas (Strombidae), is a species of great ecological and socioeconomic importance in the Caribbean area of Colombia. However, it is currently catalogued as “vulnerable”; there is limited information concerning the bacterial species associated with conch and important in the management of hatcheries for higher productivity and safety of these gastropods. In this study, we used a microbiology and molecular approach using the 16S-23S intergenic region, the 16S rDNA analysis and sequencing to determine the bacterial populations associated with Queen Conch (S. gigas). Also, the capacity to grow in marine agar and selective culture media was used to evaluate the composition of bacteria associated.
The 28 total samples analysed we found the number of bacteria recovered after aerobic culture about 106 cfu mL-1 and most belong to the Vibrionaceae family in the order of 105 ufc mL-1. The molecular results of the spacer regions between the 16 and 23S genes from the different analyzed samples indicated a great complexity in the bacterial population associated to S. gigas. The sequencing of the amplicons of 16S rDNA identifiesPseudoalteromonas sp, Halomonas sp., Psycrobacter sp., Cobetia sp., Pseudomonas sp. and Vibrios sp. This suggests these bacteria can play an important role as components of the bacterial community associated to S. gigas. This information can help to improve both the management of hatcheries for higher productivity and for the implementation for the conservation processes of Colombian S. gigas.
Key words: Strombus gigas, Queen Conch, Bacteria, 16S-23S intergenic region, 16S rDNA.
INTRODUCCIÓN
Los animales portan una gran cantidad de microorganismos que puedan llegar a formar comunidades complejas. Se conoce como microflora normal a aquella población de microorganismos que no causa daños al hospedero, a pesar de estar en continuo contacto con sus tejidos (Backhed et al., 2004). A la microflora se le atribuyen efectos benéficos para el hospedero, como en nutrición complementando procesos de digestión, contribuyendo al metabolismo o jugando un papel protector al prevenir la colonización por patógenos, controlando su crecimiento y modulando el sistema inmune del hospedero (Kesarcodi et al., 2007).
En Colombia como en otros países la explotación de extracción o cultivo de alimentos marinos en sus aguas costeras, está continuamente amenazada por la aparición de microorganismos marinos patógenos tanto para los alimentos marinos (mariscos) como para el consumidor (Sapkota et al., 2008.). Un alimento marino de importancia no solo económica y cultural en Colombia es el caracol Strombus gigas. Actualmente la pesquería se ejerce de manera comercial y en aproximadamente 25 países y territorios dependientes del área de distribución de la especie (Theile, 2001). El caracol S. giga denominado caracol Pala en Colombia (Randall, 1964), ha sido recientemente incluido en Libro Rojo de Invertebrados Marinos de Colombia en la categoría “vulnerable”. La sobrepesca, la pérdida de importantes hábitats de cría (como prados de pastos marinos), actividades humanas como la urbanización, la contaminación, y otros aparejos destructivos son causas del declive poblacional (Glazer y Quintero, 1998). De acuerdo a la información del Instituto Colombiano Agropecuario (ICA), los volúmenes de exportación de recursos como la langosta espinosa, la Cherna criolla y el caracol Pala, en la actualidad son de alrededor de 200 toneladas anuales y el 90% es enviado hacía el mercado norteamericano, que le representan a la economía colombiana ingresos superiores a los diez millones de dólares anuales.
La especie es dioica y la reproducción generalmente ocurre en los meses más calurosos, aunque, en algunas áreas puede continuar a lo largo de todo el año (Brownell, 1977). Las larvas conocidas como velígera son pelágicas, eclosionan a los cinco o seis días del desove (D’Asaro, 1965) y flotan en las corrientes de las capas superiores de las aguas propiciando el intercambio larval a distancias de hasta 900 km durante las primeras tres semanas (Davis et al., 1984). Las larvas pueden establecerse en los hábitats bentónicos entre los 17 y los 22 días después de la eclosión, aunque pueden permanecer en el plancton hasta por dos meses (Posada y Appeldoorn, 1994; Stoner, 1997). Se ha planteado que existe una estrecha relación entre la nutrición principalmente por algas las cuales forman una asociación con bacterias que estarían implicadas en la metamorfosis del caracol (Aldana-Aranda y Patiño, 1998; Davis y Stoner, 1994). Los juveniles (< 1 año) permanecen enterrados la mayor parte del tiempo, posiblemente para evitar la depredación y empiezan a salir cuando la longitud de la concha es entre 5 y 10 cm, en esta etapa sufren una alta mortalidad (hasta un 63%) por depredación (Alcolado, 1976). La madurez sexual normalmente tiene lugar entre los cuatro y cinco años de edad, cuando el labio acampanado se ha engrosado a aproximadamente 0,5 cm (Appeldoorn, 1988; Appeldoorn y Rodríguez, 1994; Chiquillo et al., 1997). La especie tiene una longevidad entre 20 y 30 años (Anon, 1999).
Dentro de las alternativas para recuperar las poblaciones sobre explotadas de S. gigas están las áreas marinas protegidas (AMP’s; Anon, 1999; Appeldoorn y Rodríguez, 1994; Stoner, 1997) porque permiten la preservación de planteles de desove a densidades altas y mantienen un refugio de los adultos con mayor capacidad de reproducción (Anon, 1999). Otra alternativa es la acuicultura con técnicas orientadas a producir juveniles (Brownell, 1977; Creswell, 1994), o el semicultivo que se plantea como una buena alternativa de manejo la cual está en estudio para establecer si es factible para el redoblamiento (Márquez et al., sometido). Estas alternativas deben comprometer como parte fundamental para su éxito, el estudio de todos los aspectos relacionados con el cultivo y evaluar la eficacia de las estrategias de manejo que se proponen para conservar y restaurar poblaciones del caracol, incluyendo el estudio de la microbiota que se ha mostrado relacionada de manera directa en el crecimiento y desarrollo de este recurso (Kirjavainen y Gibson 1999; Davis et al., 2004).
Es poca la información existente sobre la flora asociada al caracol y su relación con su desarrollo. La flora bacteriana en caracol ha sido descrita a través de caracterización fenotípica de cepas aisladas por técnicas cultivo-dependiente, describiendo géneros bacterianos como Pseudomonas, Acinetobacter, Aeromonas, Vibrio,con un predominio de la presencia de Vibrio extorquens y Pasteurella sp. Estas bacterias corresponderían tanto a microflora normal como a bacterias presentes en la alimentación del caracol. (Ducklow et al., 1979) también se han descrito comunidades de cianobacterias ensambladas a los compuesto carbonatados del caracol (Chacón et al., 2006). Evidencias del rol de la microbiota ha sido investigada en diversos organismos marinos, donde se destaca en importantes procesos de estimulación de la proliferación celular, promoción del metabolismo de nutrientes, respuesta inmune, entre otras (Rawls et al., 2004; Ringo et al., 2006; Hovda et al., 2007). Una mezcla compleja de factores ambientales perfiles microbiológicos y manejo de prácticas influencian el éxito de los ciclos de producción de organismos marinos (Kennedy et al., 2006). La composición de la microflora en un determinado hospedero, probablemente depende de sus propiedades, su historial de contactos con bacterias colonizadoras, su dieta y las condiciones ambientales que prevalecen en su hábitat (Kirjavainen y Gibson, 1999). Algunas bacterias establecen asociaciones a organismos superiores, proporcionándoles una mejor adaptación al medio en el que se encuentran, como son las relaciones simbióticas (Nyholm et al., 2004). Sin embargo, bacterias de la flora normal pueden en algunos casos ser patógenos oportunistas, siendo involucrados en el declive poblacional en los sistemas de cultivo (Tonguthai, 1995; Kaspar y Tamplin, 1993). Actualmente, la disponibilidad de métodos para la detección y caracterización genética de bacterias en forma independiente de su habilidad de crecer en medios de cultivo, ofrece la oportunidad de estudiar la microflora bacteriana de diferentes especies animales, incluyendo los caracoles (Amman et al., 1995; Wagner et al., 1994). Los avances en biología molecular han permitido diseñar nuevas técnicas para la identificación de bacterias utilizando métodos basados en la determinación de las secuencias de DNA y han permitido establecer relaciones filogenéticas principalmente usando la amplificación y secuenciación de los genes ribosomales (Giovannoni et al., 1990). En este estudio, nosotros usamos el análisis de genes ribosomales para determinar las bacterias asociadas al caracol Pala. La composición bacteriana fue determinada por el análisis de la región intergénica entre los genes 16S y 23S rDNA, del gen del 16S rDNA y secuenciación. Los resultados de los espaciadores amplificados del DNA extraído directamente de la muestra fueron comparados para obtener una mayor comprensión de las comunidades presentes en las muestras del caracol Pala. El propósito general de este proyecto fue el estudio de poblaciones bacterianas de muestras de la baba e intestino de caracol Pala silvestre y en cautiverio. También analizamos los estanques donde se cría los caracoles y el alimento, para explorar la influencia del ambiente y la búsqueda específica de bacterias del géneroVibrio. En nuestro conocimiento, el presente trabajo es el primer estudio de las bacterias asociadas a S. gigasusando análisis molecular de genes ribosomales.
MATERIALES Y MÉTODOS
OBTENCIÓN DE LAS CEPAS
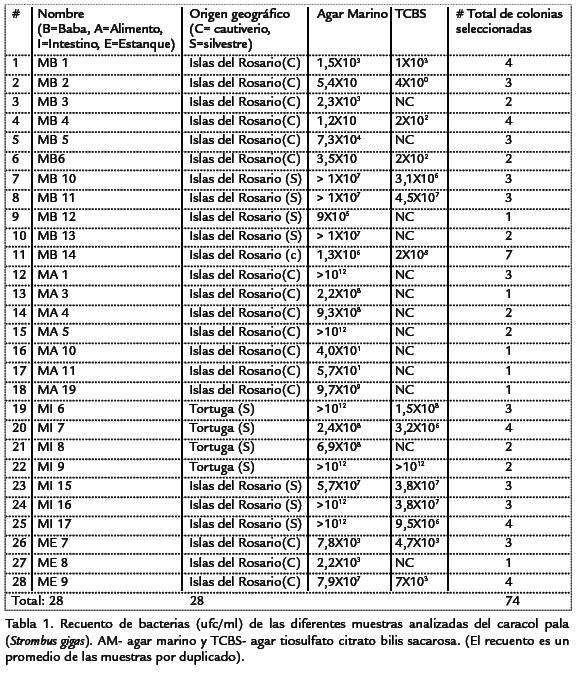
Durante junio y octubre de 2007 se recolectaron las muestras de baba y de intestino de caracoles silvestres y en diciembre 2007 se recolectaron muestras de baba, alimento (algas de los géneros Isocrysis y Chaetoceros) y de las paredes de los estanques donde se encuentran los caracoles en cautiverio (Tabla 1). El muestreo se realizó con la colaboración del proyecto Colciencias titulado “Estructura Genética Poblacional y Zoocría del Caracol PalaStrombus gigas, para Repoblamiento de Habitats Naturales del Caribe Colombiano”. Se recolectaron 28 muestras para ser procesadas: 11 a partir de baba de caracol (cinco caracoles silvestres y seis en cautiverio), siete muestras de tejido intestinal, tres a partir de las paredes de los estanques y siete del alimento donde se encontraban cultivados los caracoles. Las muestras fueron transportadas a 4 ºC hasta su análisis en el laboratorio, donde fueron procesadas. Las muestras de baba se obtuvieron colocando los caracoles sobre una superficie de vidrio estéril y el rastro de la baba fue colectada para cada caracol, para las muestras de alimento se recolectó 1 L de la muestra en un recipiente estéril, la muestra fue filtrada y resuspendida en 25 mL de agua estéril y enviadas al laboratorio para su procesamiento, la muestras de estanque fueron recolectadas removiendo de las paredes con asas y aplicadores de algodón y esparcidos sobre medios de cultivo para el transporte hasta el laboratorio. Las muestras de intestino fueron recolectadas asépticamente sin incluir el estomago y el ano. 0,5 g de tejido de la parte media del intestino fue lavada con agua estéril, fragmentada y depositada en los medios de transporte para su procesamiento. En el laboratorio el material fue resuspendido en 1 mL de agua estéril y esparcido usando copos de algodón y asas estériles sobre medios que contenían agar marino (DifcoTM), agua peptonada y agar de tiosulfato-citrato-sales biliares (TCBS), diluciones seriadas de cada muestra fueron esparcidas sobre cada medio de cultivo y luego incubadas por dos o tres días a 20 ºC y 37 ºC en aerobiosis.
CULTIVO Y CARACTERIZACIÓN MICROBIOLÓGICA
El recuento de colonias se realizó luego de dos y tres días, el número de unidades formadoras de colonias (ufc) fue calculado por mL para las diferentes muestras analizadas en agar marino (AM) y en el medio de agar tiosulfato citrato bilis sacarosa (TCBS). Las cajas que contenían alrededor de 50-200 colonias fueron seleccionadas para los análisis. Una selección al azar de 74 colonias de las muestras fueron subcultivadas para obtener cultivos puros y caracterizados usando las siguientes pruebas, como lo describe Farto et al., 1999: tinción de Gram, morfología celular, motilidad, metabolismo de la glucosa fermentativo/oxidativo(O/F), crecimiento sobre TCBS y agar marino (DifcoTM), voges proskauer, indol, rojo de metilo, lisina Moeller, citrato, úrea, (sim) gelatina, esculina, reducción de nitrato, crecimiento en solución de NaCl 4%, sacarosa y arabinosa. Las placas fueron incubadas aeróbicamente a 20 ºC y 37 ºC. Adicionalmente, para algunas cepas seleccionadas se efectúo una caracterización microbiológica por API20E y Biolog Inc. (Thompson et al., 2004).
Se consideraron como presuntivos miembros del género Vibrio las cepas que fueron Gram negativas, de forma cocobacilar, mótiles, con metabolismo fermentador de glucosa y con crecimiento sobre TCBS. Las ufc en el medio selectivo TCBS fueron purificadas y sembradas en medio de conservación para vibrios (3 g de extracto de levadura, 10 g de peptona, 20 g de NaCl, 10 g agar y 1.000 mL de agua) para su preservación. El análisis estadístico fue realizado para determinar diferencias entre las poblaciones cultivables desde las muestras procedentes de caracoles silvestres y en cautiverio en los medios de agar marino y TCBS usando el MINITABsoftware (Minitab Inc., Pennsylvania, USA).
ANÁLISIS MOLECULAR
Extracción del DNA. Para la caracterización molecular se realizó la extracción del DNA de las diferentes muestras originales recolectadas del caracol para una extracción directa (muestras originales), partiendo de aproximadamente 5 mL de baba, 0,5 g de intestino, 20 mL del filtrado del alimento y del medio que contenía las muestras de estanque, del total de las colonias obtenidas en los medios de cultivo (alrededor de 200 ufc, para la fracción cultivable) y de 60 colonias aisladas. El DNA fue extraído y purificado mediante una dilución en 8% P/V de TE 10X (0,1 M Tris, 0,01 M de EDTA, pH 8,0), 0,5 M NaCl, tratamiento con SDS al 1% e incubación de 20 min a 70 °C, el lisado fue tratado con una solución de fenol/cloroformo/alcohol isoamílico (25:24:1) seguido por precipitación con etanol. El DNA extraído fue resuspendido en 50 µL de agua ultra pura. Se les realizó tratamiento con RNasa (Fermentas) a 1 µg/µL durante 2 h a 37 ºC (Romero et al., 2002).
Iniciadores y amplificación por PCR (Reacción en cadena de la polimerasa). Todos los oligonucleótidos usados en este estudio fueron sintetizados por Gentech (Inc.). La amplificación de la región espaciadora entre el 16S-23S rDNA se realizó con los iniciadores universales de la región conservada del extremo 3’ y 5’ del 16S y 23S rDNAs, respectivamente de eubacterias. El par de iniciadores corresponden a L1 (CAAGGCATCCACCGT GT) y G1 (GTGAAGTCGTAACAAGG) (Jensen et al., 1993) y para la región del 16S rDNA con los iniciadores Eubac 27F (AGA GTT TGA TCC TGG CTC AG), 1492R (GGT TAC CTT GTT ACG ACT T) y 1100R (GGG TTG CGC TCG TTG). Aproximadamente 25 ng/ µLdel DNA bacteriano fue usado para la reacción en un volumen final de 30 µL, la mezcla y el programa de la reacción fueron los descritos por Moreno et al., 2002. El producto de amplificación fue analizado por una electroforesis en un gel de agarosa al 1,8% teñido con un marcador fluorescente de visualización instantánea EZ.VisionTM (AmrescoR) según las recomendaciones del fabricante. Las bandas de DNA fueron visualizadas por iluminación con LUV y fotografiadas por Sistem UV-Transiluminator (Biometra).
Caracterización molecular. La caracterización de los productos de las muestras con extracción directa (muestras originales), fracción cultivable (total de bacterias crecidas en agar marino) y de las colonias aisladas, se realizó mediante la amplificación de la región espaciadora entre los genes del 16S-23S rRNA (ITS) usando los partidores L1 y G1 descritos por Jensen et al., 1993. Los fragmentos resultantes fueron analizados por electroforesis en geles de poliacrilamida y teñidos con nitrato de plata (Moreno et al., 2002). Para el análisis de restricción de los productos de amplificación de 16S rDNA fueron digeridos directamente con 1,5 U de las enzimas de restricción /AluI o /HaeIII (Gibco) durante 3 h a 37 ºC. El producto de amplificación fue analizado por una electroforesis en un gel de agarosa al 1,8% teñido con un marcador fluorescente de visualización instantánea EZ visiónTM(AmrescoR) según las recomendaciones del fabricante. Las bandas de DNA fueron visualizadas por iluminación con LUV y fotografiadas por Sistem UV-Transiluminator (Biometra).
Secuenciación de los 16S rDNA de cultivo puro. Un aislado obtenido de cada grupo de ITS fue identificado por la secuenciación parcial del 16S rDNA. Los aislados fueron amplificados usando PCR y los partidores Eubac 27F y 1100R del 16S rDNA descritos por DeLong, 1992. Los productos de amplificación del 16S rDNA fueron purificados y enviados a secuenciar en ambas direcciones (con los partidores 27F y 1100R) por Macrogen Inc. La relación filogenética de los productos secuenciados en este estudio, fue determinada por comparación de secuencias individuales del gen 16S rRNA publicadas en el banco de bases del servidor BLAST del National Centre for Biotechnology Information (NCBI) y en el rRNA Database Project (RDP II).
RESULTADOS
ANÁLISIS MICROBIOLÓGICO
Recuento de bacterias. Todas las muestras fueron procesadas para estimar el número total de colonias en agar marino y el recuento de Vibrio (TVC) en TCBS, usando la técnica de recuento en placa. Se hicieron diluciones seriadas de las muestras en agua peptonada y el recuento bacteriano sobre agar marino de las diferentes muestras analizadas tanto de los caracoles silvestres como en cautiverio, estuvieron en un rango de 101 -1012ufc. mL-1 (ver Tabla 1 ). El análisis estadístico para determinar diferencias entre las poblaciones cultivables desde las muestras procedentes de caracoles silvestres y en cautiverio determinó que hay diferencias significativas con un recuento total mayor en el medio de agar marino (alrededor de 107 ufc) para las muestras de baba e intestino de caracoles silvestres. Los resultados del recuento de Vibrios (TVC) en TCBS, se muestra en la Tabla 1, y se observó una diferencia significativa entre las muestras asociadas al caracol Pala con un mayor recuento de Vibrio(TVC) en los caracoles silvestres (>106 ufc), exceptuando en dos muestras que no presentaron ningún crecimiento (MB12-13; Tabla 1). El resultado del TVC podría sugerir que la presencia de bacterias cultivables pertenecientes a la familia Vibrionacea, es uno de los grupos bacterianos más abundantes en las muestras analizadas; (P-value = 0,0385 para el medio agar marino y 0,0068 para medio TCBS), los datos fueron analizados usando MINITAB software con la prueba Mann-Whitney Test.
Caracterización de cultivos puros. Del total de colonias crecidas en agar marino se analizaron 74 colonias por sus características de color y forma (Tabla 1). Las colonias subcultivadas y purificadas fueron probadas por tinción de Gram, reacción de catalasa y oxidasa. De las 74 colonias 20 fueron seleccionadas para un análisis por API20E y Biolog, en el resultado de estos dos análisis no se identificó ninguno de los aislados con una discriminación satisfactoria.
Análisis molecular. Aparte del análisis morfológico se realizó una caracterización para determinar la composición de poblaciones bacterianas por análisis molecular de las muestras con extracción directa (muestras originales), la fracción cultivable (poblaciones crecidas en agar marino) y de una selección de 60 colonias aisladas de las muestras del caracol. Se determinó un perfil molecular utilizando la amplificación de la región espaciadora entre los genes del 16S y 23S rRNA y finalmente el análisis de secuencia del amplificado del gen 16S rRNA. Los resultados del análisis de RFLP del rDNA 16S (AluI y HaeIII) de las muestras de baba de caracoles y su relación con algunos perfiles de digestión de los aislados obtenidos, no arrojó diferencias claras (datos no se muestran), por lo cual se optó por una herramienta más resolutiva como los análisis de espaciadores entre los genes del 16S y 23S rRNA (ITS).
Análisis de la región espaciadora entre los genes del 16S y 23S rRNA (ITS). El análisis de ITS mostró que entre los amplicones de los diversos DNA (de extracción directa, fracción cultivable y de colonias aisladas) se observaban bandas que coincidían en migración y otras que presentaban migraciones diferentes. Se observó que los amplificados de las muestras con extracción directa todas presentaron un patrón diferente entre si, indicando la diversidad de poblaciones bacterianas asociadas a las muestras de caracol (Fig. 1 y Fig. 2); carril OB5-6, OI7-8; OA1-2 y OE1

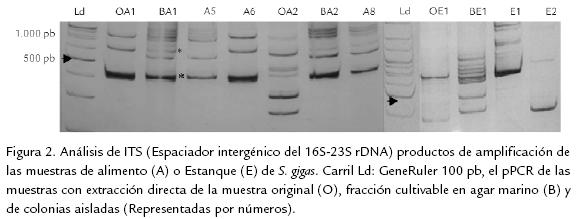
Los perfiles de los espaciadores amplificados generaron un patrón de aproximadamente 10 bandas entre 250 pb y 1.500 pb, el cual fue usado para el análisis y se observó que los espaciadores de aproximadamente 380 y 600 pb (*) estuvieron presentes en todas las muestras analizadas (Fig. 1; Fig. 2; Fig. 3). Sin embargo, se observan otras bandas más tenues que podrían indicar una mayor diversidad en la composición de la microbiota de acuerdo a los diferentes perfiles de ITS observados. La Figura 1 muestra los amplificados de las muestras del caracol pala por extracción directa (O), fracción cultivable (B) y de colonias aisladas de baba (B; carriles OB, BB y B respectivamente), para muestras procedentes de intestino (carriles OI, BI, e I) y en la Figura 2 de alimento y estanque (carriles OA, BA, OE, BE, A y E para amplicones de colonias individuales respectivamente).
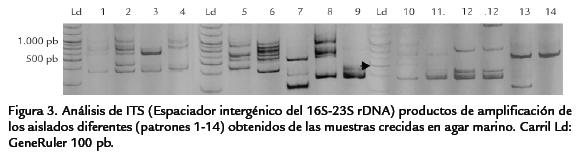
El análisis de los resultados a partir de los productos de PCR realizados al DNA de las colonias seleccionadas, identificaron aproximadamente 12 patrones diferentes de los espaciadores (Fig. 3; carriles 1 al 11 y 14), identificando las diferencias de las bandas principalmente alrededor de los 0,6 kb. Podemos destacar que los patrones de 4, 7; 9 y 10 estuvieron presentes en todas las muestras analizadas, principalmente en la fracción cultivable representando el 50 % (30 ufc) de los ITS de las 60 colonias analizadas, tanto en caracoles silvestres como en cautiverio. Los resultados del análisis de la secuencia de estos patrones se muestran en la Tabla 2. Cabe anotar que el 40% los patrones de ITS de las colonias aisladas de muestra de estanque eran semejantes a los patrones de las colonias de intestino (Fig. 2; carril E2; Fig. 3; carril 14), que fueron identificados por secuenciación como Psycrobacter sp. (Tabla 2).

El análisis de los patrones de la región íntergénica del 16S-23S rDNA para cada uno de los aislados desde TCBS, logró identificar 5 patrones diferentes de espaciadores, incluidos dentro de las colonias analizadas de agar marino (Fig. 3; carriles 3, 5, 6, 8, y 10), mostrando predominio y diversidad de bacterias pertenecientes a la familia Vibrinacea en las muestras tanto de caracoles silvestre y en cautiverio. Estos datos fueron confirmados por los ensayos de secuenciación. Veinticinco colonias seleccionadas por su patrón de ITS, fueron analizadas filogenéticamente por secuenciación. Los resultados de la identidad de las secuencias se muestran en la tabla 2.
DISCUSIÓN
En este estudio, se analizó la composición de bacterias en diferentes muestras asociadas con el caracol pala S. gigas del Caribe colombiano, usando métodos de cultivo y métodos moleculares para obtener un contexto general de las bacterias que podrían estar asociadas con el caracol pala, especie en peligro de extinción. Las secuencias encontradas revelaron miembros dominantes de alpha- y gamma proteo-bacterias, donde las familias: /pseudoalteromonadaceae/, /Moraxellaceae/, /Vibrionaceae/, /Holomonadaceae/, /Pseudomonadaceae/, fueron las mas abundantes en todas las muestras analizadas, luego de la secuenciación parcial del 16S rDNA (900 nucleótidos) con porcentajes de similitud superiores al 98%. Estos resultados son correspondientes con los estudios que reportan a las gamma proteobacterias como los mayores contribuyentes de la comunidades bacterianas identificados en diferentes ambientes marinos (Griffiths et al., 2001; Kennedy et al., 2006; Schulze et al., 2006; Hovda et al., 2007; Navarrete et al., 2008). Los estudios han incluido métodos moleculares como PCR-DGGE que identifican a las gamma proteobacteria (Marinomonas sp.,Pseudoalteromonas sp., Pseudomonas sp. y Vibrio sp.) y en un porcentaje mucho menor bacterias Gram positivas y alpha proteobacterias (Roseobacter sp. y Sulfitobacter sp.). El recuento bacteriano sobre agar marino en nuestros resultados mostró que hay diferencias significativas de crecimiento en los medios de cultivo a partir de las muestras tanto de los caracoles silvestres como en cautiverio, donde el 55% de las colonias analizadas resultaron ser Gram negativas y el otro 45% Gram positivas (resultados no se muestran), lo que en medida confirma los reportes sobre la abundancia de especies bacterianas del género Gram negativo en organismos marinos (Schulze et al., 2006). Algunos autores han sugerido que las bacterias de los estanques o del alimento pueden sobrevivir y multiplicarse en el tracto digestivo de los animales marinos (Olafsen, 2001). En nuestro estudio encontramos que la mayoría de los grupos bacterianos estaban presentes en todas las muestras, Pseudoalteromonas sp., Halomonas sp., Psycrobacter sp., Cobetia sp. y Vibrios; excepto el grupo de Pseudomonas sp., que fueron cultivados a partir de las muestras del alimento. El espaciador de 650 pb que identifica a este grupo de bacterias, se detectó muy tenue, en las muestras con extracción directa de intestino del caracol. Estos resultados sugieren que las Pseudomonas representarían un grupo menor en la fracción cultivable de la microbiota de los caracoles, proporcionada por el alimento; estos datos contribuirían al concepto mencionado por otros autores, donde la composición y diversidad de la microbiota puede depender no solo de la selección pero también de las condiciones locales donde se encuentran los animales marinos (Navarrete et al., 2008). Sin embargo, es importante resaltar que aunque diversos grupos de bacterias puedan estar presentes en las muestras originales, su abundancia puede no ser suficiente para ser detectado por los métodos empleados en este estudio, destacando que la utilización de medios de cultivo es una limitante que puede representar solo una parte de la diversidad bacteriana presente.
En el presente estudio, criterios tradicionales para la identificación morfológica y bioquímica de bacterias fueron usados y fallaron para identificar los aislados bacterianos y no fue posible clasificar en ningún grupo taxonómico. La ineficacia de los sistemas API o Biolog, para la identificación de muestras ambientales ya ha sido discutida por varios autores (Popovic et al., 2007; Hovda et al., 2007), en nuestro estudio se emplearon técnicas moleculares que permitieron una identificación parcial de las bacterias (Tabla 2). Los resultados de las muestras que presentaron crecimiento en el medio TCBS, indicaron que las bacterias pertenecientes a la familia Vibironacea, son dominantes, resultado que se confirmó por el análisis molecular de las colonias analizadas. Estos resultados sugieren que bacterias del género Vibrio podrían tener un rol como componentes de la microbiota asociada al caracol Pala, varios artículos han discutido la importancia del género Vibrio en acuicultura (Vandenberghe et al., 2003; Thompson et al., 2004). Este trabajo es el primer reporte con métodos moleculares de bacterias asociadas a la baba e intestino del caracol S. gigas.
Esta información y otros estudios sobre la microbiota aplicando métodos moleculares adicionales, podrían beneficiar y complementar los estudios que se están implementando de procesos para la conservación y repoblamiento de las poblaciones de caracol Pala en el Caribe colombiano.
AGRADECIMIENTOS
Agradecemos a la profesora Edna Márquez por su ayuda y colaboración dentro del proyecto COLCIENCIAS denominado “Estructura Genética Poblacional y Zoocría del Caracol Pala Strombus gigas, Para Repoblamiento de Habitats Naturales del Caribe Colombiano”, en San Andrés, Providencia y Santa Catalina, islas del Rosario y San Bernardo. Este trabajo fue financiado por el proyecto DIME 6574 (2007) de la Universidad Nacional de Colombia, Sede Medellín.
BIBLIOGRAFÍA
ALCOLADO PM. Crecimiento, variaciones morfológicas de la Concha y algunos datos biológicos del cobo Strombus gigas L. (Mollusca, Mesogastropoda). Acad Cien Cuba Ser Oceanol. 1976;34-36.
ALDANA-ARANDA D, PATIÑO SV. Overview of diets used in larviculture of three Caribbean Conchs: Queen ConchStrombus gigas, Milk Conch Strombus costatus and Fighting Conch Strombus pugilis. Aquaculture. 1998;167:163-178.
AMMAN RI, LUDWIG W, SCHLEIFER KH. Phylogenetic identification and in-situ detection of individual microbial-cells without cultivation. Microbiol Rev. 1995;59:143-169.
ANON. Report on the /Queen Conch/ Stock Assessment and Management Workshop. Belize City, Belize, 15-22 March 1999. Caribbean Fisheries Management Council and CARICOM Fisheries Resources Assessment and Management Programme. http://www.strombusgigas.com/belizeConchWGreportFinal. pdf.
APPELDOORN RS. Age determination, growth and mortality and age of first reproduction in adult queen conchStrombus gigas L., off Puerto Rico. Fish Res. 1988;6:363-378.
APPELDOORN RS, RODRÍGUEZ B. Queen Conch biology, fisheries and mariculture. Fundación Científica Los Roques, Caracas, Venezuela; 1994.
BACKHED F, DING H, WANG T, HOOPER LV, KOH GY, et al. The gut microbiota as an environmental factor that regulates fat storage. Proc Natl Acad Sci USA. 2004;101:15718-15723.
BROWNELL WN. Reproduction, laboratory culture and growth of Strombus gigas, Strombus costatus and Strombus pugilis in Los Roques; 1977.
CHACÓN E, BERRENDERO E, GARCIA PICHEL F: Biogeological signatures of microboring cyanobacterial communities in marine carbonates from Cabo Rojo, Puerto Rico. Sediment Geol. 2006;185:215-228.
CHIQUILLO-ESPITIA E, OSPINA JF, GALLO N. Aspectos biológicos del Caracol Pala Strombus gigas Linnaeus, 1758 (Mollusca: Gastropoda: Strombidae) en el Departamento Archipiélago de San Andrés, Providencia y Santa Catalina (Caribe colombiano). Boletín Científico N.º 5 / Santa Fe de Bogotá; 1997. p. 159-179.
CRESWELL R. An historical overview of queen conch mariculture. In: R.S. Appeldoorn and B. Rodríguez (Eds.): Queen Conch biology, fisheries and mariculture, Fundación Científica Los Roques; 1994.
D'ASARO CN. Organogenesis, development and metamorphosis in the queen conch, Strombus gigas, with notes on breeding habits. Bull Mar Sci. 1965;15:359-416.
DAVIS M, SHAWL A, KOWALIK G, GLAZER R, DELGADO G, EVANS C. Metamorphic response of queen conch (Strombus gigas) larvae exposed to sediment and water from nearshore and offshore sites in the Florida Keys. 2004. http://www.savetheconch.org/research.html.
DAVIS M, STONER AW. Trophic cues induce metamorphosis of Queen Conch larvae (Strombus gigas Linnaeus). J Exp Mar Biol Ecol. 1994;180:83-102.
DAVIS M, MITCHELL BA, BROWN JL. Breeding behaviour of the Queen Conch Strombus gigas Linne held in natural closed environment. J Shellfish Res. 1984;4:17-21.
DELONG EF. Archaea in coastal marine environments. Proc Natl Acad Sci USA. 1992;89:5685-5689.
DUCKLOW HW, BOYLE PJ, MAUGEL PW, STRONG C, MITCHELL R. Bacterial Flora of the Schistosome Vector Snail Biomphalaria glabrata. Appl Environ Microbiol. 1979;38:667-672.
FARTO R, MONTES M, PÉREZ MJ, NIETO TP, LARSEN JL, PEDERSEN K. Characterization by numerical taxonomy and ribotyping of Vibrio splendidus biovar I and Vibrio scophthalmi strains associated with turbot cultures. J Appl Microbiol. 1999;86:796-804.
GIOVANNONI SJ, BRITSCHGI TB, MOYER CL, FIELD KG. Genetic diversity in Sargasso Sea bacterioplankton. Nature. 1990;345:60-63.
GLAZER RA, QUINTERO I. Observations on the sensitivity of Queen Conch to water quality: Implications for coastal development. 50th Proc Gulf Carib Fish Inst. 1998. p. 78-93.
GRIFFITHS S, MELVILLE K, COOK M, VINCENT S. Profiling of bacterial species associated with haddock larviculture by PCR amplification of 16S rDNA and denaturing gradient gel electrophoresis, J Aquat Anim Health. 2001;13:355-363.
HOVDA MB, LUNESTAD BT, FONTANILLAS R, ROSNES JT. Molecular characterisation of the intestinal microbiota of farmed Atlantic salmon (Salmo salar L.). Aquaculture. 2007;272:581-588.
JENSEN MA, WEBSTER JA, STRAUS N. Rapid identification of bacteria on the basis of polymerase chain reaction-amplified ribosomal DNA polymorphisms. Appl Environ Microbiol. 1993;59:945-952.
KASPAR CW, TAMPLIN M. Effects of temperature and salinity on thesurvival of Vibrio vulnificus in seawater and shellfish. Appl Environm Microbiol. 1993;59:2425-2429.
KENNEDY B, VENUGOPAL MN, KARUNASAGAR I, KARUNASAGAR I. Bacterial flora associated with the giant freshwater prawn Macrobrachium rosenbergii, in the hatchery system. Aquaculture. 2006;261:1156-1167.
KESARCODI-WATSON A, HEINRICH K, LATEGAN MJ, GIBSON L. Probiotics in aquaculture: The need, principles and mechanisms of action and screening processes. Aqua. 2007;274:1-14.
KIRJAVAINEN PV, GIBSON G. Healthy gut microflora and allergy: factors influencing development of the microbiota. Ann Med. 1999;31:288-292.
MÁRQUEZ E, LANDÍNEZ-GARCÍA RM, OSPINA-GUERRERO SP. Genetic structure of populations of Queen ConchStrombus gigas from Southwest Caribbean based on microsatellite analysis. Sometido.
MORENO C, ROMERO J, ESPEJO RT: Polymorphism in repeated 16S rRNA genes is a common property of type strains and environmental isolates of the genus Vibrio. Microbiology. 2002;148:1233-1239.
NAVARRETE P, ESPEJO RT, ROMERO J. Molecular analysis of microbiota along the digestive tract of juvenile Atlantic Salmon (Salmo salar L.). Microb Ecol. 2008;75:510-561.
NYHOLM SV, MCFALL-NGAI M. The winnowing: establishing the squid-vibrio symbiosis. Nat Rev Microbiol. 2004;2:632-642.
OLAFSEN JA. Interactions between fish larvae and bacteria in marine aquaculture. Aquaculture. 2001;200:223-247.
POPOVIC NT, COZ-RAKOVAC R, STRUNJAK-PEROVIC I. Commercial phenotypic tests (API 20E) in diagnosis of fish bacteria, a review. Vet Med. 2007;52:49-53.
POSADA JM, APPELDOORN RS. Preliminary observations on the distribution of Strombus larvae in the eastern Caribbean. In: R.S. Appeldoorn and B. Rodríguez (eds.). Queen Conch biology, fisheries and mariculture, Fundación Científica Los Roques, Caracas, Venezuela; 1994. p. 191-200.
RANDALL JE. Contributions to the biology of the queen conch Strombus gigas. Bull Mar Sci Gulf Carib. 1964;14:246-295.
RAWLS JF, SAMUEL BS, GORDON JI. Gnotobiotic zebrafish reveal evolutionarily conserved responses to the gut microbiota. Proc Natl Acad Sci USA. 2004;101:4596-4601.
RINGO E, SPERSTAD S, MYKLEBUST R, REFSTIE S, KROGDAHL A. Characterisation of the microbiota associated with intestine of Atlantic cod (Gadus morhua L.): the effect of fish meal, standard soybean meal and a bioprocessed soybean meal. Aquaculture. 2006;261:829-841.
ROMERO J, GARCÍA-VARELA M, LACLETTE JP, ESPEJO RT. Bacterial 16S rRNA gene analysis revealed that bacteria related to Arcobacter spp. constitute an abundant and common component of the oyster microbiota (Tiostrea chilensis). Microb Ecol. 2002;44:365-371.
SAPKOTA A, SAPKOTA AR, KUCHARSKI M, BURKE J, et al. Aquaculture practices and potential human health risks: Current knowledge and future priorities. Environ Int. 2008;34:1215-1226.
SCHULZE AD, ALABI AO, TATTERSALL-SHELDRAKE AR, MILLER KM. Bacterial diversity in a marine hatchery: Balance between pathogenic and potentially probiotic bacterial strains. Aquaculture. 2006;256:50-73.
STONER AW. The Status of Queen Conch, Strombus gigas, Research in the Caribbean. Mar Fish Rev. 1997;59:14-22.
THEILE S. Queen Conch fisheries and their management in the Caribbean. Technical report to the CITES Secretariat. TRAFFIC Europe, Brussels, Belgium. 2001.
THOMPSON FL, IIDA T, SWINGS J. Biodiversity of vibrios. Microbiol Mol Biol Rev. 2004;68:403-431.
TONGUTHAI K. Diseases of the freshwater prawn, Macrobrachium rosenbergii, The Aquat. Anim Health Res Inst Newsletter. 1995;4:1-4.
VANDENBERGHE J, THOMPSON FL, GOMEZ-GIL B, SWINGS J. Phenotypic diversity amongst Vibrio isolates from marine aquaculture systems. Aquaculture. 2003;219:9-20.
WAGNER MR, ERHART W, MANZ R, et al. Development of an rRNA-targeted oligonucleotide probe specific for the genus Acinetobacter and its application for in situ monitoring in activated sludge. Appl Environ Microbiol. 1994;60:792-800.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2009 Acta Biológica Colombiana

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
1. La aceptación de manuscritos por parte de la revista implicará, además de su edición electrónica de acceso abierto bajo licencia Attribution-NonCommercial-ShareAlike 4.0 (CC BY NC SA), la inclusión y difusión del texto completo a través del repositorio institucional de la Universidad Nacional de Colombia y en todas aquellas bases de datos especializadas que el editor considere adecuadas para su indización con miras a incrementar la visibilidad de la revista.
2. Acta Biológica Colombiana permite a los autores archivar, descargar y compartir, la versión final publicada, así como las versiones pre-print y post-print incluyendo un encabezado con la referencia bibliográfica del articulo publicado.
3. Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
4. Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos institucionales, en su página web o en redes sociales cientificas como Academia, Researchgate; Mendelay) lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).