Caracterización molecular con marcadores RAM
de árboles nativos de Psidium guajava (guayaba)
en el Valle del Cauca1
Hilsy L. Sanabria O.,2 Mario A. García,3 Jaime E. Muñoz4 Héctor A. Díaz5
Compendio | Abstract | Introducción | Materiales y Métodos
Resultados y Discusión | Conclusiones | Bibliografía
Se evaluaron 53 accesiones de Psidium guajava Valle del Cauca, utilizando marcadores microsatélites aleatorios RAM. Los seis primeros usados generaron 74 bandas polimórficas, con pesos moleculares de 100 a 700 pb. El porcentaje de loci Polimórfico en cinco de los primeros resultó ser del 100% (P<0.05) y la diversidad génica de 0.4386. El Análisis de Varianza Molecular (AMOVA) reveló que el 64.51% de la varianza total haplotípica contabiliza para variaciones dentro de transectos. El análisis de clasificación reflejó un agrupamiento que no correspondía a un patrón geográfico, lo cual indica que la especie de guayaba tiene alta diseminación. El análisis conjunto de las características morfológicas y moleculares suministró una caracterización confiable. La variación total de los descriptores morfológicos y valores moleculares fue explicada en un 77.58% mediante CP, donde las variables originales alcanzaron valores de comunalidad desde 57.22 a un 95.99% dentro de cinco variables sintéticas generadas. El ACM permitió caracterizar las accesiones en cuatro grupos, en los cuales según el análisis discriminante casi el 100% de las accesiones quedaron debidamente clasificadas.
Palabras claves: Psidium guajava, accesiones, biodiversidad, marcadores genéticos, análisis molecular, clasificación.
Molecular characteristics of Psidium guajava in the Cauca Valley, Colombia. 53 accessions of Psidium guajava were collected in 9 zones of the Valley of the Cauca. The accessions were characterized molecularly by Microsatellite Amplificated Random RAMs, with the purpose to estimate the diversity and genetic structure that this specie possesses. Six primers were utilized and generated 74 polymorphisms bands with molecular weights from 100 to 700 pb. Five primers resulted to be 100% (P<0.05) polymorphic and the estimated gene diversity was 0.4386. The Analysis of Molecular Variance (AMOVA) showed that the 64.51% of the haplotypic total variance counts for variations whitin transects, while the 35.49% remainder is due to differences among transepts or populations. The analysis of classification reflected a grouping that did not correspond to a geographical distribution evaluated, which indicates that the species of Psidium has a high dissemination. The altogether analysis of the molecular and morphological characteristics supplied a dependable characterization. The total variation of the descriptors morphological and molecular values was explained in a 77.58% by analysis of principal components (PCA), where the original variables showed communal values since 57.22 to a 95.99% within five generated synthetic variables. The MCA, permitted to characterize the 53 accessions in four groups, in which according to the discriminant analysis almost all of the 100% of the accesions remain properly classified.
Keywords: Psidium guajava, accessions, biodiversity, genetic markers, molecular analysis, classification.
La variabilidad genética de las especies se presenta como respuesta evolutiva al interaccionar con diferentes ambientes y afecta la variación de nucleótidos dentro del genoma, provocando cambios en las proteínas. Esta variación puede ocurrir en varios loci de un cromosoma (Bisby & Coddinton, 1995, citado por Gaitán, 2003).
Al caracterizar una especie se está estimando la variabilidad existente en el genoma vegetal de la población de individuos que la conforman, así toda la información codificada por los genes establece su identidad morfológica (Franco e Hidalgo, 2003).
La diversidad genética es la base para la selección de variedades sobresalientes, promisorias o mejoradas dentro de un contexto frutícola en particular. La guayaba, por ser una especie de polinización cruzada o alógama, principalmente dado el sistema predominante de explotación en que se encuentra en el país, se cruza naturalmente con otras variedades dando origen a genotipos diferentes en apariencia, producción y calidad (Lozano et al., 2002).
La diversidad genética de las especies vegetales se ha estudiado directamente a nivel del ADN o indirectamente a través de marcadores moleculares (Pérez y Ramírez, 1999). Los marcadores moleculares de tipo RAM (Random Amplified Microsatellite) son favorables para evaluar la diversidad genética en plantas y animales, muestran la base misma de la variación de los individuos, permiten seleccionar regiones concretas dentro de la molécula de ADN para estudios determinados, el número de polimorfismos detectables es teóricamente ilimitado y permiten analizar toda la información (Zietkiewicz et al., 1994).
Esta investigación tuvo como objetivo principal caracterizar molecularmente con el marcador RAM los árboles nativos de Psidium guajava en el Valle del Cauca. Además realizó el análisis conjunto de la información morfológica (Sanabria et al., 2005) y molecular.
La caracterización de las accesiones de Psidium guajava se realizó en el laboratorio de Biología Molecular de la Universidad Nacional de Colombia Sede Palmira.
Se extrajo el ADN a muestras de hojas jóvenes conservadas en nitrógeno líquido, mediante el kit DNeasy plant (QIAGEN, 2004). Para evaluar la diversidad genética, las muestras de ADN se probaron con 6 “primer” o cebadores sintetizados por Technologies Inc (Tabla 1).
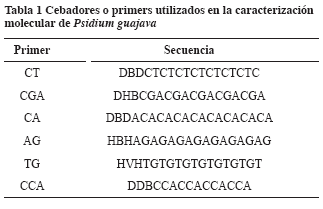
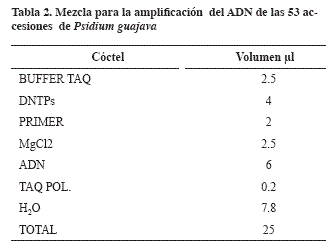
Para la estandarización de las condiciones de cada uno de los cebadores, se preparó una mezcla de reactivos en un tubo estéril de microcentrífuga (1.5 ml) para un volumen final de 25 μl (Tabla2). Los seis primers o cebadores se probaron con las muestras colectadas. La amplificación se produjo en un termociclador PTC-100 Programmable Termal Controller de MJ Research, Inc. La desnaturalización inicial fue de 95 ºC durante 5 minutos; desnaturalización a 95 ºC por 30 segundos. Hibridación a una temperatura de 50ºC (primer AG y CA), 55ºC (primer CCA-TG-CT) y 58ºC (primer GT-CGA) durante 45 segundos, una extensión de 72 ºC por 2 minutos, 37 ciclos desde la desnaturalización a extensión y por último una extensión a 72 ºC durante 7 minutos. El producto amplificado se observó mediante electroforesis en geles poliacrilamida a 160 voltios por 1 hora.
Análisis de diversidad y estructura genética
Para evaluar diversidad genética se estimó la heterocigosidad insesgada y el porcentaje de loci polimórfico al 95% mediante el programa TFPGA® (Tools for population genetic analyses, versión 1.3, 1997). La estructura genética se infirió del análisis de varianza molecular (AMOVA) basado en las frecuencias génicas de las 9 poblaciones como un grupo en dos niveles jerárquicos (entre y dentro de transectos) y la diferenciación entre poblaciones se estimó a través de Fst por pares de poblaciones mediante el programa Arlequín Ver 2000 (Weir y Cockerham, 1984 y Excofier et al., 1992). Las distancias se calcularon mediante las distancias mínimas insesgadas de Nei (1978) y el dendrograma se construyó con el programa TFPGA.
Los “primers” o cebadores generaron 74 bandas polimórficas, un número superior al trabajo de Rueda (2003) y suficientemente confiable para el análisis estadístico (Nybom y Bartish, 2000).
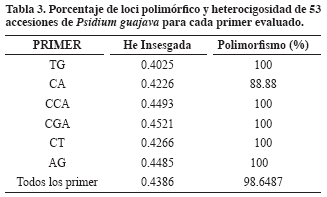
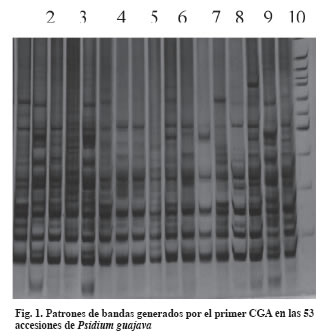
Los “primers” o cebadores fueron altamente polimórficos con valores de 88 al 100 %, igualmente presentaron valores altos de heterocigosidad insesgada, con un valor mínimo de He= 0.4025 para el primer TG y un máximo He = 0.4493 para el primer CGA, la diversidad promedio fue He=0.4386 (Tabla 3 y Fig.1). Poco se conoce de reportes de diversidad genética en guayaba, con excepción de un análisis de diversidad media (Hs), dentro de un grupo de 20 accesiones de Psidium guajava colectadas en América (Rueda, 2003), en el que obtuvo un valor de 0.1975; por tanto puede decirse que existe alta diversidad genética en las accesiones colectadas en el Valle del Cauca.
Estructura y diferenciación genética
El Análisis de Varianza Molecular (AMOVA) reveló que el 64.51% de la varianza total haplotípica contabiliza para variaciones dentro de transectos, mientras que el 35.49% restante se debe a diferencias entre transectos o zonas (Tabla 4).
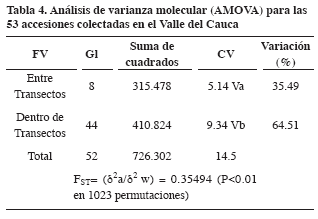
El valor de FST promedio de los nueve transectos 0.3549, mostró una diferenciación genética muy grande entre las zonas de colecta de Psidium guajava evaluados en el Valle del Cauca.
Valores de FST pareados entre transectos mostraron diferencias significativas (P<0.05) entre todas las zonas muestreadas, con excepción de Candelaria y UNAL. No se conoce la historia genética de estas dos últimas poblaciones de guayaba para poder inferir sobre la razón de la no diferenciación genética.
A pesar de que los análisis de estructura y diferenciación genética mostraron alta diferenciación genética entre transectos, no se descarta la existencia del flujo genético entre transectos, debido a que por lo general las semillas son diseminadas por aves o murciélagos frugívoros, monos, ganado, etc. y el hombre (Rodríguez et al., 1991). Por lo anterior, es posible que en el análisis de clasificación a partir de las distancias genéticas de Nei-Li (1978) (Figura 2), algunas accesiones no se agrupen de acuerdo con el origen; no obstante si ha habido diseminación, parece que aún no ha tenido efecto influyente sobre la estructura poblacional. Sin embargo, no existe suficiente información en la literatura para comprobarlo.
Distancia genética
Los valores de distancia genética oscilaron entre 0.0715 ( Restrepo y Ginebra) y 0.2914 (Ginebra y Caicedonia) Estos resultados demuestran que existe variabilidad entre las accesiones colectadas, la cual fue cuantificada en el AMOVA.
A partir de los valores de distancia mínima se construyó un dendrograma (Fig. 3) mediante el método de UPGMA, haciendo uso del programa TPGA, en el cual se agruparon los nueve transectos en tres grupos bien diferenciados. El primer grupo está conformado por Restrepo, Ginebra y Bolo Italia; el segundo por UNAL, Candelaria y CORPOICA (accesiones colectadas a nivel nacional) y el tercero por Jamundí, Caicedonia y Roldanillo. No existe información sobre la dinámica poblacional entre estos transectos que permita inferir sobre su relación genética, así como también los grupos conformados no obedecen a un patrón geográfico definido; sin embargo los resultados de “bootstraping” llevados a cabo con 1000 permutaciones por el programa de TPGA, revelaron que existe una proporción de réplicas similares para cada nudo formado en el árbol que va desde 59.2 a 100%, es decir que la relación entre transectos es bastante significativa.
Correlaciones entre las variables y dimensiones morfológicas y moleculares
Para explicar las correlaciones reveladas en la estadística descriptiva del ACP se relacionó gráficamente la variable grados brix y peso de pulpa del fruto dentro del árbol de distancias entre transectos de la población de Psidium guajava en el Valle del Cauca. Se encontró que además de la diferenciación molecular también existe una diferenciación en relación con los valores morfológicos cuantitativos, que dan lugar a dos grandes grupos con una distancia media según el método UPGMA de 0.2461 (Figura 3). El gran Grupo 2 (accesiones de: Jamundí, Caicedonia y Roldanillo), posee valores altos para grados brix y bajos para peso de pulpa (con la observación que esta variable tiene altas correlaciones con muchas variables de rendimiento del fruto).
El Grupo 1 (Restrepo, Bolo Italia, Ginebra, UNAL, Candelaria y CORPOICA) posee valores moderadamente bajos de grados brix y altos para variables de rendimiento. Sobre la relación entre los datos moleculares y morfológicos cuantitativos se confirmó que entre el número de bandas obtenidas para algunos “primers” versus alguna característica cuantitativa existió una correlación moderada (Tablas 5 y Tabla 6).
Establecer el porqué de la relación y consistencia de la correlación entre el número de bandas leídas por accesión en algunos “primer” con sus valores morfológicos cuantitativos, está fuera de los objetivos de este trabajo; Sin embargo es importante agregar que según Zietkiewicz et al., (1994) los marcadores RAM son secuencias de ADN pequeñas y repetitivas, que pueden estar o no asociadas con genes. Este marcador, según Hantula et al., (1996) en el orden de ser usado como un marcador genético, debe probar su universalidad; por lo cual más estudios deben llevarse a cabo en P. guajava en poblaciones del Valle del Cauca en las que se busquen relaciones de valores morfológicos cuantitativos con los moleculares. Además al igual que como concluye Hantula et al. (1996) si estas bandas pudieran considerarse como loci, y formas detalladas como diferentes alelos, pueden últimamente ser confirmadas por análisis de segregación en trabajos de fitomejoramiento y biología molecular en P. guajava.
Clasificación de las accesiones considerando sus valores morfológicos y moleculares
Según el IPGRI y la Universidad de Cornell (2004) los datos moleculares casi siempre son complementarios a otros datos de caracterización como la de tipo morfológico; y el análisis combinado de esto puede ofrecer un escenario más integral para la interpretación. En tal sentido para obtener la clasificación más apropiada de las accesiones colectadas, se unió la información de la caracterización molecular y morfológica. Cada set de datos moleculares y morfológico cualitativo se transformó desde sus valores binarios y discretos respectivos a dos variables o dimensiones de valores continuos en un análisis de correspondencia múltiple. Las dimensiones formadas por los valores morfológicos cualitativos y moleculares junto con los valores originales de las variables morfológicas cuantitativas se sometieron a análisis de componentes principales y clasificación o clúster.
Análisis de componentes principales (ACP) para valores morfológicos y moleculares
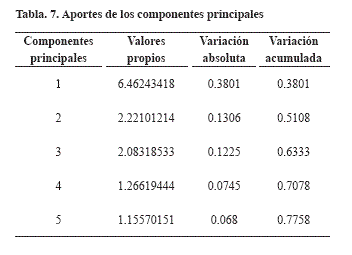
El nuevo ACP proveyó valores más confiables que los expuestos en la caracterización morfológica cuantitativa. Los primeros cinco componentes explicaron una variación acumulada del 77.58% de la variabilidad total de los descriptores evaluados (Tabla 7), y comunalidades (R2) desde un 57.223 a un 95.99% de variación total de cada variable que es explicada por los cinco CP, indicando que estas variables sintéticas que resumen la caracterización tanto morfológica como molecular, suministran suficiente información para resumir los diferentes descriptores y valores moleculares en forma precisa y de esta manera hacer una buena clasificación de las accesiones.
Análisis de clasificación de las accesiones a partir de los datos de la caracterización molecular y morfológica.
La misma matriz considerada para el ACP, que contenía la información de la caracterización morfológica y molecular, se utilizó para el análisis de clasificación. Las diferentes accesiones se clasificaron en 4 grupos (Tabla 8 y Figura 4).
Análisis discriminante (AD)
Para confirmar el agrupamiento brindado por el análisis de clasificación, se llevó a cabo un AD. El análisis discriminante determinó que el 98.11 % de las accesiones estaban debidamente clasificadas. El 1.89 %, es decir solamente 1 accesión (CA4) debía ser reordenada del grupo 4 al 2.
El análisis conjunto de los datos morfológicos y moleculares permitió una clasificación de las accesiones con una precisión casi del 100%, confirmando las relaciones entre accesiones que se dieron independientemente para cada set de datos. En este análisis la clasificación de las accesiones fue congruente con la obtenida en forma individual para los datos morfológicos cuantitativos y moleculares en los cuales se podía agrupar el mayor número de accesiones casi por su zona geográfica o conjuntos de transectos independientes del origen; de tal forma que la mayoría de accesiones de Corpoica formaron siempre un mismo grupo, como también los transectos de Jamundí, Roldanillo y Caicedonia se mostraron muy similares en conjunto, e igualmente Ginebra, Restrepo y algunas accesiones de Bolo Italia. No ocurrió lo mismo para el análisis morfológico cualitativo en el cual las accesiones si bien se agruparon principalmente por la forma del fruto, no hubo clasificación clara por origen, Sin embargo, una de las dimensiones morfológicas cualitativas mostró correlaciones moderadas con la mayoría de las variables morfológicas cuantitativas, lo cual indica que los valores cualitativos pudieron tener un efecto sobre el análisis conjunto que dio lugar al agrupamiento final (Tabla 8)
También se observó que las accesiones de los transectos UNAL y Candelaria no llegaron a formar un grupo definido sino que se distribuyeron dentro de diferentes grupos tanto en el análisis morfológico como en el molecular.
Valores de FST y He obtenidos en el análisis molecular indicaron que existen grandes niveles de diferenciación genética entre la mayoría de los transectos o zonas de colecta de Psidium guajava en el Valle del Cauca, así como también alta diversidad genética, respectivamente.
Los datos moleculares diferenciaron dos grupos de accesiones, que de acuerdo con los valores morfológicos cuantitativos difieren en rendimiento y calidad del fruto.
El análisis conjunto de los datos morfológicos y moleculares permitió una clasificación de las accesiones con una precisión casi del 100% en el cual se formaron 4 grupos de más de un transecto conteniendo casi todas sus accesiones.
Alí, Z; y Lazan, H. Guava. En: Cab International. Postharvest Phisiology and Storage of Tropical and Subtropical Fruits. 1997. pp 145-146.
CORPOICA. 2004. Proyecto guayaba. disponible en http://www.fondohortifruticola.com.co/fichas/tr186.htm
Excofier, L.; Smouse P. E; Quattro, J. M. (1992) Analysis of Molecular Variance Inferred From Metric Distances Among DNA Haplotypes: Application to Human Mitochondrial DNA Restriction Data. Genetics. Vol. 131; p.479-491.
Franco, T.; Hidalgo, R. Análisis estadístico de datos de caracterización morfológica de Recursos Fitogenéticos. Boletín técnico No. 8. Instituto Internacional de Recursos Fitogenéticos (IPGRI), Cali, Colombia, 2003. 89 p.
Gaitán, E. Obtención y uso de secuencias microsatélites ga/ca en estudios de diversidad genética en las especies de palmas colombianas Ceroxylon sasalmae, Ceroxylon alpinum y Attalea amygdalinna Tesis doctoral. Universidad Nacional de Colombia. Palmira, Colombia, 2003 157 p.
Hantula, J; Dusabenyagasani, M; Hamelin, R. 1996. Random amplifield microsatellites (RAMs) a novel method for characterizing genetic variation within Fungi. Eur J For Path. 26: 159-166 pág.
Lozano, J.C.; Toro, J.C.; García, R. y Tafur, R. Manual sobre el cultivo del guayabo en Colombia. Cali, Colombia, 2002. 278 p.
Nei, M. (1972) Genetic distance between populations. Am Nat 106: 83-292.
Nybom; Bartish, I. Effects of life history traits and sampling strategies on genetic diversity estimates obtained with RAPD markers in plants. Perspect Plant Ecol Evol Syst Vol 3/2 (2000); pp 93-114.
Oslinger G, A. 2003. Caracterización molecular de cerdos criollos colombianos mediante la técnica molecular RAM (Random Amplified Microsatellites). Trabajo de grado. Universidad Nacional de Colombia, Sede Palmira.
Pérez, Maolphe ; Balch, E ; Ramírez, R. 1999. Introducción al cultivo de tejidos vegetales, Aguas Calientes, México. Pixelarte, 180 p.
Qiagen, 2004. Dneasy plant mini and maxi handbook (Web: www.Qiagen.com)
Rodríguez, G.; Almaguer, G.; Vargas, T.; Espinoza, J. Psidium guajava L. 1991. Species Plantarum 1: 470. 1753
Rueda, A. 2003. Caracterización molecular del banco de germoplasma de Guayaba Psidium spp. del Centro de Investigación de CORPOICA Palmira Trabajo de grado (Ing.Agr.) Universidad Nacional de Colombia. 65 P.
Sanabria, O., H.L.; García, M.A.; Díaz, H.A.; Muñoz, J.E. Caracterización morfológica en árboles nativos de guayaba en el Valle del Cauca. Acta Agron (Colombia) 54 (4): 1-6. 2005.
Weir, B. S.; Cockerham, C. C., 1984: Estimating F-statistics for the analysis of population structure. Evolution 38: 1358–1370.
Zietkiewicz, E.; Rafalsfi, A.; Laubuda, D. 1994. Genome fingerprinting by simple sequence repeat (ssr) anchored polymerase chain reaction amplification. Genomics 20: 176-183 P.
1. |
Artículo derivado de la tesis de Maestría en Ciencias del primero de los autores.REC.: 08-11-05 ACEPT.: 06-02-06 |
2. |
Profesora –Investigadora de la Universidad Nacional de Agricultura. Honduras, C.A. hlsany@yahoo.com |
3. |
Profesor Asociado. Universidad Nacional de Colombia. Palmira, Colombia. magarciad@palmiraunal.edu.co |
4. |
Profesor Asociado. Universidad Nacional de Colombia. Palmira, Colombia. posgrados@palmiraunal.edu.co |
| 5. | Profesor –Investigador de la Universidad Nacional de Agricultura. Honduras, C.A. hdiaz76@yahoo.com |