
Diagnóstico serológico y molecular del Potato leafroll virus (PLRV) en tubérculos–semilla de papa en Antioquia, Colombia
Serological and molecular diagnosis of Potato leafroll virus (PLRV) in potato tuber–seeds in Antioquia, Colombia
Mauricio Mesa Medina, Martha González Ramírez, Pablo Gutiérrez Sánchez y Mauricio Marín Montoya*
Laboratorios de Microbiología Industrial y Biología Celular y Molecular, Facultad de Ciencias, Universidad Nacional de Colombia sede Medellín, Colombia. *Autor para correspondencia: mamarinm@unal.edu.co
Rec.: 21.05.2015 Acep.: 11.06.2015
Resumen
Las virosis son uno de los problemas fitosanitarios más limitantes para la producción de papa, siendo los virus Potato leafroll virus (PLRV), Potato virus Y (PVY), Potato virus S (PVS) y Potato yellow vein virus (PYVV) los que generan mayores pérdidas económicas en la región andina. Generalmente estos virus son transmitidos por insectos hemípteros y por tubérculos–semilla. En esta investigación se evaluó la presencia y distribución del PLRV en tubérculos de Solanum tuberosum variedad Diacol–Capiro y S. phureja variedad Criolla–Colombia obtenidos en Antioquia, utilizando pruebas serológicas y moleculares. Mediante DAS–ELISA se evaluaron 128 muestras consistentes en tres tipos de tejidos (piel, yemas latentes y brotes), mientras que con RT–PCR en tiempo real (RT–qPCR), se utilizó un subgrupo de 12 muestras de cada variedad. Utilizando DAS–ELISA, el PLRV se encontró en el 3,1% y 10,1% de las muestras de Diacol–Capiro y Criolla–Colombia, respectivamente; siendo evidente una mayor detección del virus en la piel de los tubérculos de ésta última variedad. Las pruebas de RT–qPCR detectaron el PLRV en 83,3% y 91,6% de las 12 muestras de Diacol–Capiro y de Criolla–Colombia, respectivamente; con valores de Ciclo umbral (CT) entre 17,72 y 30,59. La naturaleza de los amplicones obtenidos por RT–qPCR fue confirmada por secuenciación como parte de una región codificante de la cápside del PLRV (Identidad=99%–100%). Estos resultados enfatizan en la necesidad de utilizar tubérculo–semilla certificados por su sanidad viral en los cultivos de papa del país y de fortalecer dichos programas a partir del empleo de técnicas de detección altamente sensibles como RT–qPCR.
Palabras clave: DAS–ELISA, Detección viral, Polerovirus, RT–qPCR, Solanum phureja, Solanum tuberosum.
Abstract
Viral diseases are one of the most limiting phytosanitary problems in the production of potato with Potato leafroll virus (PLRV), Potato virus Y (PVY), Potato virus S (PVS) and Potato yellow vein virus (PYVV) having the biggest economic impact in crops of the Andean region. These viruses are transmitted by hemipteran insects and tubers. In this work, the presence and distribution of PLRV in Solanum tuberosum v. Diacol–Capiro and S. phureja v. Criolla–Colombia tubers was investigated using serological and molecular tests. 128 samples from three types of tissues (skin, dormant buds and sprouts) were tested using DAS–ELISA while a 12–sample subset from each variety was used for real time RT–PCR (RT–qPCR). With DAS–ELISA, PLRV was detected in 3,1% and 10,1% of Diacol–Capiro and Criolla–Colombia samples, respectively; in the latter, PLRV was detected mostly in the tuber skin. PLRV was detected in 83,3% and 91,6% of Diacol–Capiro and Criolla–Colombia samples, respectively, using RT–qPCR. Threshold cycles (Ct) were in the 17,72 to 30,59 range. Sequencing of RT–qPCR fragments resulted in 99–100% nucleotide identity with respect to the PLRV coat protein. These results highlight the need of using certified tuber–seed in potato crops in Colombia and strengthening the seed certification programs using highly sensitive techniques such as RT–qPCR.
Keywords: DAS–ELISA, Polerovirus, RT–qPCR, Solanum phureja, Solanum tuberosum, Virus detection.
Introducción
El Potato leafroll virus (PLRV) (Polerovirus, Luteoviridae), es uno de los virus que generan mayores pérdidas en los cultivos de papa a nivel mundial, con reducciones en los rendimientos que oscilan entre 20 y 60%; incluso cuando se presenta en coinfecciones con otros virus como Potato virus Y (PVY) o Potato virus X (PVX), sus efectos pueden ser aún mayores (Taliansky, Mayo, & Barker, 2003).
Los viriones del PLRV consisten de una cápside icosahédrica de 23–25 nm y un genoma de ARN de cadena sencilla positiva de 5800 a 5900 nt, con extremos 5' y 3' asociados a una proteína VPg y a un grupo OH, respectivamente, (Taliansky, Mayo, & Barker, 2003). Cerca de 16 aislamientos de este virus se han secuenciado completamente, en su mayoría de Europa (Ej. JQ346191, AF453391) y China (Ej. KC456053, KC456054), y tan sólo uno de Suramérica (Ej. AF453392, Perú). El genoma del PLRV presenta nueve marcos de lectura abiertos (ORFs): el ORF 0 codifica para P0, una proteína supresora del silenciamiento de genes, P1 está involucrada en la replicación y produce la VPg por auto–proteolísis; el ORF 2 codifica para la replicasa viral (RdRP), mientras que el ORF 3 lo hace para la cápside viral (CP) de 23 kDa; el ORF 4 codifica para una proteína de movimiento y el ORF 5 para una proteína estructural de 80 kDa, aparentemente relacionada con la transmisión por áfidos. Los ORFs 6 y 7 han sido descritos recientemente y aunque sus funciones no se conocen plenamente, se especula que P7 tiene propiedades de unión a ácidos nucleicos. Finalmente, el ORF8 codifica para la proteína Rap1, involucrada en la replicación del virus (Taliansky et al., 2003; Pooramini, Heydarnejad, & Massumi, 2010). Una característica diferencial del genoma de PLRV es el traslape de sus ORFs, que alcanza cerca de un tercio de su secuencia; así, el ORF 1 se solapa parcialmente con el ORF 0 y completamente con el ORF 8; mientras que el ORF 3 se presenta traslapado con el ORF 4 y el ORF 5 con los ORFs 6 y 7 (Pooramini, Heydarnejad, & Massumi, 2010); (Zarghani, Shams–Bakhsh, Zand, Sokhandan–Bashir, & Pazhouhandeh, 2012).
El PLRV es transmitido por diferentes especies de áfidos de manera persistente no propagativa, siendo los más eficientes Myzus persicae y Macrosiphum euphorbiae; además es fácilmente transmisible por tubérculos–semilla (Agindotan, Shiel, & Berger, 2007). El proceso infectivo de este virus está restringido a los tejidos vasculares del floema y los principales síntomas incluyen enanismos y enrollamiento de hojas, acompañados de una coloración rojiza en el borde los folíolos y finalmente reducciones importantes en el rendimiento (Plchova, Cerovska, Moravec, & Dedic, 2009 ).
En Colombia, estudios recientes realizados por (Gil Ramírez, Cotes Torres & Marín Montoya, 2011), a partir de análisis serológicos han identificado al PLRV como uno de los más incidentes en cultivos de papa, con un promedio de detección del 20%. Adicionalmente, este virus también ha sido detectado en forma generalizada (77,4%) en accesiones de la Colección Central Colombiana (CCC) de S. phureja (Guzmán, Román, Franco, & Rodríguez, 2010), y en cerca del 25% del material de siembra de tamarillo (Solanum betaceum) evaluado por Álvarez, J.A., Cotes, J.M., & Marín, M. , mediante pruebas de DAS–ELISA. Los análisis de secuencias de CP en aislamientos de PLRV de papa y tamarillo indicaron un muy bajo nivel de variación (<4%) entre dichos aislamientos, y entre éstos y aquellos de otros lugares del mundo (Gil Ramírez, Cotes Torres & Marín Montoya, 2011; Álvarez, J.A., Cotes, J.M., & Marín, M. ).
Ya que los estudios de diagnóstico de PLRV en cultivos de papa realizados hasta el momento en Colombia se han restringido a su detección en tejido foliar, en este trabajo se evaluó mediante pruebas de DAS–ELISA la presencia del virus directamente en tubérculos–semilla del Oriente y Norte de Antioquia. Adicionalmente, se evaluó la detección del PLRV mediante RT–PCR en tiempo real (RT–qPCR), con el fin de establecer su utilidad en programas de certificación de tubérculo–semilla en el país.
Materiales y métodos
Obtención de muestras
El estudio se realizó con un total de 16 muestras de tubérculos de S. tuberosum subsp. Andigena var. Diacol–Capiro y 16 muestras de tubérculos de S. phureja var. Criolla–Colombia obtenidas en La Unión (Oriente de Antioquia) y Yarumal (Norte de Antioquia). De cada muestra se seleccionó al azar un tubérculo en estado de brotación y se tomaron con ayuda de escalpelos estériles secciones de aproximadamente 0,5 a 1 cm2 de yemas no brotadas, piel, ápice y/o base de brotes. De esta forma, cada tubérculo estuvo representado por tres tejidos, los que fueron macerados para la detección del PLRV mediante pruebas de DAS–ELISA, para un total de 128 muestras analizadas. Adicionalmente, en 12 muestras seleccionadas al azar de cada variedad, se diagnosticó el virus mediante RT–qPCR.
Pruebas de DAS–ELISA
Las muestras de tejidos de tubérculos de cada variedad se evaluaron con pruebas de DAS–ELISA (Double Antibody Sandwich) con anticuerpos policlonales (Agdia, EE.UU.). Las evaluaciones incluyeron un control negativo (tejido vegetal de papa) y uno positivo (tejido infectado por PLRV) suministrado en forma liofilizada por Agdia y las lecturas de absorbancia a 405 nm fueron realizadas en un equipo Multiscan (Labsystem, Finlandia). Los resultados de estas pruebas fueron analizados siguiendo el procedimiento descrito por la compañía serológica Bioreba (Suiza) (http://www.bioreba.ch/?idpage=6), en el que se define el valor umbral (Cut–off) mediante la formula Umbral = (promedio de absorbancias de lecturas negativas + 3 desviaciones estándar) x 1,1.
PCR y secuenciación
La naturaleza viral del control positivo y de dos de las muestras que resultaron con altos valores de absorbancia en las pruebas de DAS–ELISA fue confirmada por RT–PCR convencional y secuenciación de una región de 330 pb de CP. Las reacciones de RT–PCR se realizaron en dos pasos utilizando los cebadores PLRV–F (5' CGC GCT AAC AGA GTT CAG CC 3') y PLRV–R (5' GCA ATG GGG GTC CAA CTC CAA CTC AT 3') y y PLRV–R (Singh, Kurz, Boiteau, & Bernard, 1995), siguiendo el protocolo descrito por Gil Ramírez, Cotes Torres & Marín Montoya, 2011. Los amplicones del tamaño esperado fueron purificados del gel mediante el kit GeneJET Gel Extraction (Thermo) y secuenciados en ambos sentidos en un equipo ABI Prism 3730xl (PE Applied Biosystems) de la compañía Macrogen (Corea del Sur).
Las secuencias obtenidas fueron comparadas con las bases de datos moleculares del NCBI (http://www.ncbi.nlm.nih.gov/) y utilizadas para realizar un análisis filogenético con respecto a otras depositadas en GenBank. Para esto se alinearon las secuencias con Clustal W y se procedió a la generación del dendrograma haciendo uso del software Mega 6.0 con la metodología Máxima verosimilitud y el modelo Jukes–Cantor (Tamura, Stecher, Peterson, Filipski, & Kumar, 2013)
Detección de PLRV por RT–qPCR
Para un subgrupo de 12 muestras de cada variedad de papa, se extrajo el ARN total utilizando el kit GeneJET Plant RNA Purification (Thermo), siguiendo las instrucciones del fabricante. El ARN obtenido fue eluído en 40 µL de agua tratada con DEPC y determinada su concentración y pureza por lecturas a 260 nm y 280 nm en un equipo Nanodrop 2000C (Thermo).
Las reacciones de RT–qPCR se realizaron en dos pasos utilizando el sistema SYBR Green I y los cebadores PLRV–Sense (5'GCT CAA GCG AGA CAT TCG TG 3') y PLRV–Antisense (5'TTG AAT GCC GGA CAG TCT GA 3') que amplifican un fragmento de 100 pb de CP (Yang, Nie, Liu, & Song, 2013). Las reacciones de retrotranscripción fueron similares a las descritas por Gil Ramírez, Cotes Torres & Marín Montoya, 2011, mientras que la qPCR se realizó con el kit Maxima SYBR Green/ROX qPCR Master Mix (2X) (Thermo), en 25 µL de reacción conteniendo 12,5 µL del kit, 10 µL de agua con DEPC, 0,3 µM de cada cebador y 50–100 ng de ADNc, con un programa de amplificación de 95°C por 10 min, seguido por 40 ciclos de 95°C por 15 s y 52 °C por 45 s, en un equipo Rotor–Gene Q–5plex Platform (Qiagen, Alemania). Todas las reacciones incluyeron un control positivo (ARN extraído de control positivo de Agdia) y un control negativo libre de cDNA viral. La adquisición de fluorescencia se realizó después de cada ciclo de amplificación y los valores de ciclo umbral (Ct) para cada muestra fueron definidos utilizando los valores por defecto del equipo, siendo consideradas como positivas aquellas muestras que superaron el valor Ct antes del ciclo 40. La especificidad de los amplicones se definió por análisis de desnaturalización utilizando la herramienta HRM (High Resolution Melting) entre 50 y 99°C. Adicionalmente, la naturaleza viral de cinco amplicones (incluyendo el control positivo) fue confirmada por secuenciación Sanger en la compañía Macrogen.
Resultados y discusión
Detección de PLRV por DAS–ELISA
En este trabajo el PLRV fue detectado utilizando pruebas de DAS–ELISA en el 3,1% de las 64 muestras procedentes de tubérculos de la variedad Diacol–Capiro; mientras que se encontró en el 10,15% de los tubérculos de Criolla–Colombia, con una notable presencia en la piel de tubérculos (9 de 16 muestras), pero no en las yemas latentes. El uso de la metodología estadística de Bioreba permitió definir un valor umbral de 0,231 como la absorbancia a 405 nm que permite diferenciar las pruebas negativas de aquellas positivas (Figura 1).
La presencia generalizada de este virus afectando cultivos de papa en Colombia ya había sido reportada previamente, pero en tejido foliar. Así por ejemplo, Gil Ramírez, Cotes Torres & Marín Montoya, 2011, al estudiar la incidencia del PLRV en cultivos de Antioquia, Boyacá, Cundinamarca y Nariño, encontraron niveles de infección de entre 13 y 30%, con un promedio nacional del 20%. Así mismo, diferentes estudios realizados en otros países, han señalado repetidamente al PLRV como uno de los virus más incidentes en este cultivo (Taliansky, Mayo, & Barker, 2003; Wang et al., 2011). En Costa Rica, (Vásquez, Montero–Astúa, & Rivera, 2006), realizaron un muestreo de tejido foliar de papa en 30 fincas ubicadas en tres altitudes, analizando mediante ELISA la incidencia de 13 virus, incluyendo el PLRV. Los resultaron identificaron a los virus PVX (77%), Potato aucuba mosaic virus (PAMV) (62%) y PLRV (42%) como los de mayor incidencia en este país, siendo este último detectado en el 80 % de las parcelas de la zona alta y media, y en el 50 % de aquellas de la zona baja. Bostan y Haliloglu (2004), al evaluar mediante ELISA la sanidad viral de cerca de 880 brotes procedentes de tubérculos–semilla utilizados como material de siembra en Turkia, encontraron que el PLRV era uno de los virus más incidentes sobre dicho material, con un nivel de detección del 14,2%. Así mismo, Wang et al. (2011), en su acercamiento y evaluación del estado fitosanitario viral en el cultivo de la papa en China, reportan que después del PVY, el PLRV representa el segundo virus más incidente y limitante de esta agrindustria en dicho país, con un nivel promedio de detección del 22,1% en las siete principales provincias productoras, y pérdidas estimadas en rendimiento que oscilan entre 40 y 60%. Estos resultados reflejan la necesidad manifiesta de fortalecer los programas de certificación de semilla de papa y de facilitar el acceso a este material libre de virus, a pequeños agricultores en Colombia y en otros países cultivadores del mundo. Así mismo, resulta fundamental para este cultivo implementar medidas de manejo dirigidas a disminuir las poblaciones de los vectores del PLRV (Ej. M. persicae y M. euphorbiae) y la eliminación de plantas voluntarias de papa (“toyas”) y de hospedantes alternos solanáceos que sirven de reservorios del virus entre ciclos de siembra de papa.
Confirmación de PLRV por secuenciación
Las reacciones de RT–PCR realizadas con los cebadores PLRV–F y PLRV–R produjeron los fragmentos del tamaño esperado de 330 pb. La comparación de las secuencias obtenidas en el estudio con respecto al GenBank, presentó como primeros ‘hits’ secuencias de aislamientos de PLRV (Ej. accesiones KC456054, AF453392 y JQ346189) con 100% de identidad y cobertura, así como valores e de 3×10–15 a 6×10–16. El árbol filogenético generado presentó un único clado con 100% de soporte de bootstrap y niveles de identidad superiores al 97% entre todas las secuencias evaluadas, incluyendo aquellas de aislamientos procedentes de diferentes países (Ej. Canadá, India, Holanda, Colombia) e incluso hospedantes (Ej. tamarillo y papa) (Figura 2).
Resultados similares han sido encontrados en múltiples estudios tendientes a caracterizar la variabilidad genética del PLRV en el mundo, con niveles de variación máxima del 5% aún entre aislamientos de diferentes continentes y utilizando como base de análisis los ORFs que codifican para CP y PO (ORFs 3 y 0) (Plchova, Cerovska, Moravec, & Dedic, 2009 ; Gil Ramírez, Cotes Torres & Marín Montoya, 2011 ; Zarghani et al., 2012). Dicha situación ha sido atribuida a que posiblemente esta especie viral representa una variante recientemente divergente de un virus ancestral, que se especializó por su infección en plantas de papa. Alternativamente, se ha planteado que las fuertes presiones de selección que ejerce la estrecha base genética de las variedades de S. tuberosum cultivadas en el mundo ha sido el factor determinante para la selección genómica que presenta este virus (Plchova, Cerovska, Moravec, & Dedic, 2009; Gil Ramírez, Cotes Torres & Marín Montoya, 2011 ).
Detección de PLRV por RT–qPCR
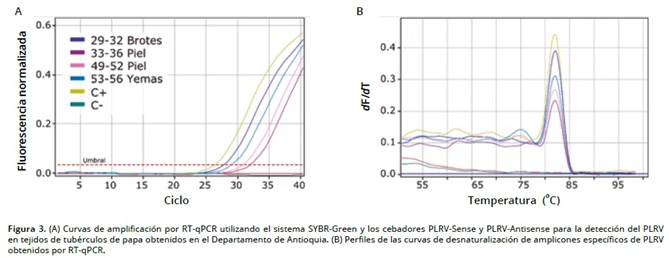
Las pruebas de RT–qPCR detectaron la presencia del PLRV en 10 (83,3%) y 11 (91,6%) de las 12 muestras evaluadas de tejidos de tubérculos de Diacol–Capiro y Criolla–Colombia, respectivamente (Figura 3A). El virus se encontró indistintamente en todos los tejidos muestreados y en los tubérculos procedentes de las dos regiones bajo estudio, obteniéndose valores de Ct que oscilaron entre 17,72 y 30,59 para Diacol–Capiro y 20,23 y 25,75 para Criolla–Colombia (Tabla 1), lo que claramente representa altos niveles de titulo viral en los tubérculos–semilla evaluados, pues existe una relación inversamente proporcional entre el valor de Ct y el titulo viral. Las pruebas de DAS–ELISA para estas mismas muestras, sólo detectaron al PLRV en dos (16,6%) y cinco (41,6%) de las muestras de las variedades Diacol–Capiro y Criolla–Colombia, respectivamente; no encontrándose ninguna muestra que hubiere resultado positiva para la detección del virus utilizando pruebas de DAS–ELISA, pero negativa con RT–qPCR (Tabla 1).
La especificidad de los amplicones obtenidos con RT–qPCR fue confirmada mediante el análisis de desnaturalización, utilizando como base los valores de temperatura de fusión (Tm), que se presentaron en el intervalo de 81,8°C +/– 0,3°C (Figura 3B). La naturaleza de dichos amplicones fue además reconfirmada por secuenciación Sanger de cinco de las muestras (Tabla 1); encontrándose, que dichas secuencias correspondían a una región de CP de 60 pb de las 100 pb originalmente amplificadas por RT–qPCR (Ej. Accesiones JQ346189, KC456054, HE617212, JF939833; Identidad=99%–100%, e=2×10–20–9×10–22), incluyendo accesiones de cepas de este virus de Colombia, previamente depositadas en GenBank por Gil Ramírez, Cotes Torres & Marín Montoya, 2011 (Ej. JF939832, JF939827 y JF939825).
La utilidad de la técnica de RT–qPCR para la detección del PLRV en tubérculos de papa, había sido previamente reportada por (Agindotan, Shiel, & Berger, 2007) y por (Mortimer–Jones, Jones, Jones, Thomson, & Dwyer, 2009), quienes de manera independiente, diseñaron pruebas utilizando el sistema de sondas Taqman para el diagnóstico simultáneo de diferentes virus de papa, incluyendo PLRV. En dichos trabajos, los niveles de sensibilidad alcanzados para la detección del PLRV fueron de hasta 100 copias virales. Interesantemente, dichas pruebas fueron también capaces de detectar el PLRV y los otros virus bajo estudio (Ej. PVX, PVS, TSWV) en una mezcla correspondiente a una proporción de 1 tubérculo infectado en un lote de 300 (Mortimer–Jones, Jones, Jones, Thomson, & Dwyer, 2009 ) o 400 tubérculos (Agindotan, Shiel, & Berger, 2007), lo que representa una gran oportunidad para su aplicación masiva en programas de certificación de semilla de papa. La evaluación de la técnica de RT–qPCR realizada en el presente trabajo, utiliza algunas de las consideraciones adelantadas en dichos estudios, incluyendo la posibilidad de detectar el PLRV directante en tubérculos latentes, y específicamente en tejido de la piel gracias a su restricción al floema. Esto favorece el proceso de toma de decisiones con respecto a la sanidad viral de un lote de tubérculo–semilla, al no requerirse de períodos de almacenamiento para la ruptura de su latencia metabólica; además el empleo de la técnica de RT–qPCR bajo el sistema SYBR–Green I, que aquí se presenta, disminuye los costos de las pruebas y permite el uso del mismo kit para detectar otros virus (si se cuenta con cebadores específicos), sin necesidad de sintetizar sondas individuales para cada uno de ellos, lo cual favorece su utilización en laboratorios de diagnóstico y de producción de semilla certificada con menor capacidad económica.
Las diferencias evidentes encontradas en este estudio con respecto a la detección del PLRV utilizando pruebas de DAS–ELISA y de aquellas basadas en RT–qPCR, resultan ajustadas a los reportes de altos niveles de sensibilidad de esta última técnica. Así por ejemplo, (Mumford, Walsh, Barker, & Boonham, 2000), indican que el uso de RT–qPCR para detectar el Potato mop–top virus (PMTV) en tubérculos de papa, ofrecía niveles de sensibilidad 10000 veces superiores que los alcanzados con las pruebas de ELISA; mientras que Bertolini et al. (2007), al evaluar la detección del Citrus tristeza virus (CTV) en plantas y áfidos, determinaron que la técnica de RT–qPCR pudo detectar hasta 17 copias del virus, presentando niveles de sensibilidad superiores en 100–500 veces con respecto a IC–RT–PCR anidada y hasta de 106 veces con relación a pruebas de DAS–ELISA.
Los resultados obtenidos en esta investigación señalan altos niveles de incidencia de PLRV en tubérculos no certificados de las variedades Diacol–Capiro y Criolla–Colombia utilizados como material de siembra por agricultores de dos de las principales regiones cultivadoras de papa de Antioquia, situación que debe ser corregida con celeridad, pues es posible que esta condición también se presente para otros virus de fácil dispersión por este medio en papa, tales como PVY, PMTV y PVS (Gil Ramírez, Cotes Torres & Marín Montoya, 2011; Frost, Groves, & Charkowski, 2013). Por esto, resulta imperativo que los organismos de sanidad vegetal estatal, gremios de agricultores y compañías de semilla, evalúen los procesos nacionales de producción y comercialización de semilla e incorporen metodologías altamente sensibles para la detección de virus como la RT–qPCR, como parte de sus procedimientos para garantizar la sanidad del material de siembra de papa en Colombia.
Conclusiones
Agradecimientos
Este trabajo fue financiado por la Vicerrectoría de Investigaciones de la Universidad Nacional de Colombia, proyecto: 201010013009 (Modalidad 2: Nuevos proyectos de investigación, creación o innovación) y por International Foundation for Science –IFS– (Suecia, Grant: C/4634–2).
Referencias
Agindotan, B. O., Shiel, P. J., & Berger, P. H. (2007). Simultaneous detection of potato viruses, PLRV, PVA, PVX and PVY from dormant potato tubers by TaqMan real–time RT–PCR. Journal of Virological Methods, 142(1–2), 1–9. http://doi.org/10.1016/j.jviromet.2006.12.012
Álvarez, J.A., Cotes, J.M., & Marín, M. (2011a). Detección de virus asociados al material de siembra de tomate de árbol en Colombia. Biotecnología En El Sector Agropecuario Y Agroindustrial, 9(1), 43–50. Recuperado de http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S1692–35612011000100006&lng=en&nrm=iso&tlng=es
Álvarez, J., Cotes, J. M., & Marín, M. (2011b). Detección molecular de virus en material de siembra de tomate de árbol en Colombia. Revista de Protección Vegeta, 26(2), 80–91. Recuperado de http://censa.mes.edu.cu/index.php/RPV/article/view/163
Bertolini, E., Moreno, A., Capote, N., Olmos, A., de Luis, A., Vidal, E., Cambra, M. (2007). Quantitative detection of Citrus tristeza virus in plant tissues and single aphids by real–time RT–PCR. European Journal of Plant Pathology, 120(2), 177–188. http://doi.org/10.1007/s10658–007–9206–9
Bostan, H.; y Haliloglu, K. (2004). Distribution of PLRV, PVS, PVX and PVY (PVYN, PVYo and PVYc) in the seed potato tubers in Turkey. Pakistan Journal of Biological Sciences, 7(7), 1140–1143. http://doi.org/10.3923/pjbs.2004.1140.1143
Frost, K. E., Groves, R. L., & Charkowski, A. O. (2013). Integrated Control of Potato Pathogens Through Seed Potato Certification and Provision of Clean Seed Potatoes. Plant Disease, 97(10), 1268–1280. http://doi.org/10.1094/PDIS–05–13–0477–FE
Gil Ramírez, J. F., Cotes Torres, J. M., & Marín Montoya, M. (2011). Incidencia y caracterización molecular del Potato Leafroll Virus (PLRV) en las principales regiones productoras de papa de Colombia. Fitosanidad, 15(1), 17–24.
Guzmán, M., Román, V., Franco, L., & Rodríguez, P. (2010). Presencia de cuatro virus en algunas accesiones de la Colección Central Colombiana de papa mantenida en campo. Agronomía Colombiana, 28(2), 225–233. Recuperado de http://www.revistas.unal.edu.co/index.php/agrocol/article/view/18066/37687
Mortimer–Jones, S. M., Jones, M. G. K., Jones, R. A. C., Thomson, G., & Dwyer, G. I. (2009). A single tube, quantitative real–time RT–PCR assay that detects four potato viruses simultaneously. Journal of Virological Methods, 161(2), 289–96. http://doi.org/10.1016/j.jviromet.2009.06.027
Mumford, R. A., Walsh, K., Barker, I., & Boonham, N. (2000). Detection of potato mop top virus and Tobacco rattle virus using a multiplex real–time fluorescent reverse–transcription polymerase chain reaction assay. Phytopathology, 90(5), 448–53. http://doi.org/10.1094/PHYTO.2000.90.5.448
Plchova, H., Cerovska, N., Moravec, T., & Dedic, P. (2009). Short communication: Molecular analysis of Potato leafroll virus isolates from the Czech Republic. Virus Genes, 39(1), 153–155. http://doi.org/10.1007/s11262–009–0374–x
Pooramini, N., Heydarnejad, J., & Massumi, H. (2010). Incidence and Molecular Analysis of Potato leafroll virus in Iran. Journal of Phytopathology, 158(3), 182–185. http://doi.org/10.1111/j.1439–0434.2009.01600.x
Singh, R. P., Kurz, J., Boiteau, G., & Bernard, G. (1995). Detection of potato leafroll virus in single aphids by the reverse transcription polymerase chain reaction and its potential epidemiological application. Journal of Virological Methods, 55(1), 133–143. http://doi.org/10.1016/0166–0934(95)00056–Z
Taliansky, M., Mayo, M. A., & Barker, H. (2003). Potato leafroll virus: a classic pathogen shows some new tricks. Molecular Plant Pathology, 4(2), 81–89. http://doi.org/10.1046/j.1364–3703.2003.00153.x
Tamura, K., Stecher, G., Peterson, D., Filipski, A., & Kumar, S. (2013). MEGA6: Molecular Evolutionary Genetics Analysis version 6.0. Molecular Biology and Evolution, 30(12), 2725–2729. http://doi.org/10.1093/molbev/mst197
Vásquez, V., Montero Astúa, M., & Rivera, C. (2006). Incidencia y distribución altitudinal de 13 virus en cultivos de Solanum tuberosum (Solanaceae) en Costa Rica. Revista de Biología Tropical, 54(4), 1135–1141. http://doi.org/10.15517/rbt.v54i4.3090
Wang, B., Ma, Y., Zhang, Z., Wu, Z., Wu, Y., Wang, Q., & Li, M. (2011). Potato viruses in China. Crop Protection, 30(9), 1117–1123. http://doi.org/10.1016/j.cropro.2011.04.001
Yang, L., Nie, B., Liu, J., & Song, B. (2013). A Reexamination of the Effectiveness of Ribavirin on Eradication of Viruses in Potato Plantlets in vitro Using ELISA and Quantitative RT–PCR. American Journal of Potato Research, 91(3), 304–311. http://doi.org/10.1007/s12230–013–9350–z
Zarghani, S. N., Shams–Bakhsh, M., Zand, N., Sokhandan–Bashir, N., & Pazhouhandeh, M. (2012). Genetic analysis of Iranian population of Potato leafroll virus based on ORF0. Virus Genes, 45(3), 567–74. http://doi.org/10.1007/s11262–012–0804–z