DETECCIÓN DE SECUENCIAS ESPECÍFICAS DE ADN DE S. subterranea
es la responsable de la depreciación en la calidad de los tubérculos debido a la presencia de lesiones en forma de pústula en la epidermis de los mismos y a la disminución en la producción de papa en variedades susceptibles. En la actualidad la sarna polvosa es una enfermedad cuya incidencia está en aumento debido a la persistencia de S. subterranea por varios años en forma de estructuras de resistencia (quistosoros), a la carencia de variedades de papa resistentes, a su fácil dispersión y poca susceptibilidad a tratamientos agroquímicos, entre otros factores (Harrison et al., 1997).
En Colombia se reportó en 1965 y se encuentra ampliamente distribuida en los departamentos de Cundinamarca, Boyacá y Nariño, afectando entre un 50 y un 80% la producción de los tubérculos según la edad de cultivo, implicando pérdidas económicas en el sector agroindustrial, problemas fitosanitarios por la diseminación del patógeno a zonas libres de la enfermedad, y para los productores de semilla de papa, el rechazo en el programa de semilla certificada.
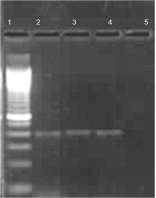
La enfermedad se diagnostica de una forma tardía en semillas y cultivos debido a su difícil detección tanto en el suelo como en semillas asintomáticas; de ahí que sea necesario implementar una metodología rápida, altamente sensible y específica que permita detectar al microorganismo en suelo y semilla de papa, para el control fitosanitario del patógeno en programas de producción de semilla certificada libre de S. subterranea.
MATERIALES Y MÉTODOS Obtención de muestras
El análisis del material biológico se efectuó a partir de tejido vegetal infectado con síntomas típicos causados por S. subterranea (agallas en raíces de plantas y pústulas en epidermis de tubérculos), y de suelos aptos para el cultivo de papa con presencia de este patógeno. Las muestras fueron suministradas y enviadas por el Programa de Producción de Semilla Certificada del ICA, seccional Cundinamarca, y por el Laboratorio de Diagnóstico Fitosanitario, seccional Nariño, al Laboratorio de Diagnóstico Molecular Vegetal ICA-Tibaitatá; una vez recibido este material, se almacenó a -20 °C para la posterior determinación macroscópica, microscópica y molecular del patógeno.
El Laboratorio de Diagnóstico Fitosanitario seccional ICA-Pasto suministró 32 muestras de suelo de un campo experimental infestado con S. subterranea; 30 de estas submuestras (suelo entre 0 cm y 20 cm de profundidad) correspondían a cada uno de los puntos muestreados de dicho campo. Las otras dos muestras restantes son mezclas de suelo de los 30 puntos señalados de las submuestras entre 0-10 cm y 10-20 cm (gráfica 1). Los detalles del tipo de muestra analizado y su procedencia se observan en la tabla 1.
Detección macroscópica y microscópica en tejido vegetal infectado
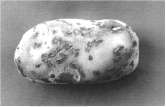
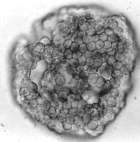
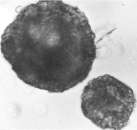
Una vez recibido y almacenado el material biológico, se efectuó una descripción macroscópica de las lesiones típicas de la enfermedad tanto en semillas de papa como en raíces infectadas. Al microscopio se determinó la presencia de quistosoros tanto en pústulas como en agallas de raíz por medio de un montaje húmedo observado bajo los aumentos de 40X y 100X.

Gráfica 1. Esquema obtención de muestras en suelo de un lote experimental de aproximadamente 1 Ha infestado con S. subterranea: recorrido realizado en zig-zag en el departamento de Pasto para la toma de 30 muestras del suelo entre 0-20 cm de profundidad (cada número corresponde a un punto de muestreo).
15