MODIFICACIONES TÉCNICAS EN EL USO DE MICROSATÉLITES Y AFLP
UNH123, UNH129, UNH132, UNH174,UNH189, UNH191, UNH192, UNH209, UNH216, UNH231, seleccionados por poseer regiones repetitivas de dinucleótidos y temperaturas de alineamiento altas para la amplificación en O. Niloticus (Kocher, 1997).
1.20 min. de alineamiento a 47-54 °C, 1.20 min de extensión a 68 o 72 °C y extensión final a 68 o 72 °C por 3 min.
Los productos amplificados fueron almacenados a -20 °C. Las amplificaciones se verificaron por medio de electroforesis en geles de agarosa 3% teñidos con bromuro de etidio a 70 V por 50 minutos en cámara de electroforesis horizontal.
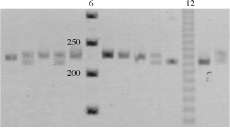
De las doce parejas de cebadores probadas, seis amplificaron positivamente en C. kraussii (tabla 2).
Amplificación en Prochilodus magdalenae y Brycon moorei sinuensis. Se utilizaron primers desarrollados para una especie relacionada, Piaractus mesopotamicus (Calcagnotto et al., 2001). Como control positivo se utilizó ADN de un individuo de la especie Piaractus brachypomus en la cual la eficacia de los primers fue evaluada por los mismos autores.
Cada reacción de PCR se realizó en un volumen total de 12.5 \iL con: 2.0 ^L de ADN, 10 mM de Buffer B de amplificación (100 mM Tris-HCl, 500 mM KCl, 1 % de Triton), 2.5mM de MgCl2 (excepto para el bocachico en el locus Pme20 donde se usaron 2.0 mM), 200 \iM de solución de dNTP, 0.8 ^M de solución de cada primer y 0.5 U de Taq polimerasa. Las condiciones de la reacción se realizaron de la siguiente manera: desnaturalización inicial del ADN a 95 °C por 4 min, 30 a 40 ciclos con 30 segundos de desnaturalización a 95 °C, 30 segundos de alineamiento a 44-57 °C y 30 segundos de extensión a 68-
72 °C. Por último, una extensión a 68-72 °C
por 10 min.

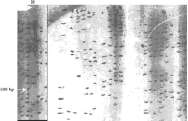
Figura 1. Gel de Agarosa 0.8% donde se verifica la calidad y cantidad de ADN en C. kraussii. Flecha: banda de ADN.
La estandarización de la amplificación se inició siguiendo el protocolo original de Kocher y colaboradores (1998), utilizando como control positivo ADN de O. niloticus. Para lograr una amplificación exitosa en C. kraussii se realizaron cambios en la temperatura de alineamiento y la concentración de MgCl2 (tabla 2).
La PCR se realizó en un volumen final de 25 ^L, con: 10mM de Buffer B de amplificación (100mM Tris-HCl, 500mM KCl, 1% de Triton), 2.5-3.0 mM de MgCl2, 0.16 - 0.18 mM de dNTPs, 0.16 ^M de primer, 1.5 ^L de ADN y 0.5 U de Taq polimerasa. Las condiciones para la PCR fueron: 1 min inicial a 95 °C, seguido de 26 a 32 ciclos a 1 min de desnaturalización a 95 °C,
Tabla 2. Loci amplificados en C. kraussii y condiciones específicas para PCR.
|
Locus
|
Motivo de repetición
|
MgCl2 (mM)
|
Tm y N°
|
||
|
UNH106
|
(CT)13(CA)20
|
2.5
|
50 °Cx 26 ciclos
|
||
|
UNH109
|
(CT)6(CA)9(CT)13(AT)7
|
2.6
|
49 °C x 5 ciclos
|
||
|
|
|
|
50 °C x 5 ciclos 51 °C x 8 ciclos 52 °C x 8 ciclos
|
||
|
UNH132
|
(GA)6(GG)(GA)7GC(GA)5
|
2.5
|
51 °Cx 26 ciclos
|
||
|
UNH191 UNH209
|
(AC)25 (AC)9
|
3.0 3.0
|
51 °C x 2 ciclos 52 °C x 5 ciclos 53 °Cx 10 ciclos 54 °Cx 15 ciclos 47 °Cx 32 ciclos
|
||
|
UNH216
|
(AC)11
|
2.8
|
48 °Cx 28 ciclos
|
||
En el caso del P. magdalenae para los locus Pme5 y Pme14, fue necesario hacer los primeros tres ciclos con una temperatura de alineamiento baja: 45 °C para Pme5 y 44 °C para Pme14. Para el resto de ciclos (37) se usó una temperatura mayor (tabla 3).
Los productos amplificados fueron verificados con el mismo método que para C. kraussii. De los ocho cebadores desarrollados por Calcagnotto y colaboradores (2001) seis fueron exitosamente genotipificados para B. moorei sinuensis y cinco para P. magdalenae. Para el locus Pme28 en B. moorei sinuensis sólo se observaron bandas en regiones no correspon-
Tm: temperatura de alineamiento; N°: número de ciclos para cada temperatura de alineamiento.
75