Detección, identificación y localización geográfica de Begomovirus que afectan al tomate en Colombia
Palabras clave:
Begomovirus, Bemisia tabaci, tomate, Dot blot, PCR, tomato (es)Detection, identification and geographical localization of tomato-infecting Begomovirus in Colombia
Título corto: Begomovirus que afectan tomate en Colombia
Resumen
En la actualidad, los Begomovirus (Familia Geminiviridae) se han convertido en la mayor amenaza para los cultivos de interés agrícola ubicados en las zonas tropicales y templadas del planeta. Estos virus son transmitidos por la mosca blanca Bemisia tabaci, la cual en los últimos años en Colombia ha tenido un aumento significativo en sus poblaciones y se ha asociado con la aparición de síntomas virales en cultivos de tomate. Muestras de tomate con síntomas virales típicos fueron recolectadas en las cinco principales zonas productoras de esta solanácea en el país. Los Begomovirus fueron detectados por medio de la técnica de hibridación de ácidos nucleicos tipo Dot blot así como por medio de la reacción en cadena de la polimerasa (PCR) en todas las muestras colectadas. Con la excepción de un reporte previo en el Valle del Cauca, este es el primer reporte de Begomovirus afectando cultivos de tomate en los departamentos de Antioquia, Santander, Boyacá y Cundinamarca. Asimismo, es la primera vez que se informa sobre Begomovirus que afectan cultivos de tomate localizados por encima de 1500 msnm en Colombia.
Palabras clave: Begomovirus, Bemisia tabaci, tomate, Dot blot, PCR.
Abstract
The begomoviruses (Family Geminiviridae) have recently emerged as
Key words: Begomovirus, Bemisia tabaci, tomato, Dot blot, PCR.
samples with typical Begomovirus symptoms were collected in five different departments, comprising the mayor tomato growing areas of the country. Begomovirus were detected by Polymerase Chain Reaction (PCR) or Dot Blot Hybridization in all tomato samples collected in whole tomato growing areas of the country. With exception for Valle del Cauca, this is the first report of tomato-infecting Begomovirus in Antioquia, Santander, Boyacá and Cundinamarca departments. Also this is the first report of tomato-infecting Begomovirus crops located above 1500 masl in Colombia.ARTÍCULO DE INVESTIGACIÓN
Detección, identificación y localización geográfica de Begomovirus que afectan al tomate en Colombia
Detection, identification and geographical localization of tomato-infecting Begomovirus in Colombia
Título corto: Begomovirus que afectan tomate en Colombia
Juan Carlos Vaca-Vaca 1 , Jhon Fredy Betancurt-Pérez 2 , Karina López-López 3.
1 M.Sc. Profesor, Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, Sede Palmira. jcvacava@unal.edu.co
2 Estudiante de doctorado, Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, Sede Palmira. Fredy.betancur@gmail.com
3 Ph.D., Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, Sede Palmira. klopezl@unal.edu.co
Recibido: agosto 14 de 2010 Aprobado: mayo 30 de 2011
Resumen
En la actualidad, los Begomovirus (Familia Geminiviridae) se han convertido en la mayor amenaza para los cultivos de interés agrícola ubicados en las zonas tropicales y templadas del planeta. Estos virus son transmitidos por la mosca blanca Bemisia tabaci, la cual en los últimos años en Colombia ha tenido un aumento significativo en sus poblaciones y se ha asociado con la aparición de síntomas virales en cultivos de tomate. Muestras de tomate con síntomas virales típicos fueron recolectadas en las cinco principales zonas productoras de esta solanácea en el país. Los Begomovirus fueron detectados por medio de la técnica de hibridación de ácidos nucleicos tipo Dot blot así como por medio de la reacción en cadena de la polimerasa (PCR) en todas las muestras colectadas. Con la excepción de un reporte previo en el Valle del Cauca, este es el primer reporte de Begomovirus afectando cultivos de tomate en los departamentos de Antioquia, Santander, Boyacá y Cundinamarca. Asimismo, es la primera vez que se informa sobre Begomovirus que afectan cultivos de tomate localizados por encima de 1500 msnm en Colombia.
Palabras clave: Begomovirus, Bemisia tabaci, tomate, Dot blot, PCR.
Abstract
The begomoviruses (Family Geminiviridae) have recently emerged as a serious pathogens of agronomic and horticultural crops in subtropical and tropical regions of world. These viruses are transmitted by the whitefly (Bemisia tabaci) that recently has got a significant increase in its populations and also has been associated with the appearance of geminivirus typical symptoms in tomato crops around Colombia. Tomato samples with typical Begomovirus symptoms were collected in five different departments, comprising the mayor tomato growing areas of the country. Begomovirus were detected by Polymerase Chain Reaction (PCR) or Dot Blot Hybridization in all tomato samples collected in whole tomato growing areas of the country. With exception for Valle del Cauca, this is the first report of tomato-infecting Begomovirus in Antioquia, Santander, Boyacá and Cundinamarca departments. Also this is the first report of tomato-infecting Begomovirus crops located above 1500 masl in Colombia.
Key words: Begomovirus, Bemisia tabaci, tomato, Dot blot, PCR.
Introducción
Los Begomovirus pertenecen a la Familia Geminiviridae, y su genoma está constituido por una o dos moléculas de ADN circular de cadena sencilla, son diseminados por la mosca blanca (Bemisia tabacci> Genn. biotipo B) a plantas dicotiledóneas y replican su genoma en el núcleo de sus hospederos por un mecanismo de círculo rodante. Este hecho hace de estos virus un excelente modelo para estudiar muchos de los fenómenos moleculares que se verifican en plantas. El nombre de geminivirus proviene de la morfología de su partícula viral la cual, vista al microscopio electrónico, se asemeja a dos poliedros fusionados por una de sus caras.
En la actualidad, los Begomovirus son considerados como la mayor amenaza para los cultivos de interés agrícola ubicados en las zonas tropicales y templadas del planeta en donde se han registrado sensibles pérdidas en cultivos de frijol, yuca, algodón, tabaco, ají, cucurbitáceas y solanáceas a causa de este virus (Seal et al., 2006).
El tomate (Solanum lycopersicum L) es la hortaliza más difundida en todo el mundo y la de mayor valor económico. Las investigaciones de Polston y Anderson (1997) y Ribeiro et al. (2003) dejan entrever que el cultivo del tomate en Latinoamérica ha sido fuertemente afectado por virosis causadas principalmente por Begomovirus. Morales et al. (2002), advierten de la creciente amenaza que este tipo de virus genera para la agroindustria tomatera del país, debido a que esta solanácea parece ser una especie particularmente susceptible a este grupo de fitopatógenos. En Colombia se han realizado escasos estudios sobre los Begomovirus que afectan el tomate. Se cuenta a la fecha con la caracterización parcial de un Begomovirus aislado en el Valle del Cauca, cuyo nombre propuesto es el de virus del mosaico amarillo del tomate (Martínez et al., 2008; Morales et al., 2002).
Materiales y métodos
Recolección del material vegetal. La recolección de material vegetal se realizó en las principales zonas productoras de tomate ubicadas en los departamentos de Cundinamarca, Santander, Valle, Boyacá y Antioquia. El muestreo en campo se llevó a cabo mediante la recolección de hojas jóvenes de plantas de tomate que presentaban síntomas típicos de la enfermedad begomoviral tales como: enanismo, mosaicos amarillo brillante, moteados cloróticos, clorosis foliar marginal, enrollamiento foliar, deformaciones foliares y arrugamientos de las hojas (Polston y Anderson, 1997; Nava et al. 2006). Asimismo, fueron registrados los datos de ubicación satelital del sitio de recolección de muestras vegetales mediante el uso de un equipo de GPS-CSX-60 (Garmin ®) con barómetro, esta última herramienta es útil también para tomar los datos de altura del cultivo en metros sobre el nivel del mar. Las muestras colectadas en campo fueron llevadas al laboratorio en donde se almacenaron a -70 °C para su análisis posterior.
Extracción de ácidos nucleicos. A partir del tejido vegetal (hojas) previamente recolectado en campo y almacenado a -70 °C, se aisló el ADN total empleando dos estrategias metodológicas: el protocolo propuesto por Dellaporta et al. (1983) y el DNeasy Plant Kit (QIAGEN ®), siguiendo para ello las instrucciones del fabricante. A fin de verificar la calidad y cantidad de los ADN totales extraídos, se realizó una electroforesis en gel de agarosa al 0,8% (p/v) siguiendo los protocolos descritos por Sambrook et al. (2001).
Hibridación de ácidos nucleicos tipo Dot blot. La hibridación de ácidos nucleicos se llevó a cabo siguiendo una estrategia no radiactiva, utilizando el kit Gene Images AlkPhos Direct Labelling and Detection System (Amersham-Pharmacia Biotech ®). Para ello, 5 μl de ADN (1 μg/μl) de cada muestra problema se colocaron sobre una membrana de Nylon Hybond (+) (Amersham ®). Con el fin de unir el ADN a la membrana, esta se colocó a 80 °C por 2 h en un horno de prehibridación (Problot 12S Hybridization Oven LabNet ®). La sonda de ADN utilizada para la hibridación corresponde a un fragmento del gen de la proteína de la cápside (CP) del virus huásteco de Chile (PHYVV), facilitado por el doctor Rafael Rivera Bustamante (Cinvestav-IPN, México). El marcaje de la sonda fue realizado con el kit Gene Images AlkPhos Direct Labelling (Amersham-Pharmacia Biotech ®) siguiendo las instrucciones del fabricante. El proceso de hibridación se realizó a 55 ?C durante toda la noche en una solución de hibridación AlkPhos Direct (Amersham-Pharmacia Biotech ®) en un horno de hibridación (Problot 12S Hybridization Oven LabNet ®). Los lavados poshibridación se realizaron como sigue: dos lavados a 55 ?C de 10 min cada uno en una solución de lavado primario (Urea 2M, 0,1% SDS, 50 mM fosfato de sodio pH 7,0, 150 mM NaCl, 1 mM MgCl2 y 0,2% de reactivo de bloqueo), y dos lavados a temperatura ambiente por 5 min cada uno en una solución de lavado secundario (Tris base 50 mM, NaCl 100 mM y 2 mM MgCl2). La generación de la señal quimiofluorescente se realizó con el sustrato ECF siguiendo las instrucciones del fabricante (Amersham-Pharmacia Biotech ®), y la detección de la señal fluorescente se llevó a cabo en el equipo Molecular Imager ® Gel Doc ™ XR System (Biorad ®).
Reacción en cadena de la polimerasa (PCR). Para implementar la estrategia de PCR se siguió la metodología propuesta por Rojas et al. (1993), empleando los primers PAL1v1978/ PARc496 y PBL1v2040/ PCRc1, que amplifican un fragmento de 1100 pb del componente ADN-A, y un fragmento de 600 pb del componente ADN-B, respectivamente. La reacción de PCR se llevó a cabo en un volumen de 25 ul empleando un termociclador Biorad ® modelo C100. Los fragmentos amplificados fueron visualizados en un gel de agarosa al 0,8% (p/v).
Resultados y discusión
Con el fin de contar con un panorama nacional de los Begomovirus (Familia Geminiviridae) que estaban afectando cultivos de tomate en el país, se realizó una colecta en los principales departamentos productores de esta solanácea. Mediante este muestreo en la región andina de Colombia se evidenció la presencia de síntomas virales típicos de la infección por geminivirus en plantas de tomate cultivadas tanto en invernadero como a cielo abierto de los departamentos de Boyacá, Cundinamarca, Antioquia, Valle del Cauca y Santander (figura 1). Asimismo, fue evidente la presencia de mosca blanca Bemisia tabaci en la mayoría de los cultivos de tomate (figura 2c), así como el uso indiscriminado de pesticidas para el control de la misma. Estos últimos utilizados con el fin de eliminar la mosca blanca, una vez que el agricultor observaba síntomas de virus. Es importante mencionar que en los cultivos de Boyacá y Antioquia se observaron síntomas leves de virosis, en especial no se observaron mosaicos y clorosis generalizados. La sintomatología observada en el Valle fue muy similar a la encontrada en Cundinamarca y en algunas zonas de Santander (figura 2b). Es importante mencionar que en algunas localidades de Santander se observó una sintomatología acentuada de clorosis y mosaicos, acompañados de una coloración amarilla fluorescente intensa (figura 2d). En total se colectaron 74 muestras prevenientes de 74 fincas o veredas localizadas en 5 departamentos (Boyacá, Cundinamarca, Antioquia, Valle del Cauca y Santander), ubicados entre los 655 a 2184 msnm (tabla 1).
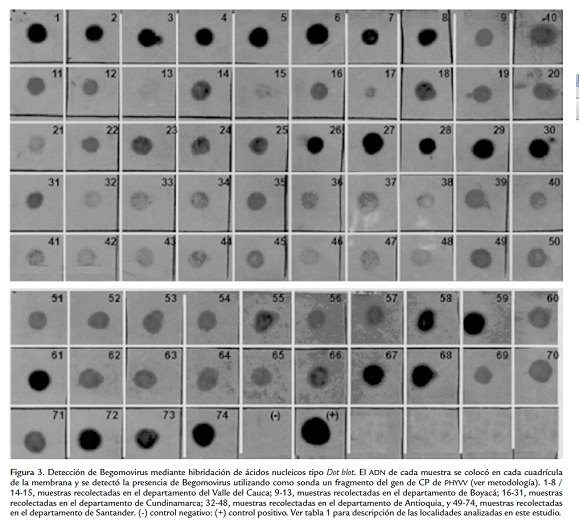
Una primera aproximación para determinar la presencia de Begomovirus en las muestras recolectadas en campo fue la implementación de la técnica de hibridización de ácidos nucleicos (NAH) tipo Dot Blot. Esta estrategia permite analizar grandes cantidades de muestras sospechosas de estar infectadas por un virus particular empleando una sonda o fragmento de ADN conservado que permite identificar de manera específica un único género o familia viral particular. En este caso, la metodología de NAH permitió evidenciar la presencia de Begomovirus pertenecientes a la Familia Geminiviridae en todas las muestras analizadas, encontrándose mayor intensidad de la señal en aquellas provenientes de Valle, Cundinamarca y Santander. Vale la pena destacar que la técnica Dot Blot se rige por el principio del todo o nada, es decir, hay o no hay presencia de una entidad viral particular, dada la alta sensibilidad de esta prueba (figura 3 y tabla 1).
Para confirmar los resultados obtenidos por medio NAH tipo Dot blot se implementó la estrategia de PCR utilizando primers o cebadores degenerados que amplifican regiones conservadas presentes en todos los Begomovirus bipartitas del hemisferio occidental (Rojas et al., 1993). Los iniciadores utilizados amplificaron productos de PCR de los tamaños esperados de 1200 y 500 pb, correspondientes a los genomas begomovirales A y B respectivamente (figura 4). Mediante este enfoque se confirmó la presencia de Begomovirus bipartitas en muestras vegetales de tomate recolectadas en campo en los departamentos de Valle (municipios de Palmira, Pradera, Tuluá, Caicedonia y Barragán), Cundinamarca (municipios de Silvania y Pasca) y Santander (municipios de Pie de Cuesta, Girón, Cepita, San Gil, Berlín y Socorro) (tabla 1). Antioquia y Boyacá no presentaron muestras positivas para Begomovirus por esta técnica (PCR), sin embargo, estas sí fueron positivas por estrategia de NAH tipo Dot blot. Esto puede estar relacionado con la concentración del virus en la planta en el momento en que se realizó la recolección, hecho que a su vez está íntimamente relacionado con el estado del ciclo infectivo del geminivirus. Es decir, la sensibilidad del NAH tipo Dot Blot es mayor e independiente del estado del ciclo infectivo del virus, pues detecta el geminivirus esté replicándose o no. Mientras que la PCR requiere que haya abundantes copias del genoma viral que permitan amplificar un fragmento de ADN de un tamaño esperado (Potter et al., 2003).
Asimismo, se pudo evidenciar que hubo una relación directa entre aquellas muestras de tomate recolectadas en campo que presentaban sintomatología típica, con aquellas que fueron positivas para la presencia de Begomovirus, diagnosticadas empleando estrategias moleculares (Dot Blot y PCR). Es decir, que en aquellas zonas del país en que hubo pocas evidencias de sintomatología viral, como lo fueron las muestras de Antioquia y Boyacá, se detectaron algunas muestras positivas, mientras que en Santander, en donde se evidenció una alta presencia de síntomas virales, fue donde mayor número de muestras positivas se diagnosticaron.
Teniendo en cuenta que los Geminivirus no son transmitidos por semilla, su detección en pisos altitudinales superiores a los 1300 msnm (límite superior reportado en Colombia para Bemisia tabaci) puede explicarse por dos hechos: primero, porque este insecto está colonizando en altura nuevos nichos agroecológicos, situación posiblemente asociada al fenómeno del calentamiento global que en este momento está sufriendo el planeta. Por otra parte, puede explicarse por actividades derivadas de la plantulación del material vegetal de tomate, práctica que puede contribuir sin saberlo a diseminar material contaminado con estos virus a zonas en altura en donde estas entidades virales no se encontraban antes. Si a eso le agregamos la colonización en altura que las poblaciones de mosca blanca parecen estar logrando en este momento en Colombia, la situación se agrava, pues se podrían estar dando las condiciones para que los Begomovirus, con el concurso de su ampliamente polífago vector biológico (la mosca blanca) se disemine y afecte no solo al tomate sino a otros cultivos cercanos que están presentes en ese piso altitudinal particular. No sobra recordar que los Geminivirus son considerados hoy en día como la principal amenaza viral de los cultivos de interés económico ubicados en zonas tropicales y subtropicales del planeta (Seal et al., 2006).
Es interesante resaltar que algunos materiales de tomate recolectados en campo y que aparentemente eran asintomáticos para enfermedad viral arrojaron resultados positivos a Begomovirus al ser analizados por medio de las estrategias moleculares. Este hecho pone de relieve la importancia creciente que el diagnóstico molecular de virus vegetales tiene hoy en día a nivel mundial. El caso concreto del diagnóstico de entidades begomovirales empleando un enfoque molecular es un excelente ejemplo de esta tendencia. Durante muchos años se usó la proteína de la cápside de los geminivirus para desarrollar anticuerpos monoclonales, los cuales eran utilizados para diagnosticar la presencia de este tipo de virus en plantas, empleando enfoques serológicos tales como la técnica Elisa. Sin embargo, la gran cantidad de motivos de aminoácidos comunes que comparten las proteínas de las cápsides de los geminivirus condujo en muchas ocasiones a reacciones cruzadas que impedían al investigador determinar con certeza qué geminivirus era el agente causal de una patología vegetal particular; asimismo, se dieron casos en que, dada la baja concentración del virus en un tejido vegetal particular, era imposible detectarlo con este tipo de estrategia (Harrison et al., 2002). Por esta razón, en los últimos años se hizo necesario que el diagnóstico de geminivirus fundamentado en estrategias serológicas diera paso a un sistema basado en las características íntimas del propio genoma del virus, es decir, al diagnóstico molecular, el cual es más sensible, confiable y además permite establecer no solo la identidad viral exacta sino también posibilita el establecimiento de las relaciones filogenéticas del virus con otros ya reportados previamente.
Conclusiones
Los resultados obtenidos demuestran que hay Begomovirus pertenecientes a la Familia Geminiviridae que afectan los cultivos de tomate en diferentes zonas geográficas de Colombia ubicadas entre los 655 a 2184 msnm. Con la excepción de un reporte previo de Begomovirus en el Valle del Cauca (Martínez et al., 2008), esta es la primera vez que se diagnostica la presencia de Begomovirus afectando el cultivo de tomate en Antioquia, Santander, Boyacá y Cundinamarca. Asimismo, en todas las zonas en donde se recolectó material vegetal de tomate se evidenció la presencia de la mosca blanca (Bemisia tabaci), el vector biológico de los Begomovirus. Para los cultivadores de tomate en Colombia este hecho es preocupante, si se tiene en cuenta que las muestras vegetales fueron recolectadas en sitios ubicados por encima de los 1300 msnm, límite superior reportado para este insecto (por ejemplo, a 2184 msnm en el municipio de Silvania, Cundinamarca). Es posible que el cambio climático mundial esté facilitando de alguna manera la conquista de nuevos nichos por Bemisia tabaci lo conllevaría que los Begomovirus que estos transmiten logren afectar a su vez nuevos cultivos ubicados en esos pisos altitudinales. Esta circunstancia es preocupante por la carencia total de material de tomate resistente al ataque de los Begomovirus en el país. El diagnóstico molecular de Geminivirus es una estrategia que además de sensible y confiable permite establecer no solo la identidad viral exacta sino también posibilita el establecimiento de las relaciones filogenéticas del virus con otros ya reportados previamente. En este sentido, nuestro equipo de trabajo está llevando a cabo estudios adicionales a nivel molecular tendientes a determinar específicamente la identidad molecular de los Begomovirus endémicos que están afectando el cultivo de tomate en Colombia, así como sus relaciones evolutivas con otros que están afectando al tomate en otras latitudes del mundo.
Agradecimientos
El presente proyecto de investigación fue financiado con recursos del Ministerio de Agricultura y Desarrollo Rural de Colombia, contrato 134-2008N6396-3460, Defrescura S.A. y la Universidad Nacional de Colombia.
Los autores agradecen el apoyo recibido para realizar las colectas de los materiales vegetales a nivel nacional a las empresas DeFrescura S.A. y Agroindustrial de Semillas S. A. Al doctor Rafael Rivera Bustamante de Cinvestav-IPN, México, por haber facilitado gentilmente la sonda de ADN utilizada para realizar la hibridación de ácidos nucleicos. Betancurt J. agradece a Colciencias la beca recibida para realizar estudios de doctorado en Ciencias Agropecuarias. A Kerly Motta, por el apoyo en la elaboración de los mapas de las zonas de muestreo con datos satelitales.
Referencias bibliográficas
1 Dellaporta, S. L., Woods, J., Hicks, J. B. 1983. A plant DNA minipreparation: Version-II. Plant Mol. Biol. Rep., 1: 19-21.
2 Harrisson, B. D., Swanson, M. M., Forguette, D. 2002. Begomovirus coat protein serology, variation and function. Physiol. Mol.Plant Pathol. (60): 257-71.
3 Harrisson, B. D., Swanson, M. M., Forguette, D. 2002. Begomovirus coat protein serology, variation and function. Physiol. Mol.Plant Pathol. (60): 257-71.
4 Martínez, A. K., Morales, F. J., Vallejo, C. F. A. 2008. Caracterización molecular de un begomovirus del tomate en el Valle del Cauca, Colombia, y búsqueda de fuentes de resistencia para el mejoramiento de la variedad Unapal Maravilla. Acta Agron., 57 (3).
5 Morales, F. J., Martínez, A. K. Velasco, A. C. 2002. Nuevos brotes de geminivirus en Colombia. Fitopatología Colombiana, 26: 76-78.
6 Nava, A., Patte, P., Hiebert, E., Polston, J. 2006. Detection and Variability of begomoviruses in tomato from the Andean states of Venezuela. Plant Dis., 90: 61-66.
7 Polston, J. E., Anderson, P. K. 1997. The emergence of whitefly-transmited geminivirus in tomato in the western hemisphere. Plant Dis., 81: 1358-1369.
8 Potter, J. L., Nakhla, M. K., Mejía, L., Maxwell, D. P. 2003. PCR and DNA hybridization methods for specific detection of bean-infected begomovirus in Americas and Caribean. Plant. Dis. 87: 1205-1212.
9 Riveiro, S., Ambrozevicious, L. P., Ávila, A., Becerra, I. C., Calegario, R., Fernández, J. J. et al. 2003. Distribution and genetic diversity of tomato-infecting Begomovirus in Brazil. Arch. Virol., 148:281-295.
10 Rojas, M. R., Gilbertson, R. L., Ruseel, D. R., Maxwell, D. P. 1993. Use of generate oligonucleótidos in the polymerase chain reaction to detect whitefly-transmitted geminivirus. Plant disease, 77: 340-347.
11 Sambrook, J., Russell, D. 2001. Molecular Cloning. A laboratory Manual. 3 ed. New York: Cold Spring Harbor Laboratory.
12 Seal, S. E., van den Bosch, F., Jeger, M. J. 2006. Factors Influencing Begomovirus Evolution and Their Increasing Global Significance: Implications for Sustainable Control. Crit. Rev. Plant. Sci. 25: 1-23.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2011 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).