Expresión diferencial durante la interacción Solanum tuberosum - Phytophthora infestans
Palabras clave:
pastusa suprema, diacol-capiro, parda pastusa, despliegue diferencial, differential display (es)Título en ingles: Differential expression during Solanum tuberosum- Phytophthora infestans interaction
Resumen: La papa (Solanum tuberosum L.) es el cuarto cultivo más importante a nivel mundial y es el producto agrícola con mayor demanda de fungicidas, insecticidas y fertilizantes químicos. Las pérdidas mundiales ocasionadas por Phytophthora infestans (Mont.) de Bary en este cultivo, ascienden a 6,7 billones de dólares al año y su control químico genera un aumento en los costos, perjudica la salud humana y el ambiente. Todo esto justifica la búsqueda constante de mecanismos alternativos para el control de la enfermedad, entre ellos la obtención de variedades resistentes mediante cisgenesis usando genotipos silvestres. Como un aporte en este sentido, y dada la falta de conocimiento de lo que controla y constituye la diferencia entre una respuesta compatible e incompatible, en el presente estudio se compararon los perfiles de expresión génica obtenidos mediante Despliegue Diferencial de variedades resistentes y susceptibles durante su interacción con P. infestans. Los resultados evidenciaron diferencias en la expresión génica, tanto a distintos tiempos post-inoculación como en el tipo de cambio de expresión, incluyendo la presencia y ausencia de bandas diferenciales y el aumento o disminución en su intensidad. Al analizar las secuencias de fragmentos diferencialmente expresados, se encontró que algunos fragmentos sobre-expresados en las variedades susceptibles, tenían homología con secuencias que codifican para una serina-acetiltranferasa y para la subunidad β de la RNA polimerasa. Por su parte, fragmentos sobre-expresados en la variedad resistente, tenían homología con una secuencia codificante para un dominio transmembranal.
Palabras claves: pastusa suprema; diacol-capiro; parda pastusa; despliegue diferencial
Abstract: Potato (Solanum tuberosum L.) is the fourth most important crop worldwide; also, is the agriculture product with most fungicides, insecticides and chemical fertilizers requirement. Worldwide losses caused by Phytophthora infestans (Mont.) de Bary in this crop, amount to 6,7 billion dollars per year and its chemical control increase production costs, harming human health and environment. For these reasons, is necessary constant research for alternative mechanisms to control disease, including development of resistant varieties using cis-genesis from wild genotypes. As a contribution in this way, and the lack of knowledge of what controls and is the difference between compatible and incompatible interaction, in this study we compared gene expression profiles obtained by Differential Display from resistant and susceptible varieties, during their interaction with P. infestans. The results showed differences in gene expression between resistant and susceptible varieties, at different times post-inoculation as well as exchange expression rate, including the presence and absence of differential bands and increase or decrease in their intensity. After analyzing the sequences of differential expressed fragments, we found that some overexpressed fragments from susceptible varieties had homology with an encoded sequence for a serine-acetyltransferase and for a RNA Polymerase β subunit. On the other hand, overexpressed fragments from resistant variety, had homology with an encoded sequence for a transmembrane domain.
Key words: pastusa suprema; diacol-capiro; parda pastusa; differential display
ARTÍCULO DE INVESTIGACIÓN
Expresión diferencial durante la interacciónSolanum tuberosum - Phytophthora infestans
Differential expression during Solanum tuberosum-Phytophthora infestans interaction
Zulma Isabel Monsalve-Fonnegra1, María Isabel Monsalve-Restrepo2, Aura Inés Urrea-Trujillo3, José Luis Zapata4.
1Ph.D. en Biotecnología. Docente Facultad de Ciencias Exactas y Naturales, Universidad de Antioquia Calle 67 N° 53-108, oficina 7-104. A. A. 1226. Medellín (Antioquia), Colombia. zmonsalv@matematicas.udea.edu.co ;zmonsalve@gmail.com
2 cM.Sc. en Biotecnología. Estudiante Corporación Académica Ambiental Universidad de Antioquia.
3 Ph.D. Docente Facultad de Ciencias Exactas y Naturales, Universidad de Antioquia.aurrea@matematicas.udea.edu.co
4 Ph.D. Investigador Corpoica-La Selva.
Recibido: febrero 01 de 2012 Aprobado: junio 20 de 2012
Resumen
La papa (Solanum tuberosum L.) es el cuarto cultivo más importante a nivel mundial y es el producto agrícola con mayor demanda de fungicidas, insecticidas y fertilizantes químicos. Las pérdidas mundiales ocasionadas porPhytophthora infestans (Mont.) de Bary en este cultivo, ascienden a 6,7 billones de dólares al año y su control químico genera un aumento en los costos, perjudica la salud humana y el ambiente. Todo esto justifica la búsqueda constante de mecanismos alternativos para el control de la enfermedad, entre ellos la obtención de variedades resistentes mediante cisgenesis usando genotipos silvestres. Como un aporte en este sentido, y dada la falta de conocimiento de lo que controla y constituye la diferencia entre una respuesta compatible e incompatible, en el presente estudio se compararon los perfiles de expresión génica obtenidos mediante Despliegue Diferencial de variedades resistentes y susceptibles durante su interacción con P. infestans. Los resultados evidenciaron diferencias en la expresión génica, tanto a distintos tiempos post-inoculación como en el tipo de cambio de expresión, incluyendo la presencia y ausencia de bandas diferenciales y el aumento o disminución en su intensidad. Al analizar las secuencias de fragmentos diferencialmente expresados, se encontró que algunos fragmentos sobre-expresados en las variedades susceptibles, tenían homología con secuencias que codifican para una serina-acetiltranferasa y para la subunidad Β de la RNA polimerasa. Por su parte, fragmentos sobre-expresados en la variedad resistente, tenían homología con una secuencia codificante para un dominio transmembranal.
Palabras clave: pastusa suprema, diacol-capiro, parda pastusa, despliegue diferencial.
Abstract
Potato (Solanum tuberosum L.) is the fourth most important crop worldwide; also, is the agriculture product with most fungicides, insecticides and chemical fertilizers requirement. Worldwide losses caused by Phytophthora infestans (Mont.) de Bary in this crop, amount to 6,7 billion dollars per year and its chemical control increase production costs, harming human health and environment. For these reasons, is necessary constant research for alternative mechanisms to control disease, including development of resistant varieties using cis-genesis from wild genotypes. As a contribution in this way, and the lack of knowledge of what controls and is the difference between compatible and incompatible interaction, in this study we compared gene expression profiles obtained by Differential Display from resistant and susceptible varieties, during their interaction with P. infestans. The results showed differences in gene expression between resistant and susceptible varieties, at different times post-inoculation as well as exchange expression rate, including the presence and absence of differential bands and increase or decrease in their intensity. After analyzing the sequences of differential expressed fragments, we found that some overexpressed fragments from susceptible varieties had homology with an encoded sequence for a serine-acetyltransferase and for a RNA Polymerase Β subunit. On the other hand, overexpressed fragments from resistant variety, had homology with an encoded sequence for a transmembrane domain.
Key words: pastusa suprema, diacol-capiro, parda pastusa, differential display.
Introducción
Constantemente, las plantas son afectadas por virus, bacterias, oomicetos, hongos, nematodos e insectos (Jacobs et al., 2010); esto ha obligado a los agricultores a realizar labores de prevención, manejo y control acordes con su disponibilidad de recursos técnicos y financieros. El manejo indiscriminado de los productos químicos para el control de los patógenos, que en la mayoría de los casos no tiene en cuenta su ciclo de vida o las condiciones meteorológicas durante el cultivo, ocasiona efectos perjudiciales en la salud, no sólo de los productores sino también de los consumidores, esto sin dejar de lado el daño generado en la biodiversidad y el medio ambiente (Giraldo et al., 2010). Además, la aparición de agentes etiológicos resistentes a los productos químicos o que exhiben una mayor agresividad, incrementan sustancialmente los costos de producción, los impactos ambientales y en la salud (Leonards-Schippers et al., 1994). Por estas razones, se han invertido muchos esfuerzos para comprender los mecanismos de resistencia innata de las plantas contra patógenos, con el fin de establecer estrategias genéticas que confieran resistencia en plantas susceptibles ante los agentes infecciosos (Cornelissen y Melchers 1993; Martinez y Osorio 2007; McDowell y Woffenden 2003; Ros et al., 2005; Stuiver y Custers 2001). Para lograr esto, es muy importante conocer detalladamente cómo se da la interacción planta-patógeno y los mecanismos de respuesta que establece el hospedero para defenderse y limitar la infección (Martini et al., 1993).
El tizón tardío de la papa (Solanum tuberosum L.), causado por Phytophthora infestans, es la enfermedad más devastadora reportada en plantas, ha sido responsable de hambrunas y grandes pérdidas económicas (Abad et al., 1995). En la actualidad, se estima que las perdidas mundiales ocasionadas por la enfermedad ascienden a 6,7 billones de dólares (Haas et al., 2009). Sus síntomas incluyen la destrucción o quemazón del follaje y la pudrición seca de los tubérculos (Díaz et al., 2003). Adicionalmente, si los campos no están protegidos y las condiciones ambientales son óptimas, los cultivos pueden ser destruidos en su totalidad en tan sólo 10 o 15 días (Fry 2008; Jaramillo 2003; Torres 2002).
El cultivo de la papa ocupa el cuarto lugar en importancia a nivel mundial después del arroz, el trigo y el maíz, debido a que es una de las principales fuentes de alimento, especialmente en países en desarrollo, dado que satisface requerimientos energéticos y nutricionales (FAO 2010; Donaire y Garcia 2006); de otro lado, es una importante fuente de empleo e ingresos económicos (Cevipapa 2010). En Colombia, es uno de los productos agrícolas con mayor demanda de fungicidas, insecticidas y fertilizantes químicos (Agrocadenas 2006).
Según la Federación Colombiana de Productores de Papa (FEDEPAPA), en Colombia existen más de 30 variedades pero sólo 10 de ellas tienen importancia comercial. La variedad denominada Parda Pastusa es la de mayor consumo, le sigue en importancia la Diacol Capiro (conocida como R12 negra) utilizada principalmente a nivel industrial. Otras variedades son la ICA-Puracé, la Tuquerreña o Sabanera y la Criolla (Solanum phureja). Desafortunadamente, la mayoría de estas variedades son altamente susceptibles a P. infestans (Maldonado et al., 2010) lo que conlleva a aplicaciones frecuentes de fungicidas, representando más del 6% de los costos totales de producción (Villareal et al., 2007).
En el genoma de Solanum sp. se encuentran codificados varios genes que confieren resistencia a diversos patógenos; inclusive, existen especies silvestres que tienen resistencia innata a P. infestans (Müller y Black 1952; Collard et al., 2005; Colon et al., 1995a; Colon et al., 1995; Mosquera et al., 2008; Landeo et al., 2000; James y Fry 1983; Malcolmson y Black 1966; Schick et al., 1958; Turkensteen 1993; Wastie 1991). En papa se han descrito los tipos de resistencia genética a P. infestans "vertical" y "horizontal". La primera de ellas es mediada por genes R y corresponde a una respuesta de hipersensibilidad (Li et al., 2009; Bormann et al., 2004; Mosqueraet al., 2008; Wastie 1991). La resistencia horizontal, es controlada por loci de rasgos cuantitativos (QTL) (Agrios 2005; Collard et al., 2005) que atenúan o detienen el progreso de la enfermedad (Mosquera et al., 2008). Las investigaciones referentes al conocimiento del patosistema Solanum sp. - P. infestans y su consiguiente respuesta de incompatibilidad o compatibilidad, también han esclarecido que además de los genes R y los QTL, en el control de la resistencia participan variedad de genes funcionales en la respuesta de defensa (Leonards-Schippers et al., 1994; Trognitz et al., 2002), los cuales codifican componentes clave en la síntesis de metabolitos primarios y secundarios (Joos and Hahlbrock 1992).
En las investigaciones encaminadas a la búsqueda de variedades resistentes, se han reportado algunas muy prometedoras, particularmente de S. phureja (Estrada 2000; Guzmán et al., 1960) y variedades como la Algodona, la Jabonilla y la Flora de la especie S. andigena (Estrada 2000). Además, gracias al cruce entre las variedades Branca Cascuda de S. tuberosum y Pana Blanca de la subespecie andigena, se desarrolló el híbrido o cultivar comercial Diacol Monserrate, el cual presenta buenos rendimientos, aun siendo afectada por la enfermedad (Estrada 2000). Otras variedades con algún grado de resistencia, son la ICA Morita, ICA Única, ICA Zipa y el Clon 88357 (Rodriguez 2000). Sin embargo, en la mayoría de los casos la importancia comercial, las características organolépticas y la resistencia no están correlacionadas. El Grupo de Investigación de la Papa de la Facultad de Agronomía de la Universidad Nacional de Colombia, desarrolló la variedad denominada Pastusa Suprema. Esta variedad es ampliamente cultivada en el departamento de Nariño y en las zonas altas de los departamentos de Cundinamarca y Boyacá (Grupo de Investigación de la Papa, 2010), es útil para fines industriales ya que tiene buena calidad culinaria y presenta alta resistencia al tizón tardío, por lo cual requiere menor cantidad de fungicidas (Jimenez et al., 2009).
Aunque se ha avanzado en el conocimiento de genes de resistencia y en su clonación en variedades susceptibles, no se ha logrado establecer una resistencia duradera al ataque de P. infestans. Además, la comprensión a nivel molecular de cómo actúan y se interrelacionan los diferentes genes para responder al ataque de los patógenos aún no es clara. Todavía no se conoce con exactitud que controla y constituye la diferencia entre una respuesta susceptible o resistente al ataque de P. infestans. No obstante, sí se sabe que el reconocimiento de la infección por P. infestans ocurre durante o después de la invasión de las primeras células de la planta y que la respuesta compatible o incompatible, se da poco después de este reconocimiento (Freytag et al., 1994). Además, es claro que una vez se da el reconocimiento del patógeno, se desencadenan una serie de señales transduccionales que resultan en una reprogramación del metabolismo celular; particularmente, se dan grandes cambios en la transcripción de genes y la incompatibilidad entre hospedero y patógeno deriva en la regulación y expresión de genes en la planta, relacionados con la defensa y la muerte celular localizada (Ros et al., 2008; Beligni, et al., 1999; Lawton y Lamb 1987; McDowell y Woffenden 2003; Nürnberger y Lipka 2005). La activación de los genes relacionados con la defensa es afectado por el genotipo de la cepa de P. infestans que infecte la planta, el cultivar de papa y la proximidad del sitio de inoculación (Wang et al., 2008). Por este motivo, estudios de expresión génica durante la interacción planta- patógeno, sumados al uso de nuevas metodologías para identificar genes que confieran resistencia a P. infestans en genotipos mejorados, silvestres, cultivares y materiales nativos (Jacobs et al., 2010; Ballesteros et al., 2010), ayudarán a comprender los eventos moleculares que determinan una respuesta de resistencia a la infección. Este conocimiento más detallado, sería de gran utilidad para poder avanzar en el mejoramiento genético de la papa y algún día conseguir cultivos con resistencia duradera a la enfermedad.
Entre las principales tecnologías para estudiar la expresión genética se encuentran el Despliegue Diferencial, Análisis en Serie de la Expresión Genética (SAGE) y los Microarreglos de DNA. Mediante los Microarreglos de DNA pueden analizarse varias muestras rápidamente (Kim 2003), pero se requiere cierto conocimiento de las secuencias a analizar (Ibrahim et al., 2005), además son específicos de especie (Liang et al., 2006), requieren de una capacidad instalada altamente costosa y gran habilidad en su aplicación e interpretación (Stein y Liang 2002). Con los SAGE, es posible investigar genes cuya secuencia aún no se conoce (Ibrahim et al., 2005; Liang 2002; Stein y Liang 2002); sin embargo, al ser una técnica basada en la secuenciación, también requieren de una infraestructura apropiada (Stein y Liang 2002; Patino et al., 2002), además pueden presentarse artefactos o perdida de transcritos específicos (Ibrahim et al. 2005). A diferencia de las técnicas anteriores, el Despliegue Diferencial (DD-PCR) no requiere del conocimiento previo de las secuencias, se basa en técnicas de biología molecular sencillas, económicas y de frecuente uso (RT-PCR y PAGE; Velculescu et al., 1995), los resultados son reproducibles (Bauer et al., 1994) y es aplicable a un gran número de sistemas biológicos; en la actualidad, ha sido empleada en el estudio de interacciones planta- patógeno (Bailey et al., 2001; Jagoueix-Eveillard et al., 2001).
Mediante el análisis de los perfiles de expresión generados a partir de interacciones planta-patógeno, bajo diferentes condiciones, es posible identificar genes que se expresan en las interacciones compatibles y no lo hacen en las incompatibles o establecer si la regulación de estos genes varia en ambas situaciones (Pinzón et al., 2009). De este modo podría identificarse genes involucrados en la resistencia, pero también genes involucrados en la susceptibilidad, los cuales son mucho menos conocidos (Henriquez y Fouad Daayf 2010) y que igualmente podrían modificarse con el fin de obtener resistencias (Vogel et al., 2006). Todo lo anterior valida la necesidad de continuar realizando investigaciones en torno a la respuesta de defensa en diferentes patosistemas, en aras de establecer estrategias de control de las enfermedades que afectan las plantas. Por tal razón, el objetivo del presente estudio fue comparar los perfiles de expresión génica de las variedades Pastusa Suprema, Diacol Capiro y Parda Pastusa durante su interacción con P. infestans, utilizando el método del Despliegue Diferencial.
Materiales y métodos
Material vegetal
Se utilizaron plantas de papa de las variedades Pastusa Suprema (resistente al tizón tardío), Diacol Capiro y Parda Pastusa (susceptibles al tizón tardío). Todas fueron multiplicadas in vitro, trasplantadas a macetas con tierra abonada y climatizadas para su cultivo en los invernaderos del Centro de Investigaciones Corpoica - La Selva (Rionegro, Antioquia). Por un periodo de cuatro semanas fueron mantenidas en condiciones promedio de 18°C y 90% de humedad relativa, sin aplicación de fungicidas y verificando periódicamente la ausencia de síntomas visibles de enfermedades.
Aislamiento del Oomiceto e inoculaciones
Como fuente de P. infestans se utilizó un aislamiento obtenido en el Centro de Investigaciones Corpoica - La Selva, a partir de un cultivo de papa infectado (variedad Diacol-Capiro cultivada en la Unión, Antioquia). El asilamiento fue caracterizado para establecer su virulencia usando clones diferenciales de papa portando individualmente genes de resistencia de R1 a R11 y un clon sin genes mayores (r). El factor de virulencia fue determinado como número de diferenciales sintomáticos después de ser inoculados con el aislamiento. La caracterización se realizó por duplicado, en dos pruebas independientes con una semana de diferencia entre ellas. El aislamiento caracterizado fue mantenido en trifolios de Diacol Capiro para posteriores ensayos.
Previo a la inoculación, se seleccionaron trifolios de Diacol Capiro conteniendo abundantes esporangios y esporangióforos de P. infestans, se lavaron con agua destilada y se filtro a través de microporo de 0.45 μm. Finalmente, se preparó la solución de inoculación ajustando a una concentración aproximada de 3x103esporangios/ml usando agua destilada estéril, la concentración fue estimada mediante microscopía.
Se llevaron a cabo infecciones de trifolios del tercio superior de las variedades Pastusa Suprema, Diacol-Capiro y Parda Pastusa con P. infestans bajo condiciones controladas. Para ello, en cada hoja del trifolio se dispusieron dos puntos de infección, cada uno con 30 µl de la solución de inoculación. Finalmente, se incubó en cámara húmeda disponiendo las hojas con el lado abaxial hacia arriba, con fotoperiodo de 12 horas luz, a 18°C y 90% de humedad relativa en promedio. Los controles negativos fueron tratados de igual forma, reemplazando el inoculo por 30 μl agua destilada estéril.
De esta forma se trataron dos trifolios de cada variedad, cada uno con seis puntos de inoculación, para un total de 12 inoculaciones por variedad.
Cada 24 horas post-inoculación, se realizó inspección visual, tanto macroscópica como microscópicamente, en búsqueda de lesiones de la enfermedad, esporangios y esporangióforos. Finalmente, se inocularon rodajas de papa (Tuquerreña) de 7 cm de diámetro con 30 μl de solución de inoculación, disponiendo como soporte un segmento de papel filtro estéril para facilitar la separación del micelio. La incubación se llevó a cabo por cinco días en las mismas condiciones ambientales descritas anteriormente (Correa y Monsalve, 2005).
Aislamiento del RNA y síntesis del DNA copia
Empleando cuchillas estériles, se tomaron muestras de tejido vegetal de aproximadamente 0,25 cm2 alrededor del sitio de inoculación a las 0, 24, 48, 72 y 96 horas post-inoculación (hpi); en el caso de los controles se tomaron muestras de las mismas características a las 0 y 96 horas post-corte (hpc). Adicionalmente, se colectaron esporangióforos y esporangios a partir del papel filtro dispuesto sobre las rodajas de papa, mediante lavados con agua destilada (Correa y Monsalve, 2005).
Se extrajo el RNA total, tanto del material vegetal como de los esporangióforos y esporangios, mediante TRIzol®(Invitrogen Corporation, CA) siguiendo las especificaciones del fabricante. La concentración del RNA extraído y su pureza fueron estimadas mediante espectrofotometría utilizando el NanoDrop® ND-2000 y electroforesis en gel de agarosa al 1,2% en condiciones desnaturalizantes, teñido con bromuro de etidio (10mg/ml) y visualizado bajo luz UV. Posteriormente, el RNA se trató con DNasaI® (Fermentas, USA) y se purificó nuevamente con TRIzol®.
Para la síntesis del DNA complementario (cDNA), se utilizó el kit RETROscript® (Ambion, USA) siguiendo las especificaciones del fabricante. Como control se amplificó una región transcrita del gen que codifica para la nitrato reductasa con un tamaño esperado de 230 pb. La mezcla de reacción se preparó como se describe a continuación: 2 µl del cDNA, 2.5 µl de DMSO, 0,2 µl de Taq Pol (5 U/µl; Fermentas, USA), 2 µl de dNTPs (2mM; Fermentas, USA), 4 µl de MgCl2 (25mM; Fermentas, USA), 5 µl Buffer Taq con (NH4)2SO4 (10X; Fermentas, USA), 1 µl de iniciador en sentido (10 µM; 5' TGAATGTGTGCAAGCCTCACAAG 3'), 1 µl de iniciador en antisentido (10 µM; 5'ATGTCCATGGACTATGATCCAAG 3'), finalmente, se ajustó a un volumen total de 50 µl. La reacción se llevó a cabo con el siguiente perfil térmico: una pre-desnaturalización a 94°C durante 7 minutos; seguida de 35 ciclos a 94°C por 30 segundos, 58°C por 30 segundos y 74°C por 30 segundos, y una extensión final a 74°C durante 5 minutos. Para visualizar el resultado de la amplificación, el producto de PCR fue sometido a electroforesis en gel de agarosa al 0,8%, teñido con bromuro de etidio y visualizado bajo luz UV.
Despliegue diferencial (DD-PCR)
El cDNA fue usado como templado para el despliegue diferencial (DD-PCR). La mezcla de reacción fue la siguiente: 1µl de cDNA, 2,5 µL de DMSO, 1,0 µl de Taq Pol (5 U/µl; Fermentas, USA), 1,25 µl de dNTPs (2mM; Fermentas, USA), 6 µl de MgCl2 (25mM; Fermentas, USA), 5 µl Buffer Taq con (NH4)2SO4 (10X; Fermentas, USA), 1 µl del coctel de iniciadores en sentido (6,25 pM/ µl), 1 µl del coctel de iniciadores en antisentido (10 µM) y finalmente, se ajustó a un volumen total de 50 µl. Los cocteles de iniciadores constaron de 9 oligos en sentido (P) y 9 Oligo(dT) en antisentido (T), sintetizados por MWG-Biotech AG (Alemania), cuyas secuencias son:
P1: 5'-ATTAACCCTCACTAAATGCTGGGGA-3'; P2: 5'-ATTAACCCTCACTAAATCG GTCATAG-3'; P3: 5'-ATTAACCCTCACTAAATGCTGGTGG-3'; P4: 5'-ATTAACCCTC ACTAAATGCTGGTAG-3'; P5: 5'-ATTAACCCTCACTAAAGATCTGACTG-3'; P6: 5'-ATTAACCCTCACTAAATGCTGGGTG-3'; P7: 5'-ATTAACCCTCACTAA ATGCTGT ATG-3'; P8: 5'-ATTAACCCTCACTAAATGGAGCTGG-3'; P9: 5'-ATTAACCCTC ACTAAATGTGGCAGG-3'; P10: 5'-ATTAACCCTCACTAAAGCACCGTCC-3'; T1:5'-CATTATGCTGAGTGATATCTTTTTTTTTAA-3'; T2: 5'-CATTATGCTGAGTGATATC TTTTTTT TTAC-3'; T3: 5'-CATTATGCTGAGTGATATCTTTTTTTTTAG-3'; T4: 5'-CA TTATGCTGAGTGATATCTTTTTTTTTCA-3'; T5: 5'-CATTATGCTGAGTGATATCTT TTTTTTTCC-3'; T6: 5'-CATTATGCTGAGTGATATCTTTTTTTTTCG-3'; T7: 5'-CATT ATGCTGAGTGATATCTTTTTTTTTGA-3'; T8: 5'- CATTATGCTGAGTGATATCTTT TTTTTTGC-3'; T9: 5'-CATTATGCTGAGTGATATCTTTTTTTTTGG-3'.
El perfil térmico usado fue: un primer ciclo a 94°C por 5 minutos, 40°C por 5 minutos y 74°C por 5 minutos; seguido de dos ciclos a 94°C por 2 minutos, 40°C por 5 minutos y 74°C por 5 minutos; 37 ciclos a 94°C por 1 minuto, 60°C por 1 minuto y 74°C por 2 minutos y una extensión final a 74°C durante 7 minutos.
Los fragmentos amplificados por DD-PCR fueron separados mediante electroforesis en gel de poliacrilamida al 6% en condiciones desnaturalizantes, utilizando una relación acrilamida/bis-acrilamida 38:2 y urea 7M. Previo a la electroforesis, 10 μl de cada muestra se mezclaron con 5 μl de buffer carga (urea 8 M, EDTA 20 mM, TrisHCl 5 mM pH 7,5, xilencianol 0,5% y azul de bromofenol 0,5%), se incubaron a 94°C durante 5 minutos y posteriormente en hielo por 1 minuto. La electroforesis se realizó en un Sequen-Gen® GT Nucleic Acid Electrophoresis Cell a 1.600 V por 2 horas, usando buffer TBE 1X.
La visualización de las bandas se realizó mediante tinción con plata. Inicialmente, se incubó el gel en etanol al 10% durante 8 minutos con agitación eventual, luego se sumergió en ácido nítrico 1% por 8 minutos y se lavó con agua desionizada; posteriormente se sumergió en la solución de tinción (2,5 g de nitrato de plata, 3,75 ml de formaldehido al 37% en 2l de agua desionizada) durante 20 minutos. Después se sumergió en solución reveladora (60 g de carbonato de sodio, 3 ml de formaldehido al 37%, 400 µl de tiosulfato de sodio y 2l de agua desionizada) pre-enfriada a 10°C con agitación constante hasta visualizar las bandas. Finalmente, se incubó en ácido acético al 10% por 3 minutos y un lavado final con agua desionizada. Se secó el gel a temperatura ambiente y la imagen se digitalizó con un scanner PowerLook 2100XL®.
El establecimiento de patrones y selección de bandas diferencialmente expresadas, se llevó a cabo por comparación visual de las imágenes digitalizadas directamente y mediante el Software Doc-ItLS Image Analysis®. Se contrastaron las condiciones infectadas (susceptibles y resistentes) y no infectadas, a las diferentes horas de muestreo. Adicionalmente, se comparó con el patrón de bandeo del patógeno.
Elusión de bandas a partir del gel de poliacrilamida y clonación
Los fragmentos seleccionados por su expresión diferencial fueron separados físicamente del gel y depositados en sendos tubos, se adicionó 20 μl de buffer TE y se incubó a 100ºC por 5 minutos. Posteriormente, se centrifugó a 8000 rpm por 2 minutos y se recuperó el sobrenadante. La integridad de los eluidos se verificó mediante re-amplificación por PCR, utilizando 1μl como templado y las condiciones descritas anteriormente. Los productos de la re-amplificación fueron sometidos a electroforesis en gel de agarosa al 0,8%, teñido con bromuro de etidio y visualizado bajo luz UV. La integridad de los extremos fue re-establecida como se describe: 3µl de cDNA, 0,2 µl de Taq Pol (5 U/µl; Fermentas, USA), 1 µl de dATP (2mM; Fermentas, USA), 0,6 µl de MgCl2 (25mM; Fermentas, USA), 1 µl Buffer Taq con (NH4)2SO4 (10X; Fermentas, USA), en un volumen total de 10 µl e incubación a 70°C durante 30 minutos.
Para la clonación se utilizó el kit QIAGEN PCR Cloning, siguiendo las instrucciones del fabricante, con condiciones de ligación de 4°C por 12 horas. 10 µl del producto de ligación se incubaron con 100 µl de células E. coli cepa DH5αF' calcio competentes en hielo por 20 minutos. Se procedió con el choque térmico a 42°C por 2 minutos y 10 minutos en hielo; luego se adicionó 1 ml de medio LB líquido y se incubó a 37°C por 2 horas con agitación constante. Finalmente, se seleccionó mediante incubación en medio LB sólido adicionado de X-Gal, IPTG, ampicilina y kanamicina a 37°C por 12 horas.
Secuenciación y análisis de secuencias
Los plásmidos recombinantes fueron recuperados mediante el sistema de lisis alcalina (Sambrook et al., 1989), verificados por electroforesis en gel de agarosa al 0,8%, teñido con bromuro de etidio y visualizado bajo luz UV. La secuenciación se realizó en Macrogen USA Corp., mediante el sistema de "single extension".
Las secuencias se editaron utilizando el software VectorNTI® y se analizaron empleando el software Blast2GO®(http://www.blast2go.com), el cual permite encontrar secuencias homologas en la base de datos pública del Centro Nacional para la Información Biotecnológica (NCBI) mediante algoritmos blast y asignar las anotaciones génicas a dichas secuencias homologas empleando diferentes algoritmos y bases de datos, como Gen Ontology (GO), Enzyme code, InterProScan y KEGG. Para realizar el blast, se utilizaron los parámetros de configuración del software: umbral de e-value 1 e-3 y una recuperación de 20 hits por secuencia.
Resultados y discusión
Caracterización del Oomiceto y establecimiento de la infección
El aislado de P. infestans empleado fue colectado a partir de un cultivo de Diacol Capiro infectado. Después de ser purificado y caracterizado el patotipo fue catalogado como CORPOICA - "La Unión-2". En el primer ensayo de caracterización la virulencia se evidenció por la reacción de compatibilidad con los diferenciales R 1,2,3,4,7,8,10 y 11. Los resultados fueron reproducidos en el segundo ensayo, excepto por la incompatibilidad con el diferencial R11 (figura 1).
P. infestans continuamente gana o pierde su virulencia con respecto a los genes R durante la reproducción asexual (posiblemente debido a mutaciones, entrecruzamientos mitóticos y/o influencia de elementos extra-cromosomales). Esta podría ser una de las causas por las cuales, a partir de algunos aislamientos de zoosporas se obtienen progenies que difieren en virulencia con respecto a sus parentales (Samen et al., 2003). Sin embargo, se debe recordar que en Colombia no ha sido reportada la reproducción sexual del oomiceto, aun así se presenta un grado de variación importante, no sólo cuando el patógeno es mantenido en rodajas de papa (Correa y Monsalve, 2005) sino también cuando es incubado en medios de cultivo o almacenado en nitrógeno líquido. Es por ello que este aspecto debe ser considerado con mayor detalle en el diseño de estrategias de control basadas en el uso de los genes R.
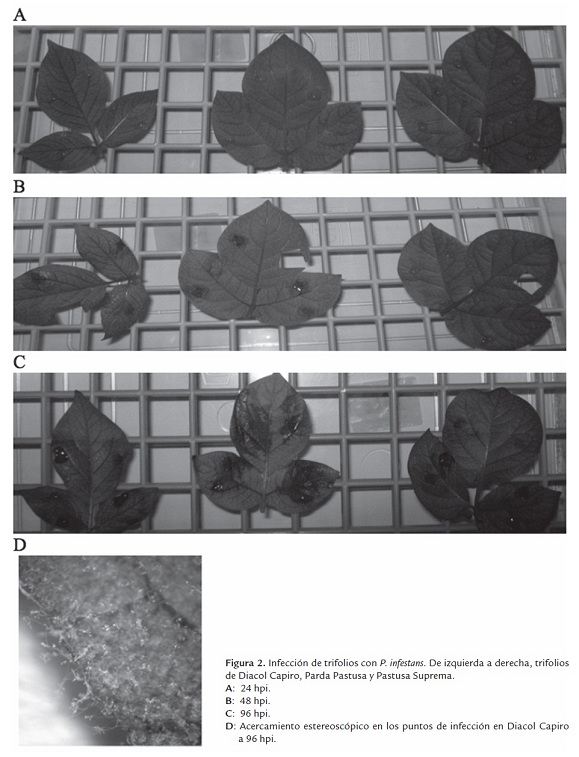
A pesar de la variación, la infección de los trifolios fue exitosa en todos los puntos de inoculación. Una vez identificadas las lesiones, éstas fueron examinadas en más detalle a fin de confirmar la esporulación del oomiceto en las hojas (figura 2D).
A las 24 hpi no se observaron síntomas en ninguna de las variedades evaluadas (figura 2A). Sin embargo, a las 48 hpi fueron evidentes los primeros síntomas en algunos trifolios de las variedades Diacol Capiro y Parda Pastusa, estas lesiones se restringieron al lugar de inoculación, a pesar de ello, no se observó esporulación. Al cabo de 72 hpi se observaron lesiones esporulantes en todos los trifolios de las mismas variedades. En este momento también fueron evidentes los primeros síntomas en algunos sitios de inoculación de la variedad Pastusa Suprema pero sin esporulación (figura 2B). Después de 96 hpi, en Diacol Capiro y Parda Pastusa las lesiones esporulantes se expandieron abarcando casi toda la hoja; mientras que en Pastusa Suprema sólo en los pocos trifolios que exhibían los síntomas se hicieron evidentes algunas lesiones esporulantes (figura 2C). Finalmente, pasadas 120 hpi todos los sitios de inoculación de las tres variedades presentaron lesiones esporulantes, siendo más severas y expandidas en las variedades Diacol Capiro y Parda Pastusa.
Estos resultados muestran, de acuerdo a reportes previos, que la variedad Pastusa Suprema no es inmune a P. infestans, pero sí presenta un retraso en la aparición de los síntomas y la formación de las estructuras reproductoras del oomiceto con respecto a otras variedades, comportamiento que se ajusta a la definición de tolerancia (Elliott, 1996) y razón por la cual este genotipo ha sido catalogado como resistente a la enfermedad.
Esta tolerancia fue demostrada por Betancourth et al. (2008) al evaluar la respuesta de nueve genotipos de papa frente a la infección con P. infestans. Estos investigadores encontraron que Pastusa Suprema, además de no der afectada sensiblemente en su índice de área foliar, mantiene su rendimiento y el número de tubérculos por planta superiores a los obtenidos en las demás variedades. No obstante, se ha observado que la resistencia se ha venido perdiendo, probablemente por la ganancia de nuevos genes de virulencia.
Perfiles de expresión
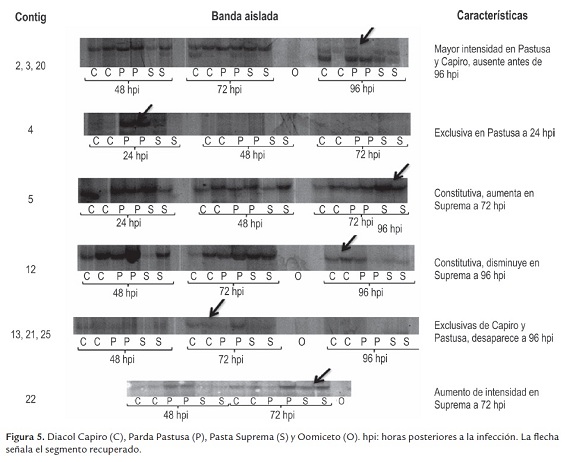
El cDNA obtenido fue de buena calidad y cantidad, óptimo para la generación de perfiles de expresión. Se evaluaron diferentes combinaciones de oligos (dato no mostrado); sin embargo, el mayor número de bandas diferenciales fue obtenido cuando se empleó un coctel de iniciadores que incluyó todos los oligos en sentido (P) y todos los Oligo-dT en antisentido (T), obteniendo alrededor de 30 bandas de interés. Estas bandas revelaron diferencias en la expresión génica a distintos tiempos post-inoculación, incluyendo la presencia y ausencia de bandas y el aumento o disminución de su intensidad; adicionalmente, fueron reproducidas en las dos repeticiones llevadas a cabo, por lo cual fueron catalogadas como posibles determinantes en la interacción P. infestans - S. tuberosum.
A través del tiempo se pudieron observar cambios en el nivel de expresión. Por ejemplo, en el segmento a de lafigura 3, se resalta la disminución de la intensidad de una de las bandas de expresión basal, a las 24 hpi la señal es muy intensa y se va reduciendo en forma paulatina hasta las 96 hpi; la posibilidad que este efecto en la expresión sea debida al corte de los trifolios (u otra razón externa distinta a la infección) queda fuera al observar que el patrón se conserva entre los perfiles a las 96 horas con y sin infección.
También se observó la aparición de bandas como respuesta a la infección. En los segmentos b y b' de la figura 3, se observa una banda diferencial presente únicamente en las variedades Parda Pastusa y Diacol capiro y ausente en Pastusa Suprema. Esta banda es perceptible a las 96 hpi y nunca se detectó en la condición sin infectar; esto hace suponer que la expresión de este gen en particular, pueda de alguna forma favorecer la susceptibilidad de la planta ante el ataque de P. infestans.
De otro lado, en los segmentos c y c' se observa lo contrario. La banda se encuentra presente en todas las variedades a las 96 hpc, 24, 48, 72 y 96 hpi, pero sólo en la variedad Parda Pastusa a las 96 hpi deja de ser detectada. Como en el caso anterior, se podría especular que corresponde a un posible gen que favorezca la susceptibilidad, pero con un efecto de silenciamiento génico, mas no de activación.
Los cambios en la expresión génica también se aprecian en la variedad Pastusa Suprema. En los segmentos d y d' se señala una banda que está presente en todos los tiempos pos-infección pero es particularmente fuerte en la variedad Pastusa Suprema a las 96 hpi. Esta misma banda es casi imperceptible en la condición sin infectar. Lo anterior hace pensar que la sobre-expresión de este gen pueda estar involucrada en algún mecanismo de defensa.
A las 48 hpi, en el fragmento e se resalta una banda de elevada expresión en las variedades reportadas como susceptibles, mientras que en la variedad Pastusa Suprema su expresión es relativamente más baja; siendo mucho más intensa en Parda Pastusa que en Diacol Capiro y en esta última es más intensa que en Pastusa Suprema. Es posible que en el caso de la variedad Pastusa Suprema este comportamiento esté relacionado con el retraso de la aparición de síntomas. Para las tres variedades, las mayores diferencias se observaron a las 72 y 96 horas post-inoculación. Esto demuestra una vez más, que el reconocimiento y la defensa de S. tuberosum ante P. infestansimplica una regulación y expresión génica, compleja y dinámica, que incluye tanto la supresión como la sobre-expresión de genes. Estos resultados son similares a los obtenidos por Restrepo et al. (2005) cuando evaluaron los perfiles de expresión génica durante interacciones compatibles e incompatibles entre S. tuberosum y P. infestans empleando microarreglos. Estos investigadores también encontraron que a medida que aumenta el tiempo post-inoculación se incrementa el número de genes expresados diferencialmente y que además, los genes expresados diferencialmente durante las primeras horas de la infección (6 y 12 hpi) correspondieron a genes que fueron inducidos, mientras que durante las 48 y 72 hpi fue mayor la cantidad de genes reprimidos que inducidos.
Por su parte, Abu-Nada et al. (2007), al evaluar la dinámica temporal de metabolitos relacionados con la patogénesis, encontraron que estos fueron detectados a niveles altos a las 24 y 48 horas post-inoculación, mientras que la abundancia de estos metabolitos decreció 96 horas post-inoculación, indicando el colapso de las respuestas de defensa primaria y secundaria. Aunque esta es una dinámica del metabolismo, y no directamente de la expresión diferencial de genes particulares, se podría pensar en la expresión diferencial de genes clave reguladores de las rutas metabólicas; sin embargo, no se debe dejar de lado que la temporalidad de acumulación de los metabolitos no es coincidente con la de los RNAm.
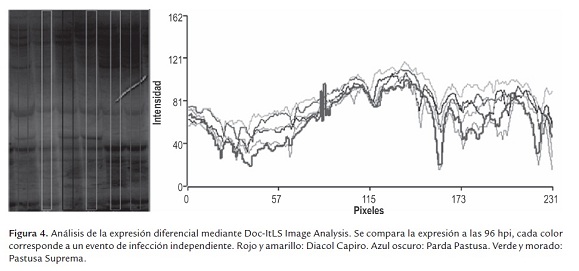
La diferencia entre los perfiles de expresión fue corroborada mediante el análisis de las imágenes digitalizadas usando el software Doc-ItLS Image Analysis® (figura 4). Adicionalmente, este análisis permitió demostrar la reproducibilidad de los ensayos, ya que las muestras individuales de cada variedad tuvieron un comportamiento similar y difirieron entre cada una de las variedades.
En todas las evaluaciones realizadas, se pudo constatar que la variedad Pastusa Suprema presenta un nivel de expresión comparativamente más alto que las demás; en tanto que el menor nivel se presenta en la variedad Diacol Capiro. Esta expresión en Pastusa Suprema resulta interesante ya que la variedad corresponde a un hibrido, relativamente nuevo, que posee una gran complejidad genética dado que tiene por parentales tres especies diferentes; el parental materno fue un hibrido inter-específico resultante de Solanum stoloniferum 230490 (variedad silvestre) × Solanum phureja var. Yema de huevo, y el parental paterno fue Solanum tuberosum sp. andigena var. Parda pastusa (López y Chaparro 2007). Adicionalmente, es de esperar que esta variedad posea importante información en referencia a la respuesta de resistencia ante la infección con P. infestans, dado que en los genomas de ambas especies del parental materno se han identificado genes R y QTLs, de gran relevancia para la resistencia a P. infestans (Lindqvist-kreuze et al., 2010). Los resultados presentados demuestran que en la variedad Pastusa Suprema existen genes que se sobre-expresan y se silencian durante la infección con P. infestans. Como resultado de estas variaciones génicas, la planta lograría su característica de tolerar el patógeno; y por ende, el estudio detallado de esta variedad, podría brindar nueva información de gran utilidad en la comprensión molecular del mecanismo de defensa de la planta.
Secuenciación y análisis de los fragmentos diferencialmente expresados
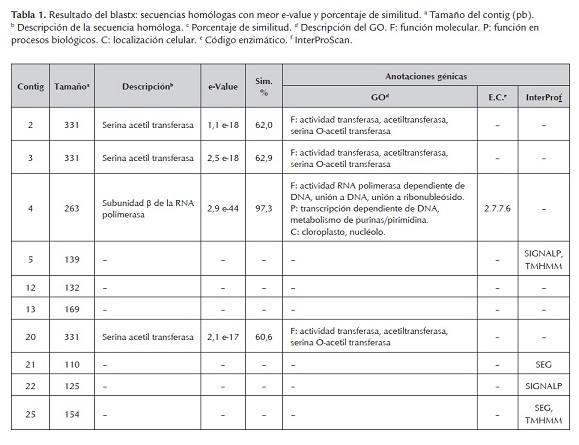
De las bandas identificadas como diferencialmente expresadas, 20 fueron clonadas y secuenciadas. Una vez editadas las secuencias, solo se obtuvieron 10 contig, los cuales fueron analizados a través del software Blast2go. Para hacer la búsqueda de las secuencias homologas, se realizaron comparaciones contra la base de datos no redundante del Centro Nacional para la Información Biotecnológica (NCBI), empleando los algoritmos blastn y blastx. En la figura 5 se muestran los contig que fueron analizados, así como la descripción de la banda a partir de la cual se realizó la clonación y secuenciación.
Al realizar el blastx, fue posible hacer las anotaciones génicas de 8 contig. Las secuencias 12 y 13 no pudieron ser anotadas. Para las secuencias 2, 3, 4 y 20 se encontraron homologías con la información contenida en la base de datos del NCBI y pudo realizarse la respectiva anotación de estas secuencias, identificando su función molecular, su función en los procesos biológicos y su localización subcelular (ontología génica -GO-); inclusive, la secuencia 4 pudo ser relacionada con el código enzimático y determinarse su participación en rutas metabólicas según la base de datos KEGG. Por su parte, para las secuencias 5, 21, 22 y 25 no pudo encontrarse su ontología génica pero tuvieron homología con la información contenida en la base de datos InterProScan y fueron relacionadas con características identificables de familias, dominios o sitios funcionales de proteínas conocidas (tabla 1).
Se encontró gran variación en las especies a las que pertenecen las secuencias con las que se presentaron los aciertos de los contig, al hacer el blastx; sin embargo, las de mayor relevancia para llevar a cabo las anotaciones genicas, fueron Curvibacter putative y Solanum panduriforme (tabla 1).
Los contig 2, 3 y 20 tuvieron homología con una secuencia que codifica para una serina acetiltranferasa (SAT). SAT es una enzima involucrada en el metabolismo del azufre (López-Martín et al., 2008; Kopriva et al., 2009), que se encuentra presente en plantas, hongos y microorganismos capaces de asimilar el azufre inorgánico presente en la naturaleza, e incorporarlo a sus compuestos orgánicos (Kopriva et al., 2009; Haas et al., 2008). En plantas, esta enzima ha sido encontrada en el citosol, mitocondria y plástidos (Harms et al., 2000; Haas et al., 2008; López-Martín et al., 2008). El producto final de la asimilación del azufre es la cisteína y a partir de este aminoácido se sintetizan, directa o indirectamente, otras bio-moléculas que tienen azufre en su estructura como glutatión, metionina y fitoalexinas (Harms et al., 2000; Riemenschneider et al., 2005). La cisteína y la metionina son aminoácidos fundamentales para la estructura y función de las proteínas, co-enzimas y grupos prostéticos; además, los compuestos sulfurados están involucrados en las respuestas a estrés biótico y abiótico, por ejemplo el glutatión, que hace parte de la detoxificación de las especies reactivas de oxígeno (ROS) (Davidian y Kopriva, 2010). Wirtz y Hell (2006) evaluaron la expresión de una SAT bacteriana en plantas transgénicas de papa y encontraron que la sobreexpresión de la enzima indujo el incremento de los niveles de cisteína y glutatión en dichas plantas.
La secuencia del contig 4 tuvo homología con una secuencia que codifica para la subunidad Β de la RNA polimerasa. La transcripción de los genes que se encuentran en el genoma de los plástidos es realizado por múltiples subunidades de RNA polimerasas dependientes de DNA, cuyas propiedades y funciones no se conocen bien (Troxler et al., 1994). En el genoma de los cloroplastos de las plantas, existen diferentes genes (rpoA, rpoB, rpoC1 y rpoC2) que codifican para proteínas homologas a las subunidades α, Β, Β' y Β'' de las RNA polimerasa de las bacterias (Dubey et al., 2009). En los tilacoides de la membrana de los cloroplastos, se encuentra el sistema transportador de electrones fotosintético y allí es donde se producen la mayor cantidad de ROS (Kariola et al., 2005); por lo tanto, el cloroplasto tiene un papel muy importante durante la resistencia a enfermedades y la respuesta de hipersensibilidad celular que se desencadena cuando se presenta una interacción planta-patógeno incompatible (Kariola et al., 2005; Dubey et al., 2009). Dubey et al. (2009) al evaluar el genoma del cloroplasto de dos variedades de plantas, con diferente respuesta ante la acción de un patógeno, encontraron que la única diferencia eran cuatro sustituciones nucleótidicas (SNP) en el gen rpoC1, que codifica para la subunidad Β' de la RNA; los investigadores concluyeron que es posible que esta diferencia sea la responsable de la respuesta susceptible o resistente ante el fitopatógeno.
En cuanto a los resultados del InterProScan, se encontró que algunas secuencias, después de ser traducidas, tendrían homología con regiones de baja complejidad (SEG), dominios transmembranales (TMHMM) o péptidos señal (SIGNALP). Este dominio transmembranal conlleva un gran interés dado que durante la inmunidad innata de las plantas existen proteínas encargadas de reconocer los patrones moleculares asociados a patógenos (PMAP) y desencadenar así la respectiva respuesta de defensa; en algunos casos, estos receptores de PMAPs están formados por dominios transmembranales, como es el caso de los receptores semejantes a Toll, FLS2, RLP y CEBiP (Castro-Mercado y García-Pineda, 2009; Ojito-Ramos y Portal, 2010).
Conclusiones
Evaluando los perfiles de expresión generados a partir de interacciones planta-patógeno, bajo diferentes condiciones, es posible identificar genes que se expresan en las interacciones compatibles y no lo hacen en las incompatibles, o viceversa. Así, esta herramienta permite clarificar que genes están involucrados en la resistencia; pero además, pueden conocerse genes involucrados en la susceptibilidad, los cuales son mucho menos conocidos y que igualmente podrían ser usados en la búsqueda de estrategias de resistencia. Los estudios de expresión génica durante la interacción planta- patógeno que se realizaron, demuestran que las diferencias en la expresión durante las interacciones compatibles e incompatibles incluyen una variedad de respuestas como la sobre-expresión, el silenciamiento y los cambios en el nivel de expresión. Puede suponerse que estas variaciones son responsables de la resistencia a la enfermedad, pero del mismo modo también podrían serlo de la susceptibilidad, por lo cual es necesario continuar con estudios más detallados. El análisis las secuencias reveló que los fragmentos sobre-expresados en las variedades susceptibles, tienen homología con una serina-acetiltranferasa y la subunidad Β de la RNA polimerasa, mientras que los fragmentos sobre-expresados en la variedad resistente, tienen homología con un dominio transmembranal, sugiriendo que estas secuencias corresponden a genes involucrados en la respuesta de S. tuberosum ante el ataque de P. infestans.
Agradecimientos
Agradecemos al soporte financiero del proyecto "Generación de marcas de secuencias expresadas (ESTS) a través de la técnica de despliegue diferencial en las interacciones papa - P. infestans y banano - M. Fijiensis", financiado por el CODI, Vicerrectoría de Investigación, Universidad de Antioquia; Cesar Julián López, Albeiro Macias y Javier Correa por su apoyo en la presente investigación. A CORPOICA por su contribución en las infecciones.
Referencias bibliográficas
1 Abad, G., Abad, J., Ochoa, C. 1995. Historical and scientific evidence that supports the modern theory of the Peruvian Andes as the centre of origin of Phytophthora infestans. En: LJ Dowley, E Bannon, LR Cooke, T Keane, and E O'Sullivan (eds.). Phytophthora infestans 150. Ireland: Boole Press Ltd. pp. 239-245.
2 Abu-Nada, Y., Kushalappa, A.C., Marshall, W.D., Al-Mughrabi, K., Murphy, A. 2007. Temporal dynamics of pathogenesis-related metabolites and their plausible pathways of induction in potato leaves following inoculation with Phytophthora infestans. European Journal of Plant Pathology 118: 375-391.
3 Agrios, G. 2005. Plant pathology. 5th ed. Nueva York: Editorial Elsevier Academic Press. pp. 922.
4 Agrocadenas. 2006. La cadena de la papa en colombia. Una mirada global de su estructura y dinámica (1991-2005) [sitio web]. Bogotá DC (Colombia): Ministerio de agricultura y desarrollo rural. Documento de trabajo N° 100; Enero de 2006 [fecha de acceso: Febrero de 2010]. Disponible en:http://www.agrocadenas.gov.co.
5 Bailey, A. M., Muñoz-Sánchez, C. I., Martínez-Hernández, P., Manríquez-Escamilla, M. 2001. Detection of Additional Gene Products Induced by the Interaction between Phytophthora capsici and its host Capsicum annuum and the identification of an Elicitin-Encoding Gene. Revista Mexicana de Fitopatología 19: 22- 31.
6 Ballesteros, D.C., Gómez, G., Delgado, M.C., Álvarez, M.F., Juyó, D., Cuéllar, D., y Mosquera, T. 2010. Posible presencia de un gen R1 en germoplasma de Solanum tuberosum Grupo Phureja. Agronomía Colombiana 28:137-146.
7 Bauer, D., Warthoe, P., Rohde, M., Strauss, M. 1994. Detection and differential display of expressed genes by DDRT-PCR. PCR methods and applications 4: S97-108.
8 Beligni, M.V., Laxalt, A.M., Lamattina, L. 1999. Temporal and spatial patterns of GAPDHc mRNA accumulation during an incompatible potato- Phytophthora infestans interaction. Comparison with a compatible interaction.Physiologia Plantarum 105: 280-287.
9 Betancourth, C., Portilla, E., Salas, H. 2008. Evaluación de la reacción de nueve genotipos de papa (Solanum tuberosum subsp. andigena) al ataque de Phytophthora infestans (Mont) de Bary. Agronomía Colombiana 26: 411-416.
10 Bormann, C., Rickert, A.M., Castillo, R.A., Paal, J., Lübeck, J., Strahwald, J., Buhr, K., Gebhardt, C. 2004. Tagging quantitative trait loci for maturity-corrected late blight resistance in tetraploid potato with PCR-based candidate gene markers. Molecular plant-microbe interactions 17: 1126-1138.
11 Cevipapa. 2010. [sitio web]. Colombia. [Fecha de acceso: Febrero 6 de 2010]. Disponible en:http://www.cevipapa.org.co/
12 Castro-Mercado, E. y García-Pineda, E. La inmunidad innata en las : una batalla molecular entre receptores y estimuladores. Revista Biológicas 11: 43- 47.
13 Collard, B., Jahufer, M., Brouwer, J., Pang, E. 2005. An introduction to markers, quantitative trait loci (QTL) mapping and marker-assisted selection for crop improvement: The basic concepts. Euphytica 142: 169-196.
14 Colon, L.T., Jansen, R.C., Budding, D.J. 1995a. Partial resistance to late blight (Phytophthora infestans) in hybrid progenies of four South American Solanum species crossed with diploid S. tuberosum. Theoretical and Applied Genetics 90: 691-698.
15 Colon, L.T., Turkensteen, L.J., Prummel, W., Budding, D.J., Hoogendoorn, J. 1995. Durable resistance to late blight (Phytophthora infestans) in old potato cultivars. European Journal of Plant Pathology 101: 387-397.
16 Cornelissen, B.J.C., Melchers, L.S. 1993. Strategies for Control of Fungal Diseases with Transgenic Plants.Plant physiology 101: 709-712.
17 Correa, J., Monsalve, Z. 2005. Patrón de expresión genética durante la interacción de Solanum tuberosum var. Parda pastusa y var. Tuquerreña con Phytophthora insfestans. Trabajo de Grado. Facultad de Ciencias Exactas, Instituto de Biología, Universidad de Antioquia, Medellin, Colombia.
18 Davidian, J.C., Kopriva, S. 2010. Regulation of Sulfate Uptake and Assimilation - the Same or Not the Same?Molecular Plant 3 (2): 314-325.
19 Donaire, E. R., García, W. 2006. Alternativa agroecológica para el control del tizón tardío, Phytophthora infestans, de la papa en Colomi - Bolivia. Acta Nova 3: 564-577.
20 Díaz, M., Fajardo, D.A., Moreno, J.D., García, C., Nuñez, V.M. 2003. Identificación de Genes R1 y R2 que confieren resistencia a Phytophthora infestans en genotipos colombianos de papa. Revista Colombiana de Biotecnología 5: 40-50.
21 Dubey, M.K., Shasany, A.K., Dhawan, O.P., Shukla, A.K., Khanuja, S.P.S. 2009. Genetic Variation revealed in the chloroplast-encoded RNA polymerase Β' Subunit of downy mildew - resistant genotype of opium poppy.Journal of Heredity 100 (1): 76-85.
22 Elliott, F. 1996. Mejoramiento de plantas citogenéticas. México: Continental.
23 Estrada, N. 2000. Mejoramiento genético de la papa para resistencia al tizón.Papas Colombianas 2000 con el mejor entorno ambiental 3: 38-39.
24 FAO. 2010. Crops statistics database. Disponible en: http://faostat.fao.org/
25 Freytag, S., Arabatzis, N., Hahlbrock, K., Schmelzer, E. 1994. Reversible cytoplasmic rearrangements precede wall apposition, hypersensitive cell death and defense-related gene activation in potato/Phytophthora infestansinteractions. Planta 194: 123-135.
26 Fry, W. 2008. Phytophthora infestans: the plant (and R gene) destroyer. Molecular Plant Pathology 9: 385-402.
27 Giraldo, D., Juarez, H., Pérez, W., Trebejo, I., Yzarra, W., Forbes, G. 2010. Severity of the potato late blight (Phytophthora infestans) in agricultural areas of Peru associated with climate change. Revista Peruana Geo-Atmosférica 67: 56-67.
28 Grupo de Investigación de la Papa, Facultad de Agronomía de la Universidad Nacional de Colombia. Catálogo de variedades colombianas. [sitio web]. [fecha de acceso: Febrero de 2010]. Disponible en:http://papaunc.com/catalogoExtendido.shtml?x=44
29 Guzmán, J., Thurston, H.D., Heidrick, L.E. 1960. Resultados sobre la naturaleza de la resistencia parcial de tres clones de papa a Phytophthora infestans (Mont) De Bary. Agricultura Tropical 16: 89-99.
30 Haas, F.H., Heeg, C., Queiroz, R., Bauer, A., Wirtz, M., Hell, R. 2008. Mitochondrial serine acetyltransferase functions as a pacemaker of cysteine synthesis in plant cells. Plant physiology 148 (2): 1055-1067.
31 Haas, B., Kamoun, S., Zody, M.C., Jiang, R.H., Handsaker, R.E., Cano, L.M., Grabherr, M. et al., 2009. Genome sequence and analysis of the Irish potato famine pathogen Phytophthora infestans. Nature 461: 393-398.
32 Harms, K., Ballmoos, P.V., Brunold, C., Höfgen, R., Hesse, H. 2000. Expression of a bacterial serine acetyltransferase in transgenic potato plants leads to increased levels of cysteine and glutathione. The Plant Journal 22 (4): 335-343.
33 Henriquez, M.A., Daayf, F. 2010. Identification and cloning of differentially expressed genes involved in the interaction between potato and Phytophthora infestans using a subtractive hybridization and cDNA-AFLP combinational approach. Journal of integrative plant biology 52: 453-467.
34 Ibrahim, A., Hedley, P.E., Cardle, L., Kruger, W., Marshall, D.F., Muehlbauer, G.J., Waugh, R. 2005. A comparative analysis of transcript abundance using SAGE and Affymetrix arrays. Functional & integrative genomics 5: 163-174.
35 Jacobs, M., Vosman, B., Vleeshouwers, V., Visser, R.G.F., Henken, B., Van den Berg, R. 2010. A novel approach to locate Phytophthora infestans resistance genes on the potato genetic map. Theoretical and applied genetics. 120: 785-796.
36 Jagoueix-Eveillard, S., Tarendeau, F., Guolter, K., Danet, J.L., Bové, J.M., Garnier, M. 2001. Catharanthus roseus genes regulated differentially by mollicute infections. Molecular plant-microbe interactions 14: 225-233.
37 James, R., Fry, W. 1983. Potential for Phytophthora infestans populations to adapt to potato cultivars with rate-reducing resistance. Phytopathology 73: 984-988.
38 Jaramillo, S. 2003. Monografía sobre Phytophthora infestans (Mont) De Bary. Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, Medellin, Colombia.
39 Jimenez, J., Chaparro-Giraldo, A., Blanco, J. 2009. Evaluación de diferentes combinaciones fitohormonales en la regeneración de Solanum tuberosum (Solanaceae) Var. Pastusa Suprema a partir de explantes internodales.Revista Colombiana de Biotecnología 11: 67-74.
40 Joos, J.H., Hahlbrock, K. 1992. Phenylalanine ammonia-lyase in potato (Solanum tuberosum L.). European Journal of Biochemistry 204:621-629.
41 Kim, H.L. 2003. Comparison of oligonucleotide-microarray and serial analysis of gene expression (SAGE) in transcript profiling analysis of megakaryocytes derived from CD34+ cells. Experimental & molecular medicine 35: 460-466.
42 Kariola, T., Brader, G., Li, J., Tapio-Palva, E. 2005. Chlorophyllase 1, a Damage control enzyme, affects the balance between defense pathways in plants. The Plant Cell 17 (1): 282-294.
43 Kopriva, S., Mugford, S.G., Matthewman, C., Koprivova, A. 2009. Plant sulfate assimilation genes: redundancy versus specialization. Plant Cell Reports 28 (12): 1769-1780.
44 Landeo, J.A., Gastelo, M., Beltran, G., Diaz, L. 2000. Quantifying genetic variance for horizontal resistance to late blight in potato breeding population B3C1. CIP Program report 1999-2000, International Potato Centre, Peru. pp 63-68.
45 Lawton, M.A., Lamb, C.J. 1987. Transcriptional activation of plant defense genes by fungal elicitor, wounding and infection. Molecular and Cellular Biology 7: 335-341.
46 Leonards-Schippers, C., Gieffers, W., Schäfer-Pregl, R., Ritter, E., Knapp, S.J., Salamini, F., Gebhardt, C., 1994. Quantitative resistance to Phytophthora infestans in potato: a case study for QTL mapping in an allogamous plant species. Genetics 137: 67-77.
47 Li, Y., Tian, Z., Liu, J., Xie, C. 2009. Comparative cDNA-AFLP analysis reveals thatdl--amino-butyric acid induces resistance through early activation of the host-defense genes in potato. Physiologia Plantarum 136: 19-29.
48 Liang, P., Meade, J.D., Pardee, A.B. 2006. Differential display methods and protocols. En Methods in molecular biology. 2nd ed. Towota: NJ Humana Press.
49 Liang, P. 2002. SAGE Genie: a suite with panoramic view of gene expression. Proceedings of the National Academy of Sciences of the United States of America 99: 11547-11548.
50 Lindqvist-kreuze, H., Carbajulca, D., Gonzalez-escobedo, G. 2010. Comparison of transcript profiles in late blight-challenged Solanum cajamarquense and B3C1 potato clones. Molecular Plant Pathology 11: 513-530.
51 López, A., Chaparro, A. 2007. Propuesta de un sistema de transformación de plantas de papa (Solanum tuberosum sp. andigena var. Pastusa suprema) mediado por Agrobacterium tumefaciens." Agronomía Colombiana25: 16-25.
52 López-Martín, M. C., Romero, L. C., Gotor, C. 2008. Cytosolic cysteine in redox signaling. Plant Physiology 3 (10): 880-881.
53 Malcolmson, J.F., Black, W. 1966. New R genes in Solanum demissum lindl. and their complementary races ofPhytophthora infestans (Mont.) de Bary. Euphytica 15: 199-203.
54 Maldonado, L.Y., Delgado, M. C., García,C. Estudio de la estructura genetica de las poblaciones dePhytophthora infestans en las regiones productoras de papa en Colombia [sitio web]. [fecha de acceso: Febrero de 2010]. Disponible en: http://www.redepapa.org/infestans3.pdf.
55 Martinez, E.P., Osorio, J.A. 2007. Estudios preliminares para la producción de un biosurfactante bacteriano activo contra Phytophthora infestans (Mont.) De Bary. Revista Corpoica - Ciencia y Tecnología Agropecuaria 8: 5-16.
56 Martini, N., Egen, M., Rüntz, I., Strittmatter, G. 1993. Promoter sequences of a potato pathogenesis-related gene mediate transcriptional activation selectively upon fungal infection. Molecular & general genetics 236: 179-186.
57 McDowell, J.M., Woffenden, B.J. 2003. Plant disease resistance genes: recent insights and potential applications. Trends in Biotechnology 21: 178-183.
58 Mosquera, T., Fenández, C., Martínez, L., Acuña, A.,Cuéllar, D. 2008. Genética de la resistencia de la papa (Solanum tuberosum) a patógenos. Estado de arte. Agronomia Colombiana 26: 7-15.
59 Müller, K.O., Black, B. 1952. Potato breeding for resistance to late blight and virus disease during the last hundred years. Zeitschrift fur Pflanzenzuchtung 31: 225-236.
60 Nürnberger, T., Lipka, V. 2005. Non-host resistance in plants: new insights into an old phenomenon. Molecular Plant Pathology 6: 335-345.
61 Ojito-Ramos, K. y Portal, O. 2010. Introducción al sistema inmune de las plantas. Biotecnología Vegetal 10 (1): 3-19.
62 Patino, W.D., Mian, O., Hwang, P.M. 2002. Serial Analysis of Gene Expression: Technical Considerations and Applications to Cardiovascular Biology. Circulation Research 9: 565-569.
63 Pinzón, A.,Barreto, E., Bernal, A., Achenie, L., González-Barrios, A.F., Isea, R., Restrepo, S. 2009. Computational models in plant-pathogen interactions: the case of Phytophthora infestans. Theoretical biology & medical modelling 6: 24
64 Restrepo, S., Myers, K.L, Pozo, O., Martin, G.B., Hart, A.L., Buell, C.R., Fry, W.E., Smart, C.D. 2005. Gene profiling of a compatible interaction between Phytophthora infestans and Solanum tuberosum suggests a role for carbonic anhydrase. Molecular plant-microbe interactions 18: 913-922.
65 Riemenschneider, A., Riedel, K., Hoefgen, R., Papenbrock, J., Hesse, H. 2005. Impact of reduced O-Acetylserine ( thiol ) lyase isoform contents on potato plant metabolism. Plant Physiology 137 (3): 892-900.
66 Rodriguez, E. 2000. Efecto de la combinación de un fungicida protectante y la resistencia poligénica aPhytophthora infestans en el manejo de la gota en papa. Universidad Nacional de Colombia, Bogotá, Colombia.
67 Ros, B., Mohler, V., Wenzel, G., Thümmler, F. 2008. Phytophthora infestans-triggered response of growth- and defense-related genes in potato cultivars with different levels of resistance under the influence of nitrogen availability. Physiologia Plantarum 133: 386-396.
68 Ros, B., Thümmler, F., Wenzel, G. 2005. Comparative analysis of Phytophthora infestans induced gene expression in potato cultivars with different levels of resistance. Plant biology 7: 686-693.
69 Sambrook, J., Fritsch, E., Maniatis, T. 1989. Molecular cloning, a laboratory manual. 2th Edtion, United States, Cold Spring Harbor Laboratory Press, pp. 6.46-6.48.
70 Samen, F., Abu-El, M., Secor, G.A., Gudmestad, N.C. 2003. Variability in Virulence Among Asexual Progenies ofPhytophthora infestans. Phytopathology 93: 293-304.
71 Schick, R.E., Schick, E., Haussdorfer, M. 1958. Ein Beitrag zur physiologischen spezialisierun von Phytophthora infestans. Phytopathologische Zeitschrift 31: 225-236.
72 Stein, J., Liang, P. 2002. Differential display technology: a general guide. Cellular and Molecular Life sciences59: 1235- 1240.
73 Stuiver, M.H., Custers, J.H. 2001. Engineering disease resistance in plants. Nature 411: 865-868.
74 Torres, H. 2002. Manual de las enfermedades más importantes de la papa en el Perú [sitio web]. Centro Internacional de la Papa. Lima, Perú [fecha de acceso: Febrero 2010]. Disponible en:http://www.cipotato.org/csd/materials/htorres/HTorresTT.pdf
75 Trognitz, F., Manosalva, P., Gysin, R., Niñio-Liu, D., Simon, R., Herrera, M.R., Trognitz, B., Ghislain, M., Nelson, R. 2002. Plant defense genes associated with quantitative resistance to potato late blight in Solanum phureja x dihaploid S. tuberosum hybrids. Molecular plant-microbe interactions (6): 587-597.
76 Troxler, R.F., Zhang, F., Hu, J., Bogorad, L. 1994. Evidence that sigma factors are components of chloroplast RNA polymerase. Plant Physiology 104 (2): 753-759.
77 Turkensteen, L.J. 1993. Durable resistance of potatoes against Phytophthora infestans. En T Jacobs and JE Parlevliet (eds.). Durability of Disease Resistance. Dordrecht: The Netherlands: Kluwer Academic Publishers. pp. 115-124.
78 Velculescu, V. E., Zhang, L., Vogelstein, B., Kinzler, KW. 1995. Serial analysis of gene expression. Science 270: 484-487.
79 Villareal, H.J., Porras, P.D., Santa, A., Lagoeyte, J., Muñoz, D. 2007. Costos de producción de papa en las principales zonas productoras de colombia. Federación Colombiana de Productores de Papa. Bogotá.
80 Vogel, J.O., Raab, T.K., Schiff, C., Somerville, S.C. 2006. PMR6, a Pectate lyase-like gene required for powdery mildew susceptibility in Arabidopsis. Plant cell 14: 2095-2106.
81 Wang, X., El Hadrami, A., Adam, L.R., Daayf, F. 2008. Differential activation and suppression of potato defence responses by Phytophthora infestans isolates representing US-1 and US-8 genotypes. Plant Pathology57: 1026-1037.
82 Wastie, R.L. 1991. Breeding for resistance. En: DS Ingram and PH Williams (eds.). Phytophthora infestans, the cause of late blight of potato. Advances in Plant Pathology. London: Academic Press. pp.193-224.
83 Wirtz, M., Hell, R. 2006. Functional analysis of the cysteine synthase protein complex from plants: structural, biochemical and regulatory properties. Journal of Plant Physiology 163 (3): 273-286.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2012 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).