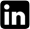LA DUALIDAD ANALOGO DIGITAL DE LA INFORMACIÓN SE EJEMPLIFICA EN EL ESTUDIO DE LAS MOLECULAS DE RNA.
LA DUALIDAD ANÁLOGO DIGITAL DE LA INFORMACIÓN SE EJEMPLIFICA EN EL ESTUDIO DE LAS MÓLECULAS DE RNA
The Analog Digital Duality of Information is Exemplified by Studying RNA Molecules
EUGENIO ANDRADE1. 1Profesor Titular, Departamento de Biología, Facultad de Ciencias, Universidad Nacional de Colombia, Sede Bogotá. leandradep@unal.edu.co
Presentado 4 de abril de 2011, aceptado 30 de mayo de 2011, correcciones 1 de julio de 2011.
RESUMEN
Las investigaciones sobre las moléculas de RNA, han contribuido a precisar y formalizar la noción física de información considerada tanto en su aspecto analógico como digital. El dogma central de la biología molecular, restringe la noción de información al aspecto digital de la misma, y por tanto al copiado fiel de una cadena de símbolos en los procesos de replicación (DNA-DNA) y transcripción (DNA-RNA), e igualmente en la traducción a proteínas mediante el código genético. Esta visión genera la paradoja sobre qué surgió primero si DNA o proteínas, la cual se resuelve en parte recurriendo a la hipótesis según la cual existió un mundo prebiótico RNA, anterior a las proteínas codificadas genéticamente y al DNA. Esta hipótesis tiene soporte empírico sólido apoyado por los descubrimientos de la actividad catalítica de los RNA y el descubrimiento de estructuras RNA altamente conservadas en todos los seres vivos. Por otra parte, los modelos inspirados en la replicación de virus RNA y modelos computacionales que muestran la relación entre estructuras secundarias y secuencias lineales confirman las ventajas de la molécula RNA como modelo para el estudio del origen de la información genética, especialmente por su rol determinante en la aparición del código genético, el papel funcional en la expresión del mismo, la plasticidad estructural y la accesibilidad a las estructuras funcionales a partir de prácticamente cualquier cadena que se someta a ciclos de mutación y selección. Al proponer que la información digital requiere de la analógica para su aparición, no solamente se está contribuyendo a resolver problemas específicos de biología, sino que se trata de una propuesta que contribuye al entendimiento del sentido físico de dos medidas diferentes de información, la de Claude Shannon y la de Gregory Chaitin.
Palabras clave: RNA, DNA, Proteínas, información digital, información analógica, dogma central, mundo RNA.
ABSTRACT
The study of RNA molecules have contributed to formalizing the physical notion of information considered in its dual nature analog and digital. The central dogma of molecular biology restricts the notion of information to its digital aspect and so to copying fidelity of symbol’s string during replication (DNA-DNA) and transcription (DNA-RNA), and also in translation to proteins, by means of a genetic code. This vision leads to the paradox about what molecule came first, DNA or proteins, that is solved by the hypotheses that postulates the existence of prebiotic RNA prior to the emergence of genetically encoded proteins and DNA. This hypothesis is based on solid empirical findings such as the discovery of catalytic RNA and RNA structures highly conserved in evolution and found in all living organisms. Besides, experimental models inspired on virus RNA replication and computational models that deal with the relations between secondary planar structures and linear sequences, confirm the advantages of RNA molecules to understand the origin of genetic information, due to their decisive role in the emergence of the genetic code, their functional role in gene expression, their structural plasticity and the accessibility of functional structures from any arbitrary sequence that undergoes cycles of mutation and selection. To state that the emergence of digital information requires the prior existence of analog information, not only contributes to solve specific problems of biology, but mainly contributes to advance in the understanding the physical meaning of the two different information measurements proposed by Claude Shannon and Gregory Chaitin.
Key words: RNA, DNA, Protein, digital information, analog information, central dogma, RNA world.
EL CONCEPTO DE INFORMACIÓN
-La materia viva, aunque no escapa a las leyes de la física tal como las conocemos, es susceptible de poner en juego otras leyes de la física: una vez sean conocidas, formarán parte de esta ciencia con el mismo título que las precedentes-.
La afirmación precedente tomada del libro -¿Qué es la vida?- de Schrödinger, 1944, describe muy concisamente el ambiente intelectual de la segunda mitad del siglo pasado, en que ante la aparente imposibilidad de explicar mecánicamente a los seres vivos, se esperaba que el estudio de una propiedad característica de la vida, como la reproducción de virus en laboratorio, diera lugar al descubrimiento de nuevas leyes físicas. Sin embargo, la búsqueda de nuevas leyes fue abandonada, puesto que prevaleció el criterio de Monod, 1970, según el cual la vida es algo marginal en el universo y por tanto un caso específico altamente improbable, cuyo estudio no aporta nada al descubrimiento de nuevas leyes físicas. Sin embargo, en la década de los 80, físicos y biólogos coincidieron en aceptar que la teoría de la información era indispensable para entender los seres vivos. El concepto de información a medida que fue formulado y discutido por una diversidad de autores penetró en la biología, hasta convertirse desde 1953 en el paradigma de la biología molecular y genética contemporánea, lo cual ha permitido su reelaboración y formalización como se mencionará más adelante.
Etimológicamente información significa comunicar, colocar o transmitir la forma a algo que no la posee, es decir amorfo. También se utiliza la palabra para referirse a un conocimiento específico, de manera que al estar informados de algo se espera un cierto tipo de comportamiento. La información no reside en una sustancia inmaterial sino que es una consecuencia del proceso de disipación de energía en condiciones de apertura a flujos de energía libre y lejos del estado de equilibrio termodinámico. A medida que se disipa energía no aprovechable al medio externo en forma de entropía, se gene-ran restricciones que favorecen la aparición de sistemas ordenados a nivel puramente local. En estas condiciones los sistemas físicos adoptan patrones organizados estables dentro de ciertos umbrales de estabilidad térmica. La información correspondería a estos procesos físicos que permiten generar estructuras o formas más o menos estables con diferentes grados de probabilidad.
La información como cualquier otra magnitud física es medible. Las medidas de información más utilizadas han sido la propuesta por Claude Shannon, 1948 y posterior-mente la propuesta por Gregory Chaitin, 1969. En el caso de la información de Shannon tenemos que la información se mide en bits. Para una población o conjunto de objetos o mensajes posibles en una fuente emisora, sean cadenas de símbolos o conjuntos de estructuras, se puede estimar el número de bits o preguntas necesarias para identificar una en particular, siempre y cuando la respuesta a la pregunta permita eliminar la mitad de las alternativas posibles. En otras palabras, ante la ignorancia del conjunto de todas las alternativas posibles y estando interesados en identificar una en particular, debemos formular preguntas que respondidas -si- o -no-, permitan reducir con cada una la mitad de las posibles hasta llegar a identificar la deseada. Por tanto el número de preguntas que permite identificarla sin ambigüedad corresponde al número de bits asociado a un mensaje específico; entre más ignorancia poseamos de los estados o mensajes posibles en una fuente, más información necesitamos para identificar un mensaje particular. Por ejemplo, con 3 bits de información encontramos un mensaje entre ocho posibles (8=23), pero si la fuente tuviera 32 (32=25) mensajes posibles, necesitaríamos 5 bits (Fig. 1).
Otra medida conocida como información algorítmica de Chaitin (Chaitin, 1969), intenta estimar la información de un objeto cualquiera una vez que este ha sido digitalizado, es decir codificado como una serie de símbolos. En este caso una cadena o serie de símbolos posee más información entre más largos sean los sectores de la misma que son completamente azarosos, irregulares y por tanto no repetidos. Entre más irregularidades posea un texto, existe mayor probabilidad de que pueda codificar información. Los textos en que un mismo símbolo, silaba o palabra se repiten monótonamente no poseen información así sean muy largos, por el contario los textos aleatorios sí contienen información. Veamos el ejemplo:
(A) 0101010101010101010101, y (B) 0110110011011110001000
Si tenemos dos series de símbolos (A) y (B), dadas en un lenguaje binario y de la misma longitud (en este caso 22 símbolos), tanto A como B contienen la misma información de Shannon, es decir requieren de 22 bits de información para poder ser identificadas cada una por aparte dentro de una población de todas las cadenas binarias de 22 dígitos, es decir entre 222 cadenas posibles. No obstante, por simple inspección podemos ver que la cadena A tiene menos información que la segunda, dado que es una secuencia en que el mismo modulo -0-1- se repite 11 veces, mientras que la B es irregular y no puede ser abreviada o comprimida, contendiendo por tanto más información.
Ambas medidas (la de Shannon y la de Chaitin) se pueden aplicar según el caso, tanto para estructuras, como para cadenas de símbolos. Hecho afortunado si tenemos en cuenta que la información tiene una naturaleza dual analógica y digital. La noción de información analógica se entiende mejor cuando examinamos los ideogramas del chino antiguo y constatamos que en realidad comenzaron como dibujos simplificados del objeto que representan. Se trata de iconos que nos transmiten un significado por su similitud con lo representado. En la época presente, Microsoft utiliza iconos que indican en virtud de su semejanza, la operación propuesta, por ejemplo unas tijeras para cortar un fragmento de un texto en Word. La información analógica tiene la interesante propiedad de que la figura usada como signo, puede soportar un rango continuo de modificaciones, hasta ciertos límites, sin que se altere su significado. Por ejemplo, para comunicar la idea de -hombre-, no existe un único ideograma, dibujo, icono o gráfico que pueda ser usado inequívocamente, puesto que este puede variar dentro de una amplia gama de estilos, tamaños, formas, proporciones y todavía ser entendidos como -hombre-. Es decir la semejanza transmite la idea, esa es la comunicación analógica y su modo de representación puede variar dentro de un rango continuo de formas que no afectan el mensaje. La importancia de la información analógica es interesante si consideramos lo que Astbury, 1938, pensaba en su momento de lo que podría ser la biología molecular. Para este autor la clave de la vida era molecular y se encontraba en las proteínas debido a su complejidad estructural a nivel tridimensional.
-(La biología molecular)... se ocupa particularmente de las formas de las moléculas biológicas, así como de la evolución y de la diversificación de estas formas cuando se pasa a niveles de organización cada vez más elevados. La biología molecular es ante todo tridimensional y estructural, -lo que no significa, sin embargo, que constituya sólo un refinamiento de la morfología. Al mismo tiempo debe estudiar la génesis y la función-. -Conociendo lo que sabemos de los estudios de proteínas fibrosas por rayos X, como están construidas por cadena polipeptídicas con grandes patrones lineales a gran escala, como se contraen y adoptan diferentes configuraciones por plegamiento intramolecular,... es natural asumir como hipótesis de trabajo que forman un largo rollo en el cual está escrito el patrón de la vida.- (Astbury, 1938).
-Estamos en el amanecer de una nueva era, la era de la biología molecular como prefiero llamarla. Es urgente hacer una aplicación más intensa de la física y la química, especialmente del análisis estructural de las proteínas, el cual no es suficientemente apreciado- (Astbury, 1947).
Es decir Astbury imaginó que la clave de la información biológica estaba en las proteínas y era de tipo analógico, mediada por afinidades estructurales que posibilitan los reconocimientos enzima-substrato y/o antígeno-anticuerpo.
Por el contrario la codificación digital es abstracta y recurre a lo simbólico, puesto que no hay conexión directa entre lo que se quiere representar con la palabra utilizada. Por ejemplo, entre la palabra -hombre- y el objeto designado, en este caso el ser humano. En términos más sencillos, decimos que no hay nada en las letras -h-, -o-, -m-, -b-, -r-, -e- que se parezca a lo que representa, del mismo modo en la palabra inglesa -m-, -a-, -n-, las letras -m-, -a- y -n- son arbitrarias, puesto que su correcta traducción exige una contextualización lingüística que por fuerza de la costumbre, a partir de circunstancias contingentes, adoptó esta palabra para transmitir inequívocamente un significado. En los lenguajes digitales, la correspondencia entre la palabra y el significado es convencional, arbitraria, y está influenciada por accidentes dependientes de la historia social y cultural de la comunidad parlante, en consecuencia hubiera podido ser otra. Además modificar la palabra sustituyendo letras por otras del mismo alfabeto en la mayoría de las veces afecta completamente el sentido, por ejemplo en -hambre-, aunque difiere de -hombre- en la sustitución de una -o- por una -a- su significación es muy diferente. Otras sustituciones posibles dan lugar a -hombro-, pero la gran mayoría de los cambios en una letra no tiene significado dentro de la lengua en cuestión, puesto que la significación depende del orden estricto en que están colocados los símbolos. Los lenguajes digitales que mejor conocemos son los lenguajes humanos, pero los lenguajes abstractos binarios que utilizan dos símbolos (simplemente ceros y unos), son suficientes para que por permutación de los mismos en palabras de diferente longitud se den posibilidades de codificación in-mensas, es decir asociar palabras a significados. Otro ejemplo de código digital es el Morse (Schrödinger, 1944), que utiliza puntos, rayas y pausas.
La información digital requiere las siguientes condiciones: 1. Existencia de un alfabeto o conjunto de símbolos claramente definidos. 2. Agrupamiento de los símbolos en cadenas o palabras de longitud variable. 3. Orden irregular y aperiódico de los símbolos y las palabras, no monótono. 4. Una clave o código que permite leerlo y traducirlo dentro de un contexto de interpretación preciso. En consecuencia Schrödinger, 1944, propuso que el orden de los seres vivos se debía al ordenamiento de macromoléculas poliméricas termodinámicamente estables, que conformaban un cristal o estructura aperiódica que permitía guardar toda la información necesaria para codificar su estructura y las instrucciones que guían su desarrollo. El carácter aperiódico se refiere a que los símbolos químicos no deben repetirse monótonamente, sino irregularmente como las letras, sílabas y palabras en un texto escrito. Para Schrödinger, 1944, a nivel de los organismos el orden viviente se fundamenta en el orden y estabilidad de las macromoléculas informativas.
-A menudo se ha preguntado cómo en el núcleo de un óvulo fertilizado, puede estar contenida una clave elaborada y que contiene todo el desarrollo futuro del organismo. Una asociación bien ordenada de átomos, capaz de mantener permanentemente su orden, parece ser la única estructura material concebible que ofrece una variedad de posibles organizaciones (isoméricas) y que es suficientemente grande como para contener un sistema complicado de determinaciones dentro de reducidos límites espaciales. En efecto, el número de átomos de una estructura tal no necesita ser muy grande para producir un número casi ilimitado de posibles combinaciones. Como ejemplo, pensemos en la clave Morse. Los dos signos diferentes del punto y raya, en grupos bien ordenados de no más de cuatro permiten treinta especificaciones diferentes. Ahora bien, si agregamos un tercer signo, usando grupos de no más de diez, podríamos formar 88.572 palabras diferentes; con cinco signos y grupos de hasta 25, el número asciende a 372.529.029.846.191.405- (Schrödinger, 1944, p. 97).
La dualidad entre sistemas de codificación digital y analógica, se constata en el caso de los registros de música que hasta hace pocas décadas se grababa en discos de acetato que poseían un labrado microscópico en los carriles marcados en la superficie del mismo que presentaban un relieve análogo o similar a las ondulaciones de las ondas musicales correspondientes. Esta codificación analógica dio paso a las tecnologías de codificación digital, donde lo único que hay son -ceros y unos-. El modo de registro digital, es mucho más eficiente, fiel, económico y durable, pero su existencia solo se hizo posible cuando el desarrollo tecnológico permitió pasar de los sistemas análogos a los digitales y no al revés.
Erwin Schrödinger, 1944, Leo Brillouin 1962 y Brooks et al., 1988 asocian la información a la estructura u organización de un sistema que lo mantiene ordenado es decir lejos del estado en el que todos los elementos constitutivos estuvieran aleatorizados. En este sentido, la información se ha equiparado a una -entropía negativa- que contrarresta el desorden molecular, más precisamente mantener un orden estructural tiene un costo en términos de energía. Por tanto un organismo mientras mantiene su organización, es decir la forma, conserva la información estructural y genética, pero al disociarse en sus componentes y degradarse químicamente muere y pierde la información estructural, pero si el mensaje contenido en sus genes se ha transmitido a su descendencia, la información genética o digital no muere, se mantiene. Esta aparente contradicción se deriva del carácter dual de la información que reúne dos aspectos el genético y el fenotípico, el mensaje codificado y la estructura. Esta dualidad digitalanalógica (Hoffmeyer et al., 1991) ha hecho que la noción de información se interprete mejor mediante la semiótica o ciencia que investiga la producción, transmisión, codificación, e interpretación de signos y símbolos (Andrade, 2003). Veremos, más adelante que un objeto natural conocido como la molécula de RNA posee información tanto digital -ella misma es una secuencia de símbolos-, como analógica -posee estructura-, constituyéndose en un modelo idóneo para estudiar este problema (Andrade, 2000; Andrade, 2003).
EL DNA COMO REGISTRO DIGITAL DE INFORMACIÓN Y EL CÓDIGO GENÉTICO
Watson y Crack, 1953, propusieron el modelo estructural del DNA, y lo describieron como una doble hélice conformada por cadenas poliméricas de cuatro desoxirribonucleótidos conformados por las bases nitrogenadas adenina (A), citocina (C), guanina (G) y timina (T). El carácter constante de los apareamientos específicos dados por puentes de hidrógeno entre A y T (A=T) y entre C y G (C�G; Chargaff, 1951), seguido de la repetición irregular de los mismos a lo largo de la secuencia, condujo a su identificación con el cristal aperiódico de Schrödinger. Desde entonces el DNA fue reconocido como la molécula informativa de la vida. Actualmente, el predomino del DNA en la investigación biológica, se corresponde curiosamente con la supremacía de los sistemas digitales de información en la tecnología.
La información digital del DNA se refiere a que tratamos con un polímero conformado por cuatro tipos de monómeros identificados con las letras A, C, G, T, que puede alcanzar extensiones de algunos millones de pares de bases en bacterias hasta miles de millones en humanos. En esta cadena los monómeros funcionan como símbolos lingüísticos y aparecen formando un texto que al leerse linealmente muestran una secuencia irregular o aperiódica, con patrones estadísticos similares a los que corresponden a las frecuencias de aparición de letras, sílabas y palabras en los lenguajes articulados humanos. En los lenguajes naturales las palabras más cortas aparecen con más frecuencia que las más largas, de la misma manera en el DNA las palabras más cortas como duplas AA, AC, AG, AT,... aparecen con más frecuencia que tripletas, y así sucesivamente (Mantegna et al., 1994). Pero al igual que en los lenguajes, las sílabas de la misma longitud en las macromoléculas biológicas no aparecen con la misma frecuencia, hay muchas que aparecen con muy poca frecuencia y pocas que aparecen con muy alta frecuencia. En este caso, hablamos de información como el mensaje codificado a la manera de un texto que informa, en cuanto contiene el programa o las instrucciones necesarias para dirigir la morfogénesis del ser vivo, a la vez que es responsable de la transmisión hereditaria de la forma de padres a hijos.
Crick (Crick, 1955; Crick, 1958) utilizó el término información, en cuanto información digital únicamente, es decir la almacenada en modo encriptado o la codificada en función del orden de los símbolos y en consecuencia restringió el fenómeno de la transferencia de información al copiado de la molécula. Entre más fiel sea el copiado más se conserva la información. Por esta razón en biología molecular cuando se habla de información se debe entender como información digital, y su manifestación más representativa es la información genética almacenada en el DNA, la molécula depositaria de la misma por excelencia. De esta manera el concepto de información se restringe a la fidelidad en el copiado de la secuencia de símbolos, es decir a la preservación del orden de monómeros en el copiado de la molécula (replicación DNA-DNA, transcripción DNA-RNA) y traducción (RNA-proteína) o síntesis proteica de acuerdo a las correspondencias entre la secuencia de nucleótidos en el DNA y RNA mensajero con la secuencia de los aminoácidos en la proteína, en conformidad con lo establecido en el código genético. Por tanto, alterar levemente el orden por sustitución de una letra por otra se constituye en una mutación que en la mayoría de los casos es neutra o no afecta la información existente, y en menor medida la puede afectar gravemente. Sin embargo, en una proporción muy baja estas mutaciones pueden conferir cambios ventajosos que son retenidos evolutivamente por la selección natural y en los casos de duplicación genética la acumulación de mutaciones puede convertirse en una innovación evolutiva (Kimura, 1979; Kimura, 1983).
EL CÓDIGO GENÉTICO
La tabla de correspondencias entre el alfabeto de DNA con cuatro símbolos (nucleótidos) y él de proteínas con 20 (aminoácidos) se denomina código genético (Fig. 2). El código muestra que mediante una asignación en tripletas (64 posibles), de modo redundante, pero sin ambigüedad es posible asignar un código para cada uno de los 20 aminoácidos. Es un código sin comas, no superpuesto.

El desciframiento del código genético iniciado por Gamow, 1954, y posteriormente resuelto por Khorana, 1965 y Niremberg, 1963 entre otros, hizo creer en su momento que se había desentrañado la base fundamental de la vida, la cual radicaba en últimas en la información contendida en el DNA. Dilucidar este código fue una de las mayores hazañas de la ciencia en el siglo XX, y mostró que la vida en la tierra tiene un lenguaje único simbólico, puesto que el mismo código está presente tanto en las bacterias más simples como en las formas de vida más complejas e inteligentes como mamíferos. La universalidad del código se constituyó en el argumento central para defender el origen único de la vida terrestre y hoy en día nos preguntamos sobre si otras vidas microbianas que pudieran ser detectadas en otros cuerpos celestes compartirían el mismo código o no. Existen algunas excepciones mínimas que fueron identificadas en mitocondrias de mamíferos (Fearnley y Walker, 1987).
La alta redundancia del código ha sido interpretada como el resultado de un proceso evolutivo tendiente a la optimización y salvaguarda contra errores en la replicación y en la traducción (Haig y Hurst, 1991).
En la mayoría de los casos la asignación de aminoácidos la definen las dos primeras posiciones del codón, mientras que en la mayoría de los casos el cambio en la tercera posición es sinónimo. El código genético en su estructura muestra como los nucleótidos en la segunda posición de los codones distinguen según sea Y (pirimidinas C o T) o R (purinas A o G) entre amino ácidos hidrofóbicos e hidrofílicos respectivamente (Fitch, 1966). Además el código genético que posibilita la traducción libre de ambigüedad del lenguaje del DNA al de las proteínas, requiere de reconocimientos estructurales específicos (información analógica) entre aminoácidos y RNA de transferencia (t-RNA) y entre aminoacil-RNA-sintetasas (aa-rnasa) y t-RNA y entre aquellas y aminoácidos. Reconocimientos químicos altamente específicos permitieron crear un código digital que por su eficiencia en la replicación y alta capacidad de almacenar información se convirtieron en la pieza fundamental de la programación genética de los organismos.
EL DOGMA CENTRAL
El análisis de relaciones entre DNA, RNA y proteínas, condujo a Crick (Crick, 1958; Crick, 1970) a concluir que de todas las trasferencias de información, por copiado del polímero, teóricamente pensables solo son factibles dado el conocimiento de la maquinaria enzimática de las células, las que conocemos como: 1) replicación o copiado de una molécula de DNA para generar la cadena de DNA complementaria, 2) transcripción o paso del DNA a una copia en forma de RNA, 3) traducción o paso de la secuencia de RNA a la secuencia de aminoácidos propia de una proteína, 4) transcripción reversa o paso de RNA a una copia en la forma de DNA, 5) replicación o paso de un RNA a la producción de una copia complementaria de RNA. Crick propuso como transferencia posible aunque poco frecuente o rara, la traducción directa de DNA a proteína, fenómeno que hasta el presente no ha sido reportado, pero resaltó con vehemencia la imposibilidad de todos los flujos de información que parten de la proteína. Es decir que es imposible la replicación proteica o el copiado de una proteína para obtener más proteínas iguales o lo que es lo mismo, es imposible que una proteína guíe la copia de ella misma produciendo otra idéntica. Igualmente, la traducción reversa o paso de proteína a RNA (o a DNA), están prohibidas, es decir que es imposible que una proteína, de acuerdo al orden de sus aminoácidos, logre fabricar el RNA o el DNA que la codifica. En este sentido según Crick (Crick, 1958) podemos afirmar que el DNA ubicado en el núcleo de una célula, es la fuente de la información, la cual se transmite al citoplasma por medio de una molécula mensajera de RNA, la cual se utiliza para ser traducida dando lugar a proteínas. Estas últimas equivalen al mensaje recibido. Este flujo de información es unidireccional e irreversible. Este flujo irreversible de información de DNA a proteína se denominó -dogma central- de la biología molecular (Fig. 3), según el cual los flujos reversos que parten de proteínas están prohibidos. En 1958 Francis Crick afirmó:
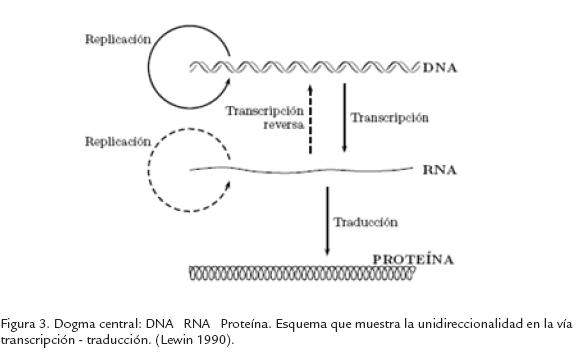
-el DNA dirige su propia replicación y transcripción a RNA, el cual dirige su traducción a proteínas. La información proteica no regresa al DNA-.
Esta formulación tuvo profundas repercusiones en la investigación biológica, puesto que reorientó toda la biología hacia el desentrañamiento del código genético que es el que permite pasar de los lenguajes codificados en los alfabetos isomorfos con cuatro letras del DNA y el RNA al lenguaje de las proteínas constituido por 20 letras o aminoácidos. El dogma central de la biología utiliza el concepto de información en el sentido de información digital arriba mencionado, pero por claridad a lo largo de este texto mantendré la distinción entre la información digital e información analógica o estructural. Además, la distinción análogo-digital es crucial si queremos entender los fundamentos fisicoquímicos de la información genética, puesto que la primera hace posible la aparición de la segunda. La actividad biológica depende de enzimas cuya estructura tridimensional les permite reconocer sus sustratos y por tanto actuar en consecuencia. La funcionalidad y el mantenimiento de la actividad metabólica requieren de enzimas que oscilan entre configuraciones estructurales muy precisas. La importancia de los reconocimientos específicos entre aminoácidos, enzimas aminoacil-RNA-sintetasas y los respectivos RNA de transferencia pone de relieve la importancia de la información analógica que posibilita la existencia y el funcionamiento de la digital.
Por otra parte, sostener que el DNA se transmite de generación en generación sin ser afectado por las proteínas, constituye una reelaboración de la idea acerca de la separación entre fenotipo (células somáticas) y genotipo (células germinales) denominada -barrera de Weismman-, formulada en 1902 (Buss, 1987), según la cual existe una continuidad e inmortalidad de las células germinales las cuales no se afectan por las modificaciones sufridas por las células somáticas. Esta idea devino en la base de la genética moderna de principios del siglo XX que prohibía cambios en el genotipo a consecuencia de modificaciones en el fenotipo inducidas por el medio ambiente.
En su momento, la formulación del dogma central, consolidó la separación conceptual entre genotipo y fenotipo, sobre la cual se había construido la genética, argumentando que el DNA varía al azar y por tanto no sufre alteraciones dirigidas o influenciadas a responder de un modo específico a factores del medio ambiente. El programa genético contendría una serie completa de instrucciones pero estaría cerrado a las influencias provenientes del medio ambiente, posición que reñía con la posición neo-lamarckiana que aceptaba que el medio ambiente puede incidir directamente en la modificación y transformación evolutiva. El rol asignado al medio ambiente, quedó restringido al de factor selectivo que como resultado de la escasez de recursos disponibles para una población creciente, retiene a las formas más adaptadas y elimina las menos.
QUÉ FUE PRIMERO, ¿DNA O PROTEÍNAS?
A partir de la formulación del dogma central hizo carrera una visión centrada en el DNA para explicar la evolución biológica mediante el mecanismo de selección natural de mutaciones genéticas aleatorias. Una posición extrema de este punto de vista se presenta en -el gen egoísta- de Dawkins, 1976, donde sostiene que los fenotipos son vehículos sofisticados generados por los genes para dispersarse y diseminarse de la manera más eficiente. En general las investigaciones biológicas se reorientaron hacia la búsqueda de secuencias génicas para medir distancias filogenéticas y reorganizar la clasificación taxonómica por ejemplo. Esta óptica, denominada geno-centrista que privilegia la información digital, encausó las investigaciones sobre el origen de la vida hacia la búsqueda de genes o moléculas informativas autorreplicantes, hecho que contradecía la visión de Oparin (Oparin, 1952 [1938]; Oparin, 1991 [1950]), y en general de los bioquímicos para quienes las enzimas (proteínas) eran las moléculas responsables del metabolismo o la actividad vital propiamente tal, las verdaderas protagonistas de la vida.
-todo organismo, ya sea animal, planta o microbio, vive solamente mientras estén pasando por él, en torrente continuo, nuevas y nuevas partículas de substancias, impregnadas de energía-, (Oparin, 1991 [1950]).
El problema sobre cómo surgió la información genética, no fue abordado sino a principios de la década de los 70 (Eigen, 1971; Eigen, 1992). Hasta entonces se pensaba que este problema remitía a una paradoja aparentemente insoluble. ¿Qué surgió primero, DNA o proteínas? El carácter paradójico de esta cuestión, resulta de considerar que la replicación de DNA requiere de la participación de un número muy grande de enzimas proteicas, y que la fabricación de una proteína requiere de la preexistencia de los genes o secuencias de DNA que las codifican. Si las proteínas surgieron primero, entonces ¿cómo se codificaba su información? Si por el contrario el DNA surgió primero, entonces, ¿cómo se efectuaba su replicación? Es decir las proteínas requieren DNA y éste de proteínas, dejándonos atrapados sin salida en una argumentación circular.
Defender la idea de que lo primero en surgir fueron las proteínas, constituía una toma de partido por una definición de lo vivo como sistema metabólico de automantenimiento y renovación permanente. Para los defensores del metabolismo primero, la capacidad de reproducirse o dividirse, nunca fue, ni ha sido el elemento definitorio de la vida. A principios del siglo XX la vida se definía por las actividades enzimáticas y catalíticas, es decir por el metabolismo o transformación química permanente. La vida se concebía como una cocina química donde se sinterizaban moléculas orgánicas de alto peso molecular, a partir de moléculas tomadas de medio, a la vez que se degradan moléculas de alto peso molecular para producir compuestos de excreción liberados al medio, mediante pasos ejecutados por enzimas que facilitan las transacciones energéticas requeridas. (Oparin, 1952 [1938]) había postulado que la atmósfera primitiva de la tierra estaba compuesta por nitrógeno, hidrógeno, metano, amoníaco, gas carbónico y vapor de agua. Debido a la acción de los rayos solares, esta mezcla gaseosa, habría dado lugar a gran cantidad y diversidad de moléculas orgánicas, que cayeron con la lluvia y se acumularon en los océanos durante largos períodos de tiempo, formando un -caldo nutritivo primitivo-. Las moléculas se asociaron entre sí, formando agregados moleculares cada vez más complejos, que denominó coacervados. Los coacervados tenían la capacidad de autosíntesis, es decir de aprovechar materia orgánica presente en la denominada -sopa prebiótica-, evolucionando hacia formas cada vez más estables y complicadas hasta convertirse en los organismos primigenios a partir de los cuales evolucionó la vida en la tierra (Lazcano, 2007).
Los coacervados se generaron espontáneamente cuando compuestos de alto peso molecular como proteínas y carbohidratos (resinas vegetales, gomas y almidones) que al suspenderse en agua, manifiestan una tendencia espontánea a asociarse formando glóbulos más o menos estables o estructuras diferenciadas en un medio acuoso.
Cuando estos glóbulos atrapan enzimas, aisladas químicamente en el laboratorio, se pueden convertir en la sede de una serie de procesos físicos y de reacciones químicas de relativa complejidad. Por ejemplo, si las enzimas atrapadas al interior del coacervado por un lado incorporan monómeros de glucosa al carbohidrato y por el otro la degradan produciendo maltosa, se genera un coacervado estable que sintetiza y degrada el almidón constituyente del mismo. Estos glóbulos adquieren estabilidad debido a que albergan una reacción metabólica tan simple y en consecuencia, ¿por qué no imaginarlos, como posibles modelos de la vida primigenia? La reproducción sería no un aspecto definitorio de la vida sino una consecuencia de una característica más notable, el metabolismo, es decir el flujo de energía procedente del sol que hizo posible la síntesis de materia orgánica en la tierra.
Por otra parte el descubrimiento de virus o pequeñas partículas responsables de la propagación de enfermedades infecciosas en plantas, que no eran retenidas por filtros de mallas ultramicroscópicas llevó a considerar que la característica más notoria de la vida sería esa capacidad de propagación asombrosa derivada de la reproducción. Además debido a la influencia del darwinismo, la vida se concebía como sistemas que tienden a reproducirse más allá de los recursos disponibles y por tanto era imperativa la acción de la selección natural como restauradora del equilibrio entre organismos y recursos disponibles.
Pero pronto la búsqueda de esta hipotética molécula de DNA primitivo autorreplicante se dejó de lado considerando que hay argumentos a favor de que el DNA fue más bien una molécula que apareció tardíamente en la evolución prebiótica. Los argumentos que dan fuerza a este planteamiento son los siguientes: 1. El DNA tiene una estructura químicamente muy estable, la cual proviene del hecho de estar conformada por desoxirribonucleótidos que son químicamente poco reactivos debido a que carecen del grupo 2’OH en la ribosa. 2. El DNA requiere de RNA para existir puesto que los monómeros de RNA (ribonucleótidos) son necesarios para producir los de DNA (desoxirribonucleótidos) y no al contrario. 3. En la replicación de DNA participan más de 20 enzimas proteicas distintas, y se requiere de un RNA iniciador de la polimerización, es decir que el copiado de DNA se hace adicionando nucleótidos a cadenas cortas de RNA. 4. El DNA son moléculas químicamente muy estables, debido a que las dobles cadenas absorben menos radiación ultravioleta que las de RNA, y por lo tanto sufren menos mutaciones que puedan alterar su secuencia, preservando el registro digital informativo de mejor manera (Lazcano et al., 1996; Lazcano, 2007).
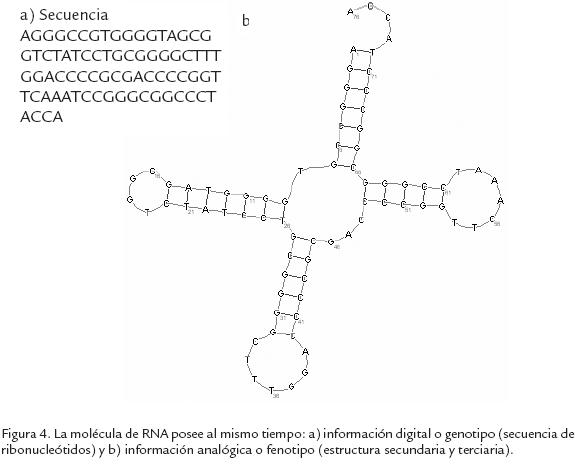
Resumiendo, el DNA es un texto lineal que puede ser copiado fielmente y se presenta en la forma de dos cadenas complementarias doble hélice. El RNA es un texto lineal copiado (transcrito) de DNA que además, se pliega sobre sí mismo, dando lugar a una estructura tridimensional. Igualmente, las proteínas son una secuencia lineal de amino ácidos traducida a partir de RNA mensajero y además se pliegan dando lugar a una estructura tridimensional (Fig. 4). En conclusión las tres macromoléculas poseen información digital y dos de ellas RNA y proteínas poseen además información estructural o analógica, aunque hoy en día se acepta que la topología sinuosa de las caras externas de la doble hélice de DNA son sede de reconocimientos específicos por proteínas y factores de transcripción lo cual indicaría que el DNA también poseería información analógica, aunque obviamente su papel principal es el de servir de depósito químicamente estable de la información digital acumulada a lo largo de la evolución. Ante la imposibilidad de decidir entre DNA y proteínas, ¿cual apareció primero?, la atención se centró en las moléculas de RNA, justamente por su carácter dual analógico y digital.
MUNDO RNA
La resolución de la paradoja o DNA o proteínas, requeriría poder justificar un proceso co-evolutivo basado en otro tipo de moléculas que como el RNA tuvieran al mismo tiempo la capacidad de comportarse como enzima y de poseer una secuencia de símbolos completamente homóloga al DNA.
Las moléculas de RNA en cuanto se producen por transcripción o copiado de una de las cadenas de DNA mantienen la información de secuencia mediante apareamientos específicos de los nucleótidos en este caso entre A y U (A=U) y entre C y G (C�G). Además de poseer genotipo (información digital), poseen fenotipo (información analógica), el cual se refiere a las estructuras secundarias de los RNA en las que observamos tallos o segmentos de doble cadena apareada y bucles sin aparear, generados mediante los apareamientos de sitios complementarios ubicados dentro de la misma molécula. Además, las estructuras planares se pliegan sobre ellas mismas dando lugar a estructuras terciarias o tridimensionales que permiten, en consecuencia, el establecimiento de interacciones específicas con otras moléculas, y por tanto de los reconocimientos enzima-sustrato necesarios para la actividad catalítica (Cech et al., 1980; Altman et al., 1983; Guerrier-Takada et al.; 1983, Zaug et al., 1986).
Crick, 1955, en contra de Gamow, 1954, planteó que no hay interacciones químicas directas entre las tripletas de DNA y los aminoácidos respectivos, y por tanto postuló la existencia de moléculas de RNA adaptadoras que por un lado interactúan químicamente con el RNA mensajero y por otra parte con los aminoácidos.
-The main idea was that it was very difficult to consider how DNA or RNA, in any conceivable form, could provide a direct template for the side-chains of the twenty standard amino acids. What any structure was likely to have was a specific pattern of atomic groups that could form hydrogen bonds. I therefore proposed a theory in which there were twenty adaptors (one for each amino acid), together with twenty special enzymes. Each enzyme would join one particular amino acid to its own special adaptor. This combination would then diffuse to the RNA template. An adaptor molecule could fit in only those places on the nucleic acid template where it could form the necessary hydrogen bonds to hold it in place. Sitting there, it would have carried its amino acid to just the right place where it was needed.- (Crick, 1955).
Esta molécula adaptadora se conoce hoy en día como RNA de transferencia (Hoagland et al., 1958) y es la molécula que explica las correspondencias establecidas en el código genético. Se trata de una molécula de aproximadamente 75 nucleótidos que se pliega dando una estructura característica denominada -hoja de trébol- y una estructura terciaria en forma de -L-, en la cual se destaca una región anticodónica no apareada que posee una tripleta complementaria al codón del RNA mensajero y una región apareada denominada tallo aceptor que es reconocida por el amino ácido correspondiente, el cual se une químicamente al extremo terminal CCA3’. Este último reconocimiento está mediado por una enzima denominada aminoacil-RNA-sintetasa. Los estudios sobre cómo pudo haberse generado en la evolución prebiótica el código genético se centran en el estudio de las moléculas de RNA de transferencia. Posteriormente, Cech, 1980, descubrió la actividad catalítica del RNA al estudiar el procesamiento de RNA ribosomal extraído de un protozoario ciliado conocido como Tetrahymena termophyla (Cech, 1980; Cech et al., 1982). Cech, 1980, mostró que tenía lugar la autoeliminación del intrón, mediante un proceso que requería como enzima al mismo RNA en cuestión, iones magnesio y un cofactor de guanosina. Esta última actúa como donador de electrones que desestabiliza mediante ataque nucleofílico el enlace fosfodiester, el cual se rompe por acción del mismo RNA. Al tratarse de moléculas cuya serie o secuencia de letras corresponde a la del DNA y que al mismo tiempo presentan estructura terciaria que las habilita para cumplir tareas enzimáticas, Gilbert, 1986, planteó que la naturaleza del RNA moderno, sugiere un mundo RNA prebiótico. Desde entonces la atención se ha centrado en el estudio del denominado -mundo RNA-. Posteriormente se han identificado moléculas de RNA pequeñas de aproximadamente 52 nucleótidos, ribozimas cabeza de martillo, gancho de pelo y otras que poseen actividad enzimática y actúan rompiendo enlaces fosfodiester en sitios específicos (Doudna et al., 2002). Además ha sido posible obtener gran diversidad de ribozimas in vitro mediante procesos de mutación y selección en un número razonable de ciclos (Bartel y Szostak, 1993).
El mundo RNA designa una etapa anterior al origen de la vida, datado en 3.500 millones de años, en la cual se definió el código genético universal, y en la cual predominaban moléculas de RNA que eran las ejecutoras de las tareas catalíticas primordiales y al mismo tiempo guardaban los registros de información digital nacientes que se comenzaban a configurar. Uno de los argumentos más fuertes a favor de esta hipótesis radica en concebir los RNA de todos los seres vivos actuales como un relicto de alguna etapa anterior, dado que en todos ellos el RNA es necesario para la síntesis de proteínas (RNA mensajero, RNA transferencia y RNA ribosomal), tratándose de funciones cruciales para la expresión genética en todos los organismos, debieron ser muy antiguas y difíciles de reemplazar. Por tanto las funciones del RNA que tienen que ver con replicación, transcripción y traducción se establecieron desde antes del último ancestro común (Pool et al., 1997).
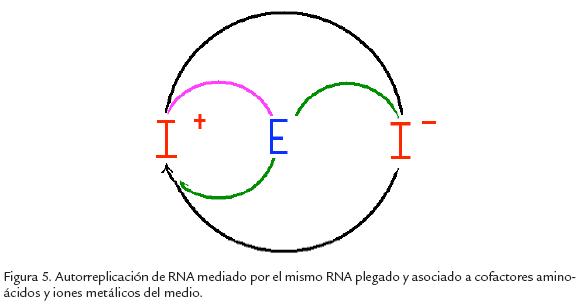
Asombrosamente, hoy en día se considera que el estudio de los RNA también nos acerca al entendimiento de los complejos procesos de regulación de la expresión genética dada la alta cantidad y diversidad de RNA funcionales de tamaño corto que se observan al interior de los núcleos de células eucarióticas. Paradójicamente con el estudio de los RNA virales se pudo entender que nunca ha existido algo así como un -gen desnudo autorreplicante- sino que en su lugar tenemos ciclos de autorreplicación de moléculas cortas de RNA (Fig. 5) mediados por ellas mismas en estado plegado y ayudadas por péptidos muy cortos. Manfred Eigen y su grupo (Eigen et al., 1981) identificaron en una cepa patogénica de Escherichia coli un virus de RNA de cadena sencilla de 4.300 nucleótidos de largo, el cual se conoce como Qβ. Este virus fue adoptado como modelo de sistema mínimo autorreplicante identificado en la naturaleza. El estudio de poblaciones de virus generadas por replicación de los mismos al interior de la bacteria le permitió inferir que a pesar de que en la población aparecen variantes mutacionales del mismo, se puede detectar un patrón constante que identificó por medio de una secuencia maestra propia de la cuasi-especie o población viral. En una serie de experimentos de selección in vitro se pudo observar que si la tasa de mutación sobrepasa cierto umbral de error, se hace imposible identificar la secuencia maestra puesto que el amplio rango de variación conduce a que la población perezca en el medio ambiente específico en que se pone a reproducir. En el caso particular de este bacteriófago se observó que la enzima de replicación está codificada dentro del mismo segmento de RNA viral que copia con cierto error, las cadenas positivas en sus cadenas complementarias. A su vez la misma enzima copiaba las cadenas negativas en su contraparte positiva, generando de esta manera la cuasi-especie es decir dos poblaciones complementarias de cadenas de RNA que aunque variables dentro de un umbral de error bajo, son identificables por una secuencia maestra. Eigen pensó que este modelo simple de replicación viral, bien podría haber sido mucho más simple en la evolución molecular prebiótica donde hubiera sido suficiente con proponer que algo parecido podría haber tenido lugar en caso de que la propia cadena de RNA en cuestión en su estado plegado hubiera actuado como enzima de replicación, obviamente en un medio ambiente propicio rico en los monómeros o ribonucleótidos en estado libre producidos presumiblemente por síntesis química abiótica. Así las cosas, los RNAs autorreplicantes proporcionarían un mecanismo de herencia y evolución por selección natural de moléculas que se autorreplicasen de la manera más eficaz (Gilbert y Souza, 1998; Joyce y Orgel, 1998).
En una población de moléculas que se replican con error, la selección natural lejos de favorecer a una única secuencia adaptada retiene una población dispersa de variantes ligeramente diferentes que se replican debajo del umbral de error pero que todavía alcanza a ser descrita por una secuencia maestra. La adaptabilidad de la población depende de que se mantenga esta diversidad propia de la cuasi-especie. En la década de los años ochenta la hipótesis de cuasi-especies de RNA replicantes constituyó un paso más allá de la contraposición entre DNA y/o proteínas, aunque un mundo RNA no está libre de sus propias paradojas, la cual había sido anunciada por el propio Eigen, 1971, de la siguiente manera. La paradoja de Eigen sostiene que la replicación de RNA al carecer de un mecanismo de corrección de errores, el tamaño máximo de un replicador sería de aproximadamente 100 ribo-nucleótidos, una longitud muy corta para poder explicar por este proceso el origen de la información genética contenida en los genes necesarios para codificar enzimas de replicación altamente específicas que reduzcan efectivamente la tasa de error. En otras palabras sin enzimas correctoras no hay genes de tamaño mayor a 100 ribo-nucleótidos, pero sin genes de ta-maño mayor, no pueden existir enzimas correctoras. Para que un genoma pueda especificar un mecanismo corrector de errores, este debe estar en capacidad para codificar ribozimas de una longitud mucho mayor de 100 ribo-nucleótidos. En esta versión de la paradoja -qué fue primero, si el huevo o la gallina- tenemos que la replicación fiel de RNA, requiere de enzimas evolucionadas y complejas, codificadas en cadenas de RNA suficientemente largas. Es decir la ribozima (la estructura plegada) requiere al gen (la cadena lineal) y éste requiere a la ribozima.
La solución a esta paradoja reside en concebir el origen prebiótico de la vida no sola-mente como una competencia entre minirreplicadores tipo RNA, sino sobre todo como una actividad de cooperación y refuerzo autocatalítico en la que participan un conjunto de minirreplicadores. Este modelo de hiperciclos (Eigen, 1971; Eigen et al., 1981; Eigen, 1992) muestra que a partir de una actividad catalítica primigenia de RNA es posible explicar el origen de la traducción y el código genético (RNA-proteína). De esta manera, Blomberg, 1997, sostuvo que la vida comenzó con conjuntos de RNA autorreplicantes que evolucionaron gradualmente hasta adquirir las funciones necesarias para la vida de la célula (Bartel y Szostak, 1993).
Entre las actividades enzimáticas identificadas in vitro que pueden ser ejecutadas por ribozimas está la actividad replicasa o copiado del propio RNA (Ekland, 1996). Weiner y Maizels, 1987, demostraron que una variante de replicasa de RNA pudo haber funcionado como la primera aminoácil-RNA-sintetasa, puesto que se une a la región terminal 3’ CCA de t-ARN dando lugar a su aminoacilación que constituye un paso decisivo en la síntesis de proteínas. También se ha observado replicación in vitro de fragmentos de 5 a 10 ribo-nucleótidos sobre una platilla molde de RNA y síntesis en presencia de iones Zn+2 de fragmentos hasta 40 ribo-nucleótidos, los cuales se replican con alta tasa de error.
En consecuencia, las moléculas de RNA fueron la base de la vida primitiva y a partir de ellas la evolución se dio en un proceso en el cual podemos identificar al menos cinco etapas. 1) Las moléculas RNA catalítico surgieron por condensación de nucleótidos y fueron copiadas por replicasas conformadas por los mismos RNA plegados (Eigen et al., 1981). 2) A medida que emergían nuevos RNAs, aparecieron diferentes poblaciones con distintas funciones, cada una dependiente de una replicasa de RNA. Mediante replicación con error y recombinaciones, surgieron nuevas moléculas ampliando el rango de actividades enzimáticas. 3) La síntesis de péptidos comenzó con la creación de adaptadores asociados a aminoácidos. Estos adaptadores se reconocen como los precursores de los RNA de transferencia actuales, los cuales favorecieron la producción de las primeras proteínas que fueron seleccionadas por ejecutar las tareas catalíticas de modo más eficiente que las ribozimas (Szathmary, 1993). 4) El DNA surgió, tardíamente, por transcripción reversa y fue seleccionado por constituir un mejor soporte químico para preservar información y corregir errores (Eigen et al., 1981; Freeland et al., 1999; Gilbert y Souza, 1998). 5) El RNA adoptó el rol de intermediario o mensajero de información que hoy tiene, mientras que otras moléculas de RNA mantuvieron su papel catalítico (Pool et al., 1997).
Pero, ¿cómo pudo acoplarse el RNA autorreplicante (o gen primitivo) a un sistema metabólico que permitiera su replicación y traducción? Esta pregunta ha sido objeto de amplias discusiones por parte de Kauffman, 1993, quien propuso que la vida debió haber comenzado como un conjunto, con una complejidad mínima, de péptidos y RNA catalíticos que conformaban una red metabólica o un colectivo autocatalítico, sin genoma. No obstante la amplia aceptación de la hipótesis del mundo RNA, los defensores de la misma no han podido explicar la síntesis abiótica de ribo-nucleótidos o monómeros que componen al RNA dado que son muy inestables en soluciones acuosas. Para Schwartz, (Schwartz, 1997; Schwartz, 1998) no hay modelos que expliquen cómo pudieron unirse las bases nitrogenadas a ribosa y al grupo fosfato, ni cómo pudo darse la síntesis abiótica de ribosa. Además la síntesis de oligómeros es un problema (Joyce y Orgel, 1998), puesto que el grupo 5’ fosfato se une en tres posiciones distintas (5’- 5’ -pirofosfato, y enlaces fosfodiester 5’- 2’ y 5’- 3’), mientras que el RNA presenta únicamente enlaces 5’- 3’, utilizando el grupo hidroxilo 3’ que es el menos reactivo. La síntesis química debió estar mediada por catalizadores específicos como la arcilla (montmorillonita) que favorecen los enlaces 5’-3’ fosfodiester. Para que la hipótesis del mundo RNA se convierta en una teoría comprobada hay que reconstruir las condiciones del entorno físico y químico que la hicieron posible. En este sentido mantiene vigencia la pregunta sobre la existencia de un proto-metabolismo anterior al mundo RNA.
Para responder a esta inquietud debemos retomar el hilo conductor de la propuesta de Oparin e ir más adelante, veamos. Oparin (Oparin, 1952 [1938]; Oparin, 1991 [1950]), había propuesto que en la tierra primitiva la composición de la atmósfera era diferente a la actual y estaba conformada por vapor de agua (H2O), gas carbónico (CO2), nitrógeno (N2), hidrógeno (H2), metano (CH4) y amoniaco (NH3), pero solamente hasta el año de 1953 mientras el mundo científico celebraba con alborozo el descubrimiento de la estructura del DNA por Watson y Crick, al otro lado del Atlántico Urey y Miller, 1953, se dedicaban a someter a prueba la hipótesis de Oparin. Construyeron un dispositivo que permitía reproducir esas condiciones atmosféricas en el cual introdujeron un generador de chispas eléctricas de alto voltaje y un sistema de destilación y condensación que permitía recoger en un recipiente con agua los productos orgánicos solubles que se formaban. En estas condiciones demostraron que se podían generar una diversidad de moléculas entre las que se destacaban los siguientes aminoácidos: glicina, ácido glicólico, alanina, ácido láctico, ácido alfa amino-N-butírico, norvalina, ácido aspártico, ácido succínico, sarcosina, N-etilglicina, ácido α-amino-isobutirico, isovalina, β- alanina, ácido γ- aminobutírico, N-metil alanina, prolina, ácido pipecólico, etc. Además por condensación de ácido cianhídrico (HCN) se producía el nucleótido adenosina y por polimerización de formaldehido (HCHO) azúcares. Aunque muchos de estos amino ácidos no corresponden a los codificados en los organismos conocidos, si están presentes los más abundantes en las proteínas estudiadas de diferente procedencia biológica como son glicina, alanina, aspártico. El experimento de Miller y Urey aunque está lejos de explicar el origen de la vida, sí demuestra la posibilidad de generar abióticamente las unidades constitutivas de las proteínas. Sorprendentemente el meteorito de Murchison encontrado en Australia en septiembre de 1969, presenta en su materia orgánica extraterrestre los mismos aminos ácidos y en proporciones similares a los obtenidos por Miller y Urey (Orgel, 1994).
Hubo que esperar casi 30 años, para que otro investigador Sidney (Fox, 1984) ideara un sistema de polimerización abiótica de los aminoácidos obtenidos por el experimento de Miller y Urey. Utilizó la mezcla descrita de aminoácidos, la calentó y secó a 200 °C, después la dejo enfriar y solubilizar en agua para repetir el proceso encontrando que se formaban microesferas de proteinoides. Los proteinoides difieren de las proteínas que conocemos en dos aspectos: 1. están conformados en su mayoría por polímeros de aminoácidos abióticos, aunque predomina glicina y alanina al igual que en las proteínas actuales. 2. Además de los enlaces peptídicos característicos de las proteínas se presentan numerosos enlaces que involucran radicales de amino ácidos, dando así lugar a estructuras ramificadas. No obstante, estos proteinoides exhiben características notables como la de presentar actividad enzimática claramente reconocible y aglutinarse entre ellos dando estructuras globuladas o microesferas con membrana semipermeable, tipo coacervado. A partir de estos descubrimientos surgió inevitablemente la idea de que el primitivo mundo RNA pudo existir pero acoplado a proteinoides (Fox, 1984) u a otro tipo de metabolismo autótrofo en el interior de las microesferas mencionadas.
Blomberg, 1997, ha defendido que el mundo RNA surgió acoplado al sistema peptídico generado prebióticamente, a partir del cual se desarrollaron las proteínas codificadas. De Duve, 1998, sostuvo que este sistema inicial sería un protometabolismo diferente aunque precursor del metabolismo de los seres vivos actuales. La existencia de este protometabolismo es compatible con los hallazgos de Fox, 1984, que confirmaron la actividad catalítica de los péptidos obtenidos, además de demostrar que las microesferas de proteinoides presentaban crecimiento y fisión, estabilidad a largo plazo y especificidad al nivel de la secuencia de los proteinoides. Como bien lo enunció Kauffman (Kauffman, 1993; Kauffman, 1995) -ninguna molécula, incluyendo al DNA, cataliza su propia formación-. Una molécula que se autorreplicara tampoco sería capaza de organizar en torno suyo un sistema metabólico de mantenimiento; puesto que no sería capaz de sintetizar sus propios componentes, mientras que lo contrario si es posible, es decir un metabolismo si es capaz de generar replicación. De nuevo podemos inferir que la información digital supone la analógica y no al contrario, en conformidad con la afirmación de Kauffman (Kauffman, 1993; Kauffman, 1995) de que la vida primitiva funcionaba sin genoma. La información digital, por sí sola, carece de sentido, a menos que exista un sistema de información analógica, un código que relacione ambos sistemas y un mecanismo de traducción. No hay que confundir -vida- con -reproducción-; puesto que la reproducción es una consecuencia del crecimiento y éste del aprovechamiento energético que el metabolismo posibilita (Fox, 1984). En últimas de lo que se trata es de adoptar una definición más amplia de reproducción,
-resulta suficiente que los catalizadores se mantengan, a grandes rasgos, de manera estadística. La población de moléculas (...) se está reproduciendo sin que haya replicación exacta- (Dyson, 1999).
En consecuencia, aunque la vida es mucho más que genes, hay que explicar el origen de la información genética. De acuerdo a la discusión anterior la información genética contenida en el DNA ocurrió en varios pasos: 1. Emergencia por autoorganización de un protometabolismo basado en péptidos generados abióticamente. 2. Aparición de nucleótidos y RNAs con la capacidad de replicarse. 3. En este nuevo sistema conformado por péptidos y RNAs surgirían las proteínas codificadas, el código genético y por tanto la traducción. 4. Desarrollo de procesos de transcripción reversa y replicación fiel de DNA. 5. El sistema protometabolismo es remplazado gradualmente por el actual.
RELACIÓN SECUENCIA LINEAL Y ESTRUCTURA EN EL CASO DEL RNA
Un problema interesante que se ha investigado empírica y computacionalmente es el de la relación entre secuencias lineales y estructuras secundarias para el caso específico de moléculas de RNA. El estudio de estas relaciones permite entender el aspecto dual de la información contenida en este tipo de moléculas. Para empezar consideremos que el número de cadenas posibles de RNA que tiene una longitud de 100 ribonucleótidos es de 4100, lo cual equivale aproximadamente a 1060. No sobra recordar que este es un número astronómicamente inmenso si se tiene en cuenta que el número de protones en el universo está estimado en 1080. Para simplificar las cosas consideremos el espacio de todas las cadenas binarias de longitud 100 que pueden imaginarse, las cuales representamos en un hiperespacio de 100 dimensiones. De acuerdo a las siguientes figuras (Fig. 6). Este espacio que contiene 2100 puntos o cadenas posibles esta hiperconectado, eso quiere decir que los puntos más distantes se encuentran a solo 100 pasos mutaciones es decir sustituciones de un 0 por un 1 o de un 1 por un 0. Cuando se tiene una población de cadenas que se replican con cierta tasa de error en condiciones controladas de selección in vitro, se observa que después de varios ciclos de replicación-selección, es posible identificar una secuencia maestra promedio que caracteriza a la población, dado que cada cadena individual corresponde a mutantes alejadas 1, 2, 3, 4, 5, etc, pasos mutacionales de ella. Es decir, no se obtiene una única secuencia sino una población más o menos dispersa de cadenas similares. En el espacio de secuencia podemos pues ubicar una nube de puntos que corresponde a todas las variantes existentes en la cuasi-especie. No obstante, esta población no ha sido seleccionada como tal por la secuencia sino por la función o afinidad específica a una sustancia determinada e impuesta por las condiciones de selección in vitro. Esta afinidad depende de la estructura secundaria y terciaria de las cadenas de RNA involucradas. Es decir se seleccionan fenotipos, lo que trae como consecuencia un desplazamiento de la composición genética característica.

Hay que destacar que la distribución de las secuencias individuales en el espacio de secuencias no es uniforme, dado que han sido objeto de selección por la estabilidad de su estructura secundaria. La distribución irregular de secuencias en el espacio total de secuencias constituye un paisaje adaptativo rugoso de múltiples picos tipo Kauffman, 1993, en donde muchas posiciones quedan vacías y otras densamente pobladas por acción de la selección. Los experimentos de Schuster et al., 1994, han demostrado que mientras el espacio de secuencias posibles es 1060 para cadenas de 100 ribo-nucleótidos, el espacio de las configuraciones o formas es de solo 1027. Sin duda un número inmenso pero 1033 veces menor que el de secuencias. Este fenómeno es muy interesante porque da claves para la razón por la cual la evolución funciona al azar. Es decir en lugar que tener que buscar una aguja en el astronómicamente inmenso pajar del espacio de todas las secuencias posibles, en la realidad hay que buscarla en conjuntos limitados de pajares más pequeños (los picos adaptativos ocupados). El carácter funcional y la estabilidad estructural de las formas plegadas son el objeto directo de la selección pero dado que el espacio de secuencias es mucho más amplio que el de formas, se presentan redes neutras es decir trayectos largos o neutros dentro del espacio de secuencias en que la forma o estructura secundaria sigue siendo la misma. El espacio de secuencias está atravesado por una multitud de caminos neutros que conforman redes neutras las cuales se aproximan en algunos puntos donde una nueva mutación da acceso a una estructura secundaria diferente. La evolución molecular de las estructuras secundarias tiene lugar por saltos discontinuos que se presentan intercalados entre periodos muy largos de variación neutra gradual a nivel de la secuencia (Fontana y Schuster, 1998a; Fontana y Schuster, 1998b).
Muestreos estadísticos acerca de cómo varían las formas (estructuras secundarias) a medida que se introducen mutaciones puntuales en la secuencia revelaron resultados sorprendentes. Para la población de mutantes que varían en 1, 2 o 3 ribo-nucleótidos con respecto a una cadena especifica tomada al azar, la estructura secundaria que adoptan es idéntica (estas son las variaciones neutras que mencionábamos anterior-mente) o prácticamente similar a la original es decir no conlleva cambios de la forma. Cuando se estudian mutantes que varían en un número mayor de cutro se observa que dan lugar a formas similares a la original aunque francamente diferentes. Los mutantes mayor de siete dan lugar a formas muy diferentes y cuando se llega a un valor de 18 cam-bios mutacionales las cadenas resultantes se dispersan en el espacio de formas dando lugar a todas las formas posibles, en otras palabras pueden dar lugar a cualquier forma. Para mutantes mayores de 20, vuelven a aparecer formas que ya se habían obtenido es decir la situación es completamente aleatoria. Se estimó un valor estadístico de distancia mutacional promedio de 7,2 en que prácticamente cualquier forma altamente representada se encuentra. Esto quiere decir que si para el espacio de secuencias las más distantes se ubican a 100 pasos mutacionales, en el espacio de formas las más distantes están separadas en promedio solo 7,2 pasos mutacionales. Es decir existen muchas secuencias diferentes que dan lugar a la misma estructura secundaria y por tanto al cumplimiento de la misma función. Pero además, a partir de cualquier cadena tomada al azar es posible definir un espacio de cadenas vecinas que distan no más de ocho pasos mutacionales, que dan lugar a todas las formas posibles. Es decir que la correspondencia entre cadenas portadoras de información digital y sus correspondientes estructuras análogas, no hay una relación uno a uno, sino -de muchas secuencias a una forma- y -de una secuencia a muchas formas-. En otras palabras la información contenida en el RNA es muy plástica y por tanto es factible encontrar moléculas que cumplan una gran diversidad de tareas químicas o enzimáticas. Son, pues las moléculas ideales para el estudio del origen de la vida.
La vida no es un rompecabezas compuesto por un inmenso número de piezas, en el que cada una ocupa un único lugar determinado con alta precisión, y que por tanto tendría una probabilidad extremadamente baja de ser generada por azar. Por el contrario la vida es más bien una estructura plástica construida a su vez por piezas plásticas tipo LEGO, en el cual una misma pieza puede acomodarse a distintos lugares, existiendo muchas maneras de generar no una estructura perfecta, sino una estructura más o me-nos viable, pero susceptible de evolucionar por selección natural. En este caso la probabilidad de generar vida al azar es altamente probable, es incluso un hecho altamente favorecido en un mundo donde la química dependía de moléculas tipo RNA en un entorno de micro esferas conformadas por proteinoides.
De acuerdo a los estudios de Fontana y Schuster, 1998a y Fontana y Schuster, 1998b, se puede entrever que la información digital de los RNA puede ser objeto de una medida de Shannon, mediante la estimación del número de bits necesarios para identificar una en particular en el hiperespacio de todas las secuencias posibles, considerando las diferentes probabilidades con que ocupan los picos adaptativos. Igualmente puede ser medida como información de Chaitin si se comprime la secuencia eliminando los sectores repetidos dando lugar a una serie aleatoria que puede ser descrita con un algoritmo más corto. Por otra parte la información analógica es medible en términos de Shannon, teniendo en cuenta la probabilidad de encontrar una estructura secundaria dentro la población de estructuras secundarias posibles para una secuencia determinada. Por último la información analógica es medible con el algoritmo de Chaitin, si logramos identificar el número mínimo de pasos necesarios para plegar una secuencia dada en la conformación planar o estructura secundaria más probable. Aunque estas mediciones son muy interesantes en biología molecular para dilucidar la relación entre secuencia lineal y estructura, y entre estructura y función de los RNA, estos estudios proponen modelos para estudiar físicamente la dualidad análogo-digital propia de la información, a la vez que permiten captar el significado físico asociado a los conceptos de información de Shannon y Chaitin.
Los flujos de información a lo largo de la evolución y diversificación de la vida se han rastreado mediante el estudio comparativo de secuencias génicas, es decir de los registros digitales de información. Estos estudios han dado lugar a filogenias moleculares que en lo fundamental describen las diversas ramificaciones que ha sufrido la vida a partir de ancestros comunes. No obstante ha sido imposible detectar el ancestro común más antiguo (LUCA) y examinar con detalle las etapas más tempranas de la evolución biológica. Woese, 1998, propuso utilizar como molécula comparativa los RNA ribosomales, puesto que están presentes en todas las formas vivas tanto procariontes como eucariontes. Los árboles evolutivos de Woese et al., 1990, mostraron que la vida desde muy temprano se había dividido en tres grandes dominios, ramas o grupos monofiléticos conocidos como archea, bacteria y eucarya. Las archeas corresponden a bacterias que viven en ambientes extremos hiper-termófilos, sin oxígeno, altas salinidades y presiones como las chimeneas termales de los fondos oceánicos. Las bacterias corresponden a los conocidos como micro-organismos procarióticos que viven en condiciones aerobias y algunas con actividad fotosintética. Eucarya corresponde al ancestro de todos los organismos eucarióticos o poseedores de células nucleadas como los protozoarios en caso de ser unicelulares y hongos, plantas y animales en caso de ser multicelulares. Sin embargo las relaciones entre estos tres grandes dominios no han podido ser dilucidada, y se estimaba que el árbol estuviera posiblemente enraizado en la rama correspondiente a bacteria. La insuficiencia de la información digital (secuencias de RNA ribosonales) para resolver este problema dio lugar a la idea de (Caetano-Anollés, 2002) de incluir la información analógica estructural. La presencia de módulos estructurales altamente conservados en las moléculas de RNA ribosomal y el estudio de sus variaciones mediante algoritmos que permiten calcular las distancias entre estructuras secundarias mostró una señal filogenética importante que permite enraizar el árbol universal en eucarya. Aunque el problema sigue sin resolverse, la idea de que las estructuras secundarias son en sí mismas registros de información evolutiva que deben ser incluidos en los estudios filogenéticos ha sido bienvenida.
REFLEXIÓN FINAL
La idea revolucionaria de la biología molecular que nos presenta la naturaleza viviente como un texto de información digital, colocaba a la biología en consonancia con lo que Kant había escrito en carta a Hamman refiriéndose al libro de la naturaleza:
-La naturaleza es un libro, una carta, una fábula o como usted la quiera llamar. Suponiendo que conozcamos lo mejor posible todas sus letras, podemos silabear y pronunciar todas sus palabras y sepamos la lengua en que está escrito. .... Todo eso ya es suficiente para entender un libro, hacer juicios sobre él, sacar una característica o un extracto. - (Briefwechsel, Kant & Hamman, 1759 citado en Blumenberg, 2000 pp: 193-194).
Pero la metáfora de la naturaleza como texto, tiene otras connotaciones profundas puesto que remite a la pregunta sobre cómo se escribió el texto o libro de la vida. En este sentido el texto informativo, la existencia misma de DNA hay que verla como el resultado de un proceso de acumulación, registro y codificación de información a lo largo de toda la historia evolutiva de los linajes, aspecto que todavía no ha sido suficientemente entendido. La información genética no preexiste, no solo es el resultado de un proceso evolutivo, sino que además sigue evolucionando en cuanto se está actualizando permanentemente en cada generación. Por otra parte la información debe tener un significado el cual se corresponde con el cumplimiento de la función y, además, hay fragmentos de la misma que la selección natural desecha en algún momento, pero siempre manteniendo activo el procesamiento y codificación de la nueva información que surge. En otras palabras procesar información del ambiente para generar evolutivamente, gracias a la selección natural, programas genéticos informativos requiere del concurso de los organismos quienes proveen el contexto funcional de significancia de esta información. Podríamos afirmar que la selección natural favorece a los sistemas procesadores, codificadores y usuarios de información. En este sentido para entender toda la dinámica subyacente a los procesamientos de información hay que partir del reconocimiento de la dualidad analógico digital (Andrade, 2003; Andrade, 2009). En otras palabras la naturaleza viviente no es solo procesamiento de información digital sino sobre todo un sistema químico de información analógica que hizo posible la emergencia de la digital y hace posible la expresión de la misma. Completando la metáfora de Kant mencionada anteriormente, la naturaleza es un libro pero el texto es sola-mente aclaratorio y explicativo de las numerosas imágenes y figuras que contiene. Es decir la naturaleza es un texto analógico-digital.
La prioridad evolutiva de los sistemas basados en información analógica se debe a que actúan por sí solos en un medio químico y logran mantenerse en ciertos lapsos de tiempo sin necesidad de información digital. Es posible mantener la información analógica aunque cambie la información digital, si se tiene en cuenta que el espacio de secuencias es mucho más grande que el espacio de formas: en otras palabras, muchas secuencias (diferente información digital) pueden ajustarse a la misma forma (igual información analógica; Andrade, 2003; Andrade, 2009). Además, un sistema de mantenimiento puede generar un entorno químico lo suficientemente complejo como para la aparición de información digital y un sistema de codificación.
En este sentido seguir explorando el modelo dual de información representado por el RNA sigue siendo un proyecto apasionante y promisorio por los resultados que pueda seguir arrojando en un futuro cercano. No solo a nivel de problemas específicos de biología molecular como el entendimiento de los sistemas de regulación genética, sino abriendo toda una vía para el mejor entendimiento y formalización de la noción física de información. La información no sería únicamente la característica definitoria de la vida, sino de la naturaleza física en su conjunto.
AGRADECIMIENTOS
A la bióloga Lina María Caballero por la lectura paciente del manuscrito y valiosos comentarios. Al Departamento de Biología de la Universidad Nacional y en particular a la Sección de Genética y Biología molecular por su apoyo permanente.
BIBLIOGRAFÍA
ALTMAN S, et al., The RNA moiety of ribonuclease P is the catalytic subunit of the enzyme. Cell. 1983;35:849-857.
ANDRADE E. From external to internal measurement: a form theory approach to evolution. Biosystems. 2000;57:49-62.
ANDRADE E. Los Demonios de Darwin: Semiótica y Termodinámica de la Evolución Biológica. 2da Edición. Bogotá: Unibiblos; 2003.
ANDRADE E. La Ontogenia del Pensamiento Evolutivo. Colección Obra Selecta. Bogotá: Editorial Universidad Nacional de Colombia; 2009.
ASTBURY WT. Contribution to the Discussion. Cold Spring Harb. Symp Quant Biol. 1938;6:118-120.
ASTBURY WT . On the Structure of Biological Fibres and the Problem of Muscle. Proc Royal Society London. 1947;134:326.
BARTEL DP, SZOSTAK JW. Isolation of new ribozymes from a large pool of random sequences. Science. 1993;261:1411-1418.
BLOMBERG C. On the appearance of function and organisation in the origin of life. J theor Biol. 1997;187:541-554.
BLUMENBERG H. La legibilidad del mundo. Traducción de Pedro Madrigal Devesa. Ediciones Paidos Ibérica, S.A. Barcelona. 2000.
BRILLOUIN L. Science and Information Theory. New York: Academic Press; 1962.
BROOKS D, WILEY EO. Evolution as Entropy. University of Chicago Press, Chicago; 1988.
BUSS LW. The evolution of individuality. Nueva Jersey. Princeton University Press; 1987.
CAETANO-ANOLLÉS G Evolved RNA Secondary Structure and the Rooting of the Universal Tree of Life. J Mol Evol. 2002;54:333-345
CECH TR, et al., plicing RNA: Autoexcision and autocyclization of the ribosomal RNA intervening sequence of Tetrahymena. Cell. 1980;31:147-57.
CECH TR, BASS BL. Biological Catalysis by RNA. Annu Rev Biochem. 1982;55:599-629.
CHARGAFF E. Some recent studies on the composition and structure of nucleic acids. J Cell Compar Phys. 1951;38:41-59.
CHAITIN GJ. On the length of programs for computing finite binary sequences: Statical considerations. J Assoc Comput Mach.1969;16:145-159.
CRICK F. [1955]. On Degenerate templates and the Adaptor Hypothesis. Unpublished note to the RNA Tie Club. En Kay LE. Who wrote the book of life? A history of the genetic code. Stanford University Press. Stanford, California; 2000.
CRICK FHC. On protein synthesis. Symp Soc Exp Biol. 1958;12:138-163.
CRICK FHC. Central Dogma of Molecular Biology. Nature. 1970;227:561-563.
DAWKINS R. The Selfish Gene. New York City: Oxford University Press ISBN 0-19-286092-5; 1976.
DE DUVE C. Clues from present-day biology: the thioester world. En: Brack A, editor. The Molecular Origins of Life ed. Cambridge University Press. U.K; 1998.
DOUDNA J, CECH T. The Chemical Repertoire of Natural Ribozymes. Nature. 2002;418:222-228.
DYSON FJ. Los orígenes de la vida Cambridge University Press, Madrid; 1999
EIGEN M. Self-organization of Matter and the Evolution of Biological Macromolecules. Naturwissenschaften. 1971;58:465-523.
EIGEN M, GARDINER W, SCHUSTER P, WINKLER-OSWATITSCH R. The Origin oGenetic Information. Sci Am. 1981;244:78-94.
EIGEN M. Steps towards Life. Oxord University Press; 1992.
EKLAND EH. RNA-catalyzed RNA Polimerization Using Nucleoside Triphosphates. Nature. 1996;382:373-376.
FEARNLEY IM, WALKER JE. Initiation Codons in Mammalian Mitochondria: Differences in Genetic Code in the Organelle. Biochemistry. 1987;26(25):8247-8251.
FITCH WM. Evidence Suggesting a Partial Internal Duplication in the Ancestral Gene for Heme Containing Globines. J Mol Biol. 1966;16:1.
FONTANA W, SCHUSTER P. Shaping space: the possible and the attainable in RNA Genotype.phenotype mapping. J theor Biol. 1998a;194:491-515.
FONTANA W, SCHUSTER P. Continuity in evolution: On the nature of transitions. Science. 1998b;280:1451-1455.
FOX SW. Self-ordered polymers and propagative cell-like systems. Naturwissenschaften 1969;56:1-9.
FOX SW. Proteinoid Experiments and evolutionary Theory, in Beyond Neo-Darwinism. Ed. Mae-Wan Ho & Peter T. Saunders. London. Academic Press, Inc; 1984.
FREELAND SJ, KNIGHT RD, LANDWEBER LF. Do proteins predate DNA? Science. 1999;286:690-692.
GAMOW G. Possible Relation between Desoxyribonucleic Acid and Protein Structure. Nature. 1954;173:318.
GILBERT W. The RNA world. Nature. 1986;319:618.
GILBERT W, SOUZA SJ. Introns and the RNA World. En: Gesteland RF, Cech TR, Atkins JF. The RNA World. 2nd ed. Cold Spring Harbor Laboratory Press; 1998.
GUERRIER-TAKADA C, GARDINER K, MARSH T, PACE N, ALTMAN S. RNAsa P una ribozima. Cell. 1983;35:849-857.
HAIG D, HURST LD. A quantitative measure of error minimization in the genetic code. J Mol Biol. 1991;33(5):412-417.
HOFFMEYER J, EMMECHE C. Code-Duality and the Semiotics of Nature. En: Anderson M y Merrell F, editors. Semiotic Modeling. Mouton de Gruyter, Berlin and New York; 1991. p. 117-166.
HOAGLAND MB, STEPHENSON ML, SCOTT JF, HECHT LI, ZAMECNIK PC. A soluble ribonucleic acid and intermediate in protein synthesis. J Biol Chem. 1958;231:241-256.
JOYCE GF, ORGEL LE. Prospects for Understanding the Origin of the RNA world. En: Gesteland RF, Cech TR, Atkins JF. The RNA World. 2nd ed. Cold Spring Harbor Laboratory Press; 1998.
KAUFFMAN S. The Origins of Order Self-Organization and Selection in Evolution. Oxford University Press. New York; 1993.
KAUFFMAN S. At Home in the Universe: the Search for the Laws of Self-Organization and Complexity. Oxford University Press. New York; 1995.
KHORANA HG. Polynucleotide Synthesis and the Genetic Code. Fed Proc. 1965;24:1473-1587.
KIMURA M. The Neutral Theory of Molecular Evolution. Sci Am. 1979;241:94.
KIMURA M. The Neutralist Theory of Molecular Evolution. Cambridge Univesity Press, Cambridge; 1983.
LAZCANO A, MILLER SL. The Origin and Early Evolution of Life: Prebiotic Chemistry, the Pre – RNA World and Time. Cell. 1996;85:793-798.
LAZCANO A. El Origen de la Vida: Evolución Química y Evolución Biológica. 3ª edición. Editorial Trillas. México; 2007. p. 107.
LEWIN, B. Genes IV. Oxford University Press; 1990.
MANTEGNA RN, BULDYREV SV, AL GOLDBERGER AL, HAVLIN S, PENG CP,
SIMON M, STANLEY HE. Linguistic features of noncoding DNA sequences. Phys Rev Lett. 1994;73:3169-3172.
MILLER S. A Production of Amino Acid Under Possible Primitive Earth Conditions Science. 1953;117:528-529.
MONOD J. El azar y la necesidad. Ed. Orbis, S.A. Barcelona; 1970. p. 13.
NIREMBERG MW. On the Genetic Code. Cold SH Q B. 1963;28:549-557.
OPARIN A. [1950]. El origen de la vida. Editorial Panamericana. Colombia; 1991.
OPARIN AI. (1.ª publicado en 1938). El Origen de la Vida. New York: Dover; 1952.
ORGEL LE. The Origin of Life on Earth. Sci Am. 1994;77-83.
POOLE A, JEFFARES D, PENNY D. The Path from the RNA World. J Mol Evol. 1997;46:1-17.
SCHRÖDINGER E. (1944) ¿Qué es la vida? Tusquet editores; 1988.
SHANNON CE. A mathematical theory of communication. Bell Syst Tech J. 1948;27:379-423,623-656.
SCHUSTER P, FONTANA W, STADLER P, HOFACKER I. From Sequences to Shapes and back: a case study in RNA secondary structures. Proc Biol Sci. 1994;255:279-284.
SCHWARTZ AW. Speculation on the RNA Precursor Problem J theor Biol. 1997;187:523-527.
SCHWARTZ AW. Origins of the RNA World. En: Brack A, editor. The Molecular Origins of Life. Cambridge University Press. U.K; 1998.
SZATHMARY E. Coding coenzyme handles: a hypothesis for the origin of the genetic code. Proc Natl Acad Sci U S A. 1993;90:9916-9920.
VOLKESTEIN MV. 1994. Physical Approaches to Biological Evolution. Springer-Verlag. Berlin; 1994. p. 268.
WATSON J, CRICK F. Genetical Implications of the Structure of Desoxyribonucleic Acid. Nature. 1953;171:964-967.
WEINER AM, MAIZELS N. 3’ Terminal tRNA-like structure tag genomic RNA molecules for replication: implications for the origin of protein synthesis. Proc Natl Acad Sci U S A. 1987;84:7383-7387.
WOESE CR. The universal ancestor. Proc Natl Acad Sci U S A 1998;95:6854-6859.
WOESE CR, KANDLER O, WHEELIS ML Towards a natural system of organisms: proposal for the domains Archaea, Bacteria and Eucarya. Proc Natl Acad Sci U S A. 1990;87:4576-4579.
ZAUG AJ, CECH TR. The Intervening Sequence RNA of Tetrahymena is an Enzyme. Science. 1986;231:470.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2011 Acta Biológica Colombiana

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
1. La aceptación de manuscritos por parte de la revista implicará, además de su edición electrónica de acceso abierto bajo licencia Attribution-NonCommercial-ShareAlike 4.0 (CC BY NC SA), la inclusión y difusión del texto completo a través del repositorio institucional de la Universidad Nacional de Colombia y en todas aquellas bases de datos especializadas que el editor considere adecuadas para su indización con miras a incrementar la visibilidad de la revista.
2. Acta Biológica Colombiana permite a los autores archivar, descargar y compartir, la versión final publicada, así como las versiones pre-print y post-print incluyendo un encabezado con la referencia bibliográfica del articulo publicado.
3. Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
4. Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos institucionales, en su página web o en redes sociales cientificas como Academia, Researchgate; Mendelay) lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).