Estandarización del protocolo para la detección de las fusiones TMPRSS2:ERG y de la expresión de los genes EZH2, SPINK-1 y NKX3.1 en cáncer de próstata (CaP)
Protocol standardization for detection of TMPRSS2 fusions: ERG and gene expression of EZH2, SPINK-1 and NKX3.1 in prostate cancer (PCa)
DOI:
https://doi.org/10.15446/abc.v21n3.50477Palabras clave:
expresión génica, neoplasia intraepitelial prostática, neoplasias de próstata, progresión de la enfermedad, puntuación de Gleason. (es)disease progression, gene expression, Gleason score, prostate neoplasms, prostatic intraepithelial neoplasia. (en)
Descargas
En la actualidad no existe una herramienta que permita diferenciar pacientes con cáncer de próstata (CaP) de mal pronóstico de aquellos con enfermedad indolente que sólo requieren un seguimiento controlado de la enfermedad. Debido a la coexistencia de diferentes focos premalignos y malignos en el CaP, el entendimiento sobre el proceso de carcinogénesis requiere de un mejor conocimiento. Actualmente, la heterogeneidad morfológica en CaP es evaluada con la puntuación de Gleason, la cual está fuertemente relacionada con el pronóstico de la enfermedad, sin embargo, esto es insuficiente por lo que se trabaja actualmente en identificación de alteraciones moleculares que permitan identificar subtipos que puedan establecer de manera más precisa el pronóstico del paciente. Este estudio preliminar buscó la estandarización del método de cuantificación en muestras prostáticas de FFPE de la expresión de los transcritos de posibles biomarcadores, como los oncogenes SPINK-1 y EZH2, el supresor tumoral NKX3.1, en conjunto con la determinación de la presencia/ausencia del gen de fusión TMPRSS2:ERG, ya que estos transcritos se encuentran involucrados en aparentes eventos excluyentes de la evolución natural del CaP, que apoyan la posibilidad de una clasificación molecular para esta enfermedad.
At present doesn't exist tool to differentiate patients with prostate cancer (PCa) of poor prognosis of those with indolent disease that only require a controlled monitoring of the disease. Because of the coexistence of different premalignant and malignant foci in CaP, the understanding of the carcinogenesis process requires a better understanding. Currently, the morphological heterogeneity in PCa is evaluated with Gleason score, which is closely related to the prognosis of the disease, but this is insufficient so it is currently to work on identifying molecular alterations to identify subtypes that can establish more precisely the patient's prognosis. This preliminary study aimed to standardization of the method of quantification in prostatic samples of FFPE of expression of transcripts of possible biomarkers, such as the oncogenes, SPINK-1 y EZH2, the tumour suppressor, NKX3.1, together with the determination of the presence/absence of gene fusion, TMPRSS2:ERG, being that these transcripts are involved in apparent exclusive events of the natural evolution of PCa, that support the possibility of a molecular classification for this disease.
DOI: https://doi.org/10.15446/abc.v21n3.50477
ESTANDARIZACIÓN DEL PROTOCOLO PARA LA DETECCIÓN DE LAS FUSIONES TMPRSS2: ERG Y DE LA EXPRESIÓN DE LOS GENES EZH2, SPINK-1 y NKX3.1 EN CÁNCER DE PRÓSTATA (CaP)
Protocol Standardization for Detection of TMPRSS2 Fusions: ERG and Gene Expression of EZH2, SPINK-1 and NKX3.1 in Prostate Cancer (PCa)
Yenifer Yamile SEGURA MORENO1,2; Martha Lucía SERRANO LÓPEZ1,3.
1 Grupo Área de Investigaciones. Instituto Nacional de Cancerología. Calle 1 n°. 9-85. Bogotá, Colombia.
2 Departamento de Biología, Facultad de Ciencias, Universidad Nacional de Colombia. Bogotá, Colombia. Ciudad Universitaria.
3 Departamento de Química, Facultad de Ciencias, Universidad Nacional de Colombia. Ciudad Universitaria, edificio 451, oficina 326. Bogotá, Colombia.
For correspondence. mlserranol@unal.edu.co
Received: 11st May 2015, Returned for revision: 12th January 2016, Accepted: 1st June 2016.
Associate Editor: Argel Aguilar-Valles.
Citation/Citar este artículo como: Segura Moreno YY, Serrano López ML. Estandarización del protocolo para la detección de las fusiones TMPRSS2:ERG y de la expresión de los genes EZH2, SPINK-1 y NKX3.1 en cáncer de próstata (CaP). Acta biol. Colomb. 2016;21(3):533-542. DOI: https://doi.org/10.15446/abc.v21n3.50477
RESUMEN
En la actualidad no existe una herramienta que permita diferenciar pacientes con cáncer de próstata (CaP) de mal pronóstico de aquellos con enfermedad indolente que sólo requieren un seguimiento controlado de la enfermedad. Debido a la coexistencia de diferentes focos premalignos y malignos en el CaP, el entendimiento sobre el proceso de carcinogénesis requiere de un mejor conocimiento. Actualmente, la heterogeneidad morfológica en CaP es evaluada con la puntuación de Gleason, la cual está fuertemente relacionada con el pronóstico de la enfermedad, sin embargo, esto es insuficiente por lo que se trabaja actualmente en identificación de alteraciones moleculares que permitan identificar subtipos que puedan establecer de manera más precisa el pronóstico del paciente. Este estudio preliminar buscó la estandarización del método de cuantificación en muestras prostáticas de FFPE de la expresión de los transcritos de posibles biomarcadores, como los oncogenes SPINK-1 y EZH2, el supresor tumoral NKX3.1, en conjunto con la determinación de la presencia/ausencia del gen de fusión TMPRSS2:ERG, ya que estos transcritos se encuentran involucrados en aparentes eventos excluyentes de la evolución natural del CaP, que apoyan la posibilidad de una clasificación molecular para esta enfermedad.
Palabras clave: expresión génica, neoplasia intraepitelial prostática, neoplasias de próstata, progresión de la enfermedad, puntuación de Gleason.
ABSTRACT
At present doesn't exist tool to differentiate patients with prostate cancer (PCa) of poor prognosis of those with indolent disease that only require a controlled monitoring of the disease. Because of the coexistence of different premalignant and malignant foci in CaP, the understanding of the carcinogenesis process requires a better understanding. Currently, the morphological heterogeneity in PCa is evaluated with Gleason score, which is closely related to the prognosis of the disease, but this is insufficient so it is currently to work on identifying molecular alterations to identify subtypes that can establish more precisely the patient's prognosis. This preliminary study aimed to standardization of the method of quantification in prostatic samples of FFPE of expression of transcripts of possible biomarkers, such as the oncogenes, SPINK-1 y EZH2, the tumour suppressor, NKX3.1, together with the determination of the presence/absence of gene fusion, TMPRSS2:ERG, being that these transcripts are involved in apparent exclusive events of the natural evolution of PCa, that support the possibility of a molecular classification for this disease.
Keywords: disease progression, gene expression, Gleason score, prostate neoplasms, prostatic intraepithelial neoplasia.
INTRODUCCIÓN
De acuerdo al informe de la Agencia Internacional para la Investigación en Cáncer (IARC, por sus siglas en inglés), a nivel mundial el cáncer de próstata (CaP) es el segundo tipo de cáncer con mayor incidencia y la quinta causa de muerte por cáncer en hombres. En Colombia, el CaP es el cáncer con mayor incidencia y la segunda causa de mortalidad por cáncer en hombres (IARC, 2012). En la práctica clínica se cuenta con herramientas para el seguimiento y pronóstico del CaP como la medición sérica de PSA, el sistema TNM de estadificación y el puntaje de Gleason; sin embargo, no existe una herramienta que permita diferenciar pacientes con CaP de mal pronóstico de aquellos que sólo requieren un seguimiento controlado de la enfermedad (IARC, 2012).
La puntuación de Gleason es un sistema de clasificación para el CaP basado en el patrón histológico del tumor asociado a su grado de diferenciación celular. Debido a que el CaP se presenta de manera multifocal, con diferentes grados de diferenciación, el Gleason se define como la suma de los dos patrones más comunes (Epstein et al., 2005). Este sistema, debido a la heterogeneidad tanto histopatológica como molecular del tumor, es muy usado a nivel clínico pero no es suficiente para determinar el pronóstico de esta enfermedad.
En el CaP, como en otros tipos de cánceres, se estudian marcadores moleculares que permitan establecer subtipos que contribuyan a predecir la progresión de la enfermedad (Lapointe et al., 2007; Mehra et al., 2007; Tomlins et al., 2007; Alumkal et al., 2012; Barbieri et al., 2012; Börno et al., 2012). En este ámbito, se ha estudiado el papel de posibles biomarcadores como el supresor tumoral NKX3.1, el gen de fusión TMPRSS2:ERG y los oncogenes, SPINK-1 y EZH2, en aparentes eventos excluyentes en la evolución de la enfermedad, que podrían estar representando eventos tempranos y divergentes que podrían direccionar el curso de la enfermedad (Barbieri et al., 2012).
La pérdida de expresión de NKX3.1 y el incremento de la expresión de EZH2 se han asociado con CaP de mal pronóstico (Bowen et al., 2000;Varambally et al., 2002; Gurel et al., 2010). En la mayoría de los CaP se han identificado fusiones recurrentes entre el gen regulado por andrógenos, TMPRSS2 y los genes que codifican para factores de transcripción de la familia ETS, principalmente ERG. Las fusiones TMPRSS2-ERG que conducen a una sobreexpresión de ERG, están presentes en 50 % de los casos y se consideran un evento temprano en la carcinogénesis, presente en una proporción de las lesiones preneoplásicas denominadas Neoplasia Intraepitelial Prostática de Alto Grado (HGPIN por sus siglas en inglés) (Attard et al., 2008; Park et al., 2010). El inhibidor de serina peptidasa, Kazal tipo 1 (SPINK1) es una proteína secretada que se sobreexpresa específicamente en un subconjunto de cánceres con ausencia de la fusiones TMPRSS2-ERG (Paju et al., 2007; Tomlins et al., 2008; Leinonen et al., 2010; Leinonen et al., 2013; Lippolis et al., 2013) y se asocia con una disminución de la supervivencia (Ateeq et al., 2011).
Bajo este panorama, este estudio busco estandarizar los protocolos para detectar el nivel de expresión de los genes NKX3.1 y EZH2, e identificar la presencia de fusiones cromosómicas TMPRSS2-ERG en muestras de tejido fresco congelado y tejido fijado en formalina y embebido en parafina (FFPE) de CaP, que permitan realizar estudios futuros, con el fin de evaluar su potencial clínico en el pronóstico de esta enfermedad.
MATERIALES Y MÉTODOS
Muestras
Tejido fresco congelado
Se obtuvieron muestras de tejido fresco congelado conservado a -70 °C en gel Tissue-Tek (Ted Pella Inc., USA/ Canadá) provenientes de prostatectomías radicales de dos pacientes diferentes con diagnóstico de CaP que comprometen entre el 2-3 % del material procesado, con puntuación de Gleason 3+3, del Banco Nacional de Tumores Terry Fox (BNTTF) del Instituto Nacional de Cancerología de Colombia.
Tejidos fijados en formol y embebidos en parafina (FFPE por sus siglas en inglés)
Las muestras FFPE asociadas a diferentes etapas de progresión del CaP de un mismo paciente fueron tomadas de las obtenidas en el proyecto: "Exploración de potenciales biomarcadores predictores del pronóstico en pacientes con diagnóstico de Cáncer de Próstata (CaP)", del cual este proyecto forma parte y que cuenta con la aprobación del Comité de Ética e Investigaciones del Instituto Nacional de Cancerología (INC).
De este mismo paciente se obtuvieron cuatro muestras de tejido: dos focos tumorales del Gleason 3 y un foco del Gleason 4, tejido con presencia de HGPIN y tejido normal con hiperplasia prostática benigna (HPB), definidos según criterio del patólogo con base a criterios unificados, para la posterior extracción de RNA y sus respectivos análisis.
Línea celular PC3
Esta línea celular fue derivada de CaP independiente de andrógenos, es negativa para las fusiones y expresa de EZH2, por lo cual se empleó como control negativo durante la estandarización (Mertz et al., 2007; Börno et al., 2012; Shin y Kim, 2012). Esta línea fue amablemente cedida por el profesor Fabio Aristizábal de la Universidad Nacional de Colombia. Las condiciones de cultivo fueron 5 % de CO2 a 37 °C, en medio DMEM (Dulbecco/Vogt modified Eagle's minimal essential medium) suplementado con 10 % de suero fetal bovino y un coctel del 1 % compuesto de penicilina, estreptomicina y anfotericina B.
Extracción y cuantificación de ácidos nucleicos
La extracción RNA se realizó a partir de 4 mg de tejido fresco y 200 μL de sangre total, utilizando el estuche comercial MasterPure™ Complete DNA and RNA Purification (Epicentre, Madison, EUA). Para las muestras de tejido FFPE, este procedimiento se realizó a partir de siete punch de las zonas seleccionadas por microdisección y se empleó el estuche RecoverAll™ Total Nucleic Acid Isolation (Ambion, Life Technologies, CA, EUA). En el caso de la línea celular, la extracción del RNA se hizo a partir de 1x106 células empleando el estuche comercial SV Total RNA Isolation System (Promega, Madison, Wisconsin, EUA). Para todas las extracciones se tomaron en cuenta las especificaciones de los productores. Posteriormente, mediante el uso del fluorómetro Qubit® 2.0 (Invitrogen, Waltham, Massachusetts, USA) se determinó la cantidad de los ácidos nucleicos.
Retrotranscripción y amplificación (RT-PCR)
A partir del RNA extraído se llevó a cabo una RT-PCR en un solo paso con el uso del estuche comercial modificado KAPA SYBR FAST One-Step qRT-PCR (Kapa biosystems, Wilmington, Massachusetts, USA), en un termociclador PTC-200 PCR System (MJ Research, Reno, Nevada, EUA), empleando los iniciadores descritos en la Tabla 1, para las fusiones 1 y 2 de TMPRSS2:ERG, EZH2, NKX3.1, SPINK-1. Se utilizaron 10μl de KAPA SYBR FAST qPCR Master Mix (2X), 0,4μl de KAPA RT Mix (50X), 2μl de cada par de iniciadores, 4μl de muestra de RNA (1ng/μl) y 1,6μl de agua libre de nucleasas para la RT-PCR. Las condiciones térmicas empleadas fueron las establecidas por los proveedores, con excepción de la temperatura de alineamiento para cada transcrito (Tabla 1).
Los diferentes amplímeros fueron verificados en geles de agarosa al 2,5 % con bromuro de etidio, excepto para la fusión 2 que se corrió en un gel de agarosa al 1 %. La determinación de la presencia/ausencia de las fusiones TMPRSS2:ERG fue realizada por la visualización de los amplímeros correspondientes en el gel. Como genes de expresión constitutiva se usaron UBC y GAPDH.
Elaboración de las curvas estándar para análisis de la expresión génica de EZH2, NKX3.1, SPINK-1, UBC y GAPDH, por medio de qRT-PCR
Para la construcción de los estándares, se utilizó el cDNA amplificado con RT-PCR de la línea PC3, para EZH2, UBC y GAPDH, el cDNA de sangre para SPINK-1 y con cDNA de tejido normal de próstata para NKX3.1., purificado mediante el estuche comercial Illustra GFX PCR DNA and Gel Band Purification (GE Healthcare, Little Chalfont, United Kingdom) y se cuantificó mediante el uso del fluorómetro Qubit® 2.0 (Invitrogen, Waltham, Massachusetts, USA).
El cálculo del número de copias del stock de cDNA se realizó con la calculadora online de URI-Genomics and Sequencing-Center, 2004 (http://cels.uri.edu/gsc/cndna.html).
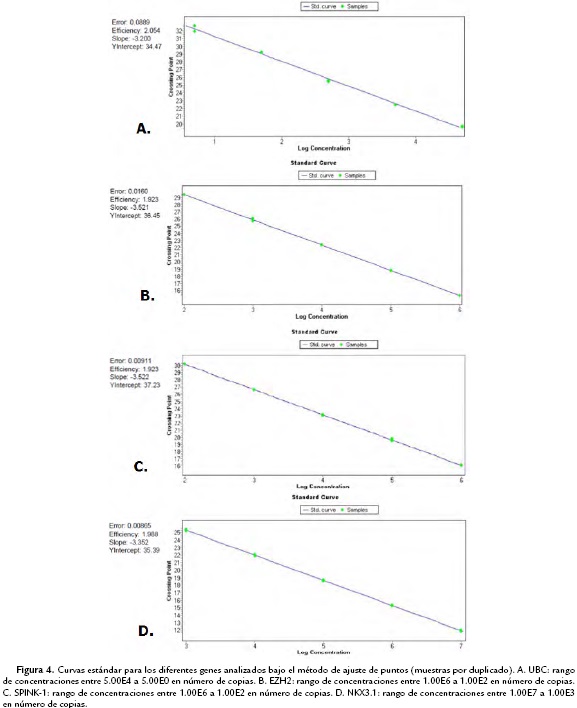
Para la curva de calibración se realizaron siete disoluciones seriadas a partir del stock de cDNA (Applied-Biosystems, 2003; Mohamed et al., 2004; Wacker y Godard, 2005; Al-Shanti et al., 2009; Wen et al., 2010). Todas las curvas comprenden un rango variable comprendidoentre 1,00E7a 1,00E1 de número de copias/μL, buscando obtener una eficiencia < 3 y un error <0,2. Se utilizaron las mismas condiciones estandarizadas para la RT-PCR, usando el termociclador LightCycler® 480 Real-Time PCR (Roche, Basilea, Suiza), omitiendo el paso de activación de la retrotranscriptasa, ya que en este caso se estaba partiendo de cDNA.
Las muestras fueron analizadas en duplicado. El cálculo de la eficiencia, según el método utilizado en este estudio, a partir de la pendiente de la curva de calibración se da de acuerdo con la ecuación: E = 10 [-1/pendiente].
Cuantificación de la expresión génica de EZH2, NKX3.1, SPINK-1, por medio de qRT-PCR.
Finalmente, con las curvas estándar de EZH2, SPINK-1, NKX3.1 y UBC se cuantificaron los niveles de expresión de estos transcritos, por duplicado, empleando una concentración de RNA de 2ng/μL. Se realizó cuantificación absoluta y a partir de esta se realizó la cuantificación relativa normalizando por el método de Pffalf (Pfaffl, 2001), el cual utiliza la siguiente ecuación:

[Normalización básica, sin uso de calibrador]. Donde E corresponde a la Eficiencia de la RT-PCR, Ref es el gen de referencia, Targ es el gen blanco y el Calibrador es el tejido sano.
RESULTADOS
Estandarización de la RT-PCR para analizar la presencia de las fusiones cromosómicas TMPRRS2:ERG
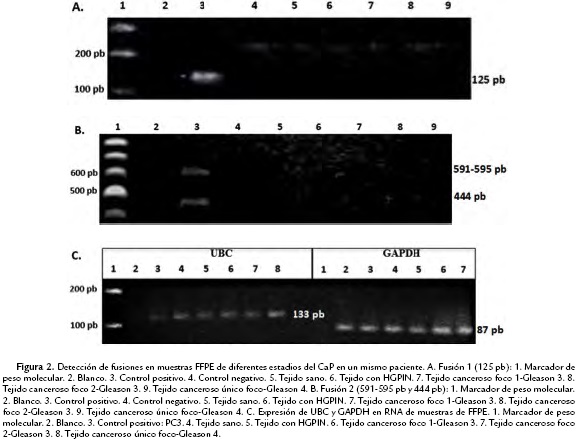
Inicialmente se evaluó la presencia de diferentes variantes transcripcionales de la fusión TMPRSS2:ERG, según el protocolo de Tomlins et al., (2005) (Tabla 1), en dos muestras de RNA extraído de tejido congelado realizando una RT-PCR con una concentración de iniciadores de 200 nM y una temperatura de anillamiento (Ta) de 60 °C. Cómo se observa en la Fig. 1A, las dos muestras fueron positivas para la fusión 2 (banda de 591 o 595 pb) y la muestra 2 fue positiva para la fusión 1 (banda de 125 pb), por lo que se tomó esta muestra como control positivo y la línea celular PC3 como el control negativo en la estandarización de la RT-PCR para la detección de dichas fusiones.
Debido a la presencia de múltiples bandas en la RT-PCR de las dos fusiones se realizaron variaciones en la Ta, entre 57 °C y 62 °C, y probando diferentes concentraciones de los iniciadores (50, 100 y 150 nM). Para la fusión 1, 61 °C y 100 nM son las condiciones óptimas para realizar su detección por RT-PCR (Fig. 1B).
Para la fusión 2 se ensayó un rango de Ta entre 62 y 68 °C y una concentración de iniciadores de 100 a 200 nM. Se encontró que la mejor amplificación se dio a una Ta de 65,5 °C y una concentración de iniciadores de 150 nM, aunque persisten las bandas de mayor tamaño (Fig 1C).
Con las condiciones determinadas en el tejido fresco se analizaron las muestras FFPE en las diferentes etapas de progresión de CaP (Figs. 2A y 2B). Aunque para la fusión 2 persisten algunas inespecificidades, al visualizar los resultados en un gel de agarosa al 1 % se pudo definir correctamente la banda obtenida en el control positivo. La adecuada extracción de RNA de todas las muestras fue confirmada por la expresión de los genes constitutivos UBC y GAPDH (Fig. 2C). Las muestras analizadas fueron negativas para dichas fusiones.
Estandarización de qRT-PCR para analizar expresión de EZH2, SPINK-1 y NKX3.1
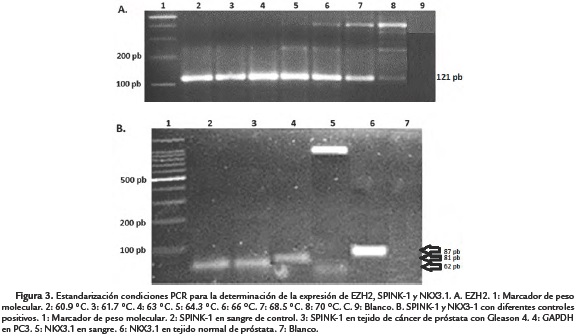
Se confirmó como ya se había mencionado, que PC3 expresa EZH2, y con el amplímero purificado de EZH2 y UBC, se realizaron las respectivas curvas de calibración. Debido a que la curva de fusión para EZH2 mostraba inespecificidades, se procedió a estandarizar la Ta, que inicialmente había sido de 60 °C, en un rango entre 60,9 °C y 70 °C, determinando que la mejor Ta para EZH2 es de 63 °C (Fig. 3A).
Con las condiciones iniciales, de 60 °C y 200 nM de iniciadores, para la RT-PCR de SPINK-1 y NKX3.1 se obtiene el producto esperado (Tabla 1). El RNA de sangre de un control y el de tejido de CaP son un buen control positivo para SPINK-1 y tan solo el RNA de tejido de próstata normal es un buen control positivo para NKX3.1, ya que en la de sangre del control se obtiene una banda de más de 1000 pb, que puede corresponder a contaminación genómica (Fig. 3B).
Con los amplímeros obtenidos se procedió a elaborar las respectivas curvas de calibración para qRT-PCR para los diferentes genes (Fig. 4). Para todos los casos se verificó la especificidad de las amplificaciones por qRT-PCR mediante la interpretación de las curvas de fusión.
Se realizó una cuantificación absoluta a partir de las curvas estándar del RNA de las mismas muestras a las cuales se les analizo el estado de fusión de TMPRSS2:ERG, las cuales fueron homogenizadas a una concentración final de 2ng/μL, y con los resultados de ésta cuantificación se obtuvieron los siguientes resultados para cuantificación relativa, usando el gen de referencia UBC (Tabla 2). En general, los datos muestran que en comparación con el tejido sano los niveles de EZH2 incrementan su expresión en todos los focos preneoplásicos y neoplásicos, mientras que la expresión de NKX3.1 y la de SPINK-1 incrementan su expresión en el foco preneoplásico pero disminuyen en los focos con CaP.
DISCUSIÓN
Clasificar los CaP de acuerdo a las alteraciones moleculares ayudaría a comprender la gran variabilidad de la enfermedad e identificar subtipos asociados con el riesgo (alto o bajo). El interés de trabajar con los biomarcadores planteados en este estudio proviene de la posible relación entre ellos en el desarrollo biológico de la enfermedad, postulada por algunos autores. Un estudio reciente ha demostrado que los altos niveles de ERG, resultantes de la fusión de TMPRSS2-ERG, reprimen la actividad de NKX3.1, un gen considerado como supresor tumoral, y plantea la posibilidad de un circuito de retro-alimentación en el CaP (Thangapazham et al., 2014). ERG reprime a NKX3.1 directamente mediante la unión a su promotor e indirectamente a través de la inducción de la EZH2, puesto que éste es un factor clave en el silenciamiento transcripcional del NKX3.1 (Kunderfranco et al., 2010).
En cuanto a SPINK-1, varios estudios han sugerido que sus niveles de mRNA se asocian a un subtipo agresivo asociado a recurrencia bioquímica con ausencia de TMPRSS2-ERG (Paju et al., 2007; Tomlins et al., 2008; Jhavar et al., 2009; Leinonen et al., 2010; Grupp et al., 2013; Leinonen et al., 2013; Lippolis et al., 2013), pero otros trabajos afirman que no hay relación (Grupp et al., 2013; Lippolis et al., 2013; Flavin et al., 2014). Este tipo de resultados resalta la importancia de realizar más estudios que logren determinar la utilidad clínica de estos biomarcadores.
En este estudio se estandarizó la técnica de RT-PCR para la identificación de la presencia/ausencia de las de fusión TMPRSS2:ERG según lo descrito en la Tabla 1. Aunque la técnica de FISH es la prueba de oro para la detección de las fusiones TMPRSS2-ERG, su uso tiene limitaciones. El FISH es costoso y dispendioso (Han et al., 2008; Pflueger et al., 2009), mientras que la RT-PCR ofrece algunas ventajas como un menor costo y su capacidad de discriminar diferentes variantes del gen de fusión TMPRSS2-ERG. Sin embargo, debido a su alta sensibilidad y la contaminación cruzada, la RT-PCR en ocasiones, puede dar resultados falsos positivos. Por lo tanto, RT-PCR es una técnica interesante como potencial complemento de FISH (Fernández-Serra et al., 2013).
Se encontró que las muestras FFPE podrían ser usadas para realizar estas pruebas de manera retrospectiva y realizar análisis sobre pronóstico. Aunque en el estudio de Tomlins et al., (2005), se utilizó el gen GAPDH para determinar de manera indirecta la cantidad e integridad del RNA y en este estudio además del GAPDH se utilizó el UBC, ambos asociados a estudios de CaP (Mori et al., 2008; Rose et al., 2005); el tamaño del amplímero de estos dos genes (87 y 133 pb, respectivamente) es inferior al esperado en la fusión 2, lo cual hace necesaria la confirmación de los casos negativos empleando un gen de referencia de mayor tamaño que demostrara que el RNA no está degradado.
Las diferentes bandas obtenidas de la amplificación de la fusión 2 (Fig. 1A y 1C; Fig. 2B), podrían corresponder con algunas de las ocho variantes detectadas por Wang et al., (2006) con el mismo par de iniciadores. El estudio de Tu et al., (2007), sugiere a partir de sus resultados y de la literatura (Clark et al., 2007; Yoshimoto et al., 2006), que las variantes: TMPRSS2 (exón 1)-ERG (exón 4) y TMPRSS2 (exón 1/2)-ERG (exón 5), coexisten en la mayoría de los casos, lo cual concuerda con lo encontrado en este estudio.
Debido a la multifocalidad del CaP, no es sencillo establecer subtipos moleculares ya que diferentes focos de un mismo paciente podrían presentar diferentes subtipos. Estudios anteriores han encontrado resultados discordantes, entre un 40 % a 50 %, con respecto a la presencia de la fusión TMPRSS2-ERG por FISH en diferentes focos de un mismo paciente, pero dentro de cualquiera foco el estado de este gen de fusión es homogéneo (Bastacky et al., 1995; Arora et al., 2004; Barry et al., 2007; Guo et al., 2009). Desde este enfoque, en este estudio es destacable el hecho que se hayan encontrado diferencias de expresión para EZH2 y NKX3.1 entre los dos focos de Gl 3, pero correspondencia entre un foco de Gl3 y el foco del Gl 4, lo cual sugiere una evolución molecular diferente en los distintos focos.
CONCLUSIONES
Este estudio contribuye a la implementación de metodologías para el estudio de la heterogeneidad del CaP y su aplicación clínica, para la discriminación entre los CaP indolentes y los agresivos mediante marcadores moleculares relacionados. Estos resultados son preliminares y se requiere de un estudio con un número significativo de pacientes con CaP para poder determinar la expresión de estos genes en el proceso de carcinogénesis.
AGRADECIMIENTOS A la Dirección de Investigación Sede Bogotá (DIB) por otorgar recursos para el desarrollo del presente trabajo. Al Grupo de Investigación en Biología del Cáncer y al Banco Nacional de Tumores Terry Fox (BNTTF) del Instituto Nacional de Cancerología por autorizar el uso de material y de sus instalaciones para la realización de este estudio.
REFERENCIAS
Al-Shanti N, Saini A, Stewart CE. Two-Step versus One-Step RNA-to-CT 2-Step and One-Step RNA-to-CT 1-Step: validity, sensitivity, and efficiency. J Biomol Tech. 2009;20(3):172-179.
Alumkal JJ y Herman JG. Distinct epigenetic mechanisms distinguish TMPRSS2-ERG fusion-positive and -negative prostate cancers. Cancer Discov. 2012;2(11):979-981. Doi:10.1158/2159-8290.CD-12-0392.
Arora R, Koch MO, Eble J N, Ulbright TM, Li L, Cheng L. Heterogeneity of Gleason grade in multifocal adenocarcinoma of the prostate. Cancer. 2004;100(11):2362-2366. Doi:10.1016/j.ccr.2010.05.026.
Applied Biosystems. Creating Standard Curves with Genomic DNA or Plasmid DNA Templates for Use in Quantitative PCR (2003). [Cited 6 may 2015]. Available at: http://www6.appliedbiosystems.com/support/tutorials/pdf/quant_pcr.pdf.
Ateeq B, Tomlins SA, Laxman B, Asangani IA, Cao Q, Cao X, et al. Therapeutic targeting of SPINK1-positive prostate cancer. Sci Transl Med. 2011;3(72):72ra17. Doi:10.1126/scitranslmed.3001498.
Attard G, Clark J, Ambroisine L, Mills IG, Fisher G, Flohr P, et al. Heterogeneity and clinical significance of ETV1 translocations in human prostate cancer. Br J Cancer. 2008;99(2):314-320. Doi:10.1038/sj.bjc.6604472.
Barbieri CE, Baca SC, Lawrence MS, Demichelis F, Blattner M, Theurillat JP, et al. Exome sequencing identifies recurrent SPOP, FOXA1 and MED12 mutations in prostate cancer. Nat Genet. 2012;44(6):685-689. Doi:10.1038/ng.2279.
Barry M, Perner S, Demichelis F, Rubin MA. TMPRSS2-ERG fusion heterogeneity in multifocal prostate cancer: clinical and biologic implications. Urology. 2007;70(4):630-633. Doi:10.1016/j.urology.2007.08.032.
Bastacky SI, Wojno KJ, Walsh PC, Carmichael MJ, Epstein JI. Pathological features of hereditary prostate cancer. J Urol. 1995;153(3 Pt 2):987-992.
Bowen C, Bubendorf L, Voeller HJ, Slack R, Willi N, Sauter G, et al. Loss of NKX3.1 expression in human prostate cancers correlates with tumor progression. Cancer Res. 2000;60(21):6111-6115.
Börno ST, Fischer A, Kerick M, Fälth M, Laible M, Brase JC, et al. Genome-wide DNA methylation events in TMPRSS2-ERG fusion-negative prostate cancers implicate an EZH2-dependent mechanism with miR-26a hypermethylation. Cancer Discov. 2012;2(11):1024-1035. Doi:10.1158/2159-8290.CD-12-0041.
Clark J, Merson S, Jhavar S, Flohr P, Edwards S, Foster CS, et al. Diversity of TMPRSS2-ERG fusion transcripts in the human prostate. Oncogene 2007;26(18):2667-73. Doi:10.1038/sj.onc.1210070.
Epstein JI, Allsbrook WC, Amin MB, Egevad LL, Committee IG. The 2005 International Society of Urological Pathology (ISUP) Consensus Conference on Gleason Grading of Prostatic Carcinoma. Am J Surg Pathol. 2005;29(9):1228-1242.
Fernández-Serra A, Rubio L, Calatrava A, Rubio-Briones J, Salgado R, Gil-Benso R. et. al. Molecular characterization and clinical impact of TMPRSS2-ERG rearrangement on prostate cancer: comparison between FISH and RT-PCR. Biomed Res Int. 2013;2013:465179. Doi:10.1155/2013/465179.
Flavin RJ, Pettersson A, Hendrickson WK, Fiorentino M, Finn SP, Kunz L, et al. SPINK1 Protein Expression and Prostate Cancer Progression. Clin Cancer Res. 2014;20(18):4904-4911. Doi:10.1158/1078-0432.CCR-13-1341.
Grupp K, Diebel F, Sirma H, Simon R, Breitmeyer K, Steurer S, et al. SPINK1 expression is tightly linked to 6q15- and 5q21-deleted ERG-fusion negative prostate cancers but unrelated to PSA recurrence. Prostate. 2013;73(15):1690-1698. Doi:10.1002/pros.22707.
Guo Cc, Zuo G, Cao D, Troncoso P, Czerniak BA. Prostate cancer of transition zone origin lacks TMPRSS2-ERG gene fusion. Mod Pathol. 2009;22(7):866-871. Doi:10.1038/modpathol.2009.57.
Gurel B, Ali TZ, Montgomery EA, Begum S, Hicks J, Goggins M, et al. NKX3.1 as a marker of prostatic origin in metastatic tumors. Am J Surg Pathol. 2010;34(8):1097-1105. Doi:10.1097/PAS.0b013e3181e6cbf3.
Han B, Mehra R, Dhanasekaran SM, Yu J, Menon A, Lonigro RJ, Wang X, Gong Y, et al. A fluorescence in situ hybridization screen for E26 transformation-specific aberrations: identification of DDX5-ETV4 fusion protein in prostate cancer. Cancer Res. 2008;68(18): 7629-7637. Doi:10.1158/0008-5472.CAN-08-2014.
IARC. International Agency for Research in Cancer. Globocan 2012: Estimated cancer incidence, mortality and prevalence worldwide in 2012. [Cited 6 may 2015]. Available at: http://globocan.iarc.fr/Pages/online.aspx.
Jhavar S, Brewer D, Edwards S, Kote-Jarai Z, Attard G, Clark J, et al. Integration of ERG gene mapping and gene-expression profiling identifies distinct categories of human prostate cancer. BJU Int. 2009;103(9):1256-1269. Doi:10.1111/j.1464-410X.2008.08200.x.
Kunderfranco P, Mello-Grand M, Cangemi R, Pellini S, Mensah A, Albertini V, et al. ETS transcription factors control transcription of EZH2 and epigenetic silencing of the tumor suppressor gene Nkx3.1 in prostate cancer: PLoS One. 2010;5(5):e10547. Doi: 10.1371/journal.pone.0010547.
Lapointe J, Li C, Giacomini CP, Salari K, Huang S, Wang P, et al. Genomic profiling reveals alternative genetic pathways of prostate tumorigenesis. Cancer Res. 2007;67(18): 8504-8510. Doi:10.1158/0008-5472.CAN-07-0673.
Leinonen KA, Saramäki OR, Furusato B. Loss of PTEN Is Associated with Aggressive Behavior in ERG-Positive Prostate Cancer, Cancer Epidemiol Biomarkers Prev. 2013;22(12):2333-44. Doi:10.1158/1055-9965.
Leinonen KA, Tolonen TT, Bracken H, Stenman UH, Tammela TL, Saramäki OR, et al. Association of SPINK1 expression and TMPRSS2: ERG fusion with prognosis in endocrine-treated prostate cancer: Clin Cancer Res. 2010;6(10):2845-2851. Doi:10.1158/1078-0432.CCR-09-2505.
Lippolis G, Edsjö A, Stenman UH, Bjartell A. A high-density tissue microarray from patients with clinically localized prostate cancer reveals ERG and TATI exclusivity in tumor cells. Prostate Cancer Prostatic Dis. 2013;16(2):145-150. Doi:10.1038/pcan.2013.7.
Mehra R, Tomlins SA, Shen R, Nadeem O, Wang L, Wei JT, et al. Comprehensive assessment of TMPRSS2 and ETS family gene aberrations in clinically localized prostate cancer. Mod Pathol. 2007;20(5):538-544. Doi:10.1038/modpathol.3800769.
Mertz KD, Setlur SR, Dhanasekaran SM, Demichelis F, Perner S, Tomlins SA, et al. Molecular characterization of TMPRSS2-ERG gene fusion in the NCI-H660 prostate cancer cell line: a new perspective for an old model. Neoplasia. 2007;9(3):200-206.
Mohamed N, Elfaitouri A, Fohlman J, Friman G, Blomberg J. A sensitive and quantitative single-tube real-time reverse transcriptase-PCR for detection of enteroviral RNA. J Clin Virol. 2004;30(2):150-156. Doi:10.1016/j.jcv.2003.08.016.
Mori R, Wang Q, Danenberg KD, Pinski JK, Danenberg PV. Both beta-actin and GAPDH are useful reference genes for normalization of quantitative RT-PCR in human FFPE tissue samples of prostate cancer. Prostate. 2008;68(14):1555-60. Doi:10.1002/pros.20815.
Paju A, Hotakainen K, Cao Y, Laurila T, Gadaleanu V, Hemminki A, et al. Increased expression of tumor-associated trypsin inhibitor, TATI, in prostate cancer and in androgen-independent 22Rv1 cells. Eur Urol. 2007;52(6):1670-1679. Doi: 10.1016/j.eururo.2007.01.096.
Park K, Tomlins SA, Mudaliar KM, Chiu YL, Esgueva R, Mehra R, et al. Antibody-based detection of ERG rearrangement-positive prostate cancer. Neoplasia. 2010;12(7):590-598.
Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res. 2001;29(9):e45.
Pflueger D, Rickman DS, Sboner A, Perner S, Lafargue CJ, Svensson MA, et al. N-myc downstream regulated gene 1 (NDRG1) is fused to ERG in prostate cancer. Neoplasia. 2009;11(8):804-811.
Rose A, Xu Y, Chen Z, Fan Z, Stamey TA, McNeal JE, et al. Comparative gene and protein expression in primary cultures of epithelial cells from benign prostatic hyperplasia and prostate cancer. Cancer Lett. 2005;227(2):213-22. Doi:10.1016/j.canlet.2005.01.037.
Shin YJ Y Kim JH. The role of EZH2 in the regulation of the activity of matrix metalloproteinases in prostate cancer cells. PLoS One. 2012;7(1):e30393. Doi: 10.1371/journal.pone.0030393.
Thangapazham R, Saenz F, Katta S, Mohamed AA, Tan SH, Petrovics G, et al. Loss of the NKX3.1 tumor suppressor promotes the TMPRSS2-ERG fusion gene expression in prostate cancer. BMC Cancer. 2014;14:16. Doi:10.1186/1471-2407-14-16.
Tomlins SA, Bjartell A, Chinnaiyan AM, Jenster G, Nam RK, Rubin MA, et al. ETS gene fusions in prostate cancer: from discovery to daily clinical practice. Eur Urol. 2009;56(2):275-286. Doi:10.1016/j.eururo.2009.04.036.
Tomlins SA, Laxman B, Dhanasekaran SM, Helgeson BE, Cao X, Morris DS, et al. Distinct classes of chromosomal rearrangements create oncogenic ETS gene fusions in prostate cancer. Nature. 2007;448(7153):595-599. Doi:10.1038/nature06024.
Tomlins SA, Rhodes DR, Perner S, Dhanasekaran SM, Mehra R, Sun X-W, et al. Recurrent Fusion of TMPRSS2 and ETS Transcription Factor Genes in Prostate Cancer. Science. 2005;310(5748): 644-648. Doi:10.1126/science.1117679.
Tomlins SA, Rhodes DR, Yu J, Varambally S, Mehra R, Perner S, et al. The role of SPINK1 in ETS rearrangement-negative prostate cancers. Cancer Cell. 2008;13(6):519-528. Doi:10.1016/j.ccr.2008.04.016.
Tu JJ, Rohan S, Kao J, Kitabayashi N, Mathew S, Chen YT. Gene fusions between TMPRSS2 and ETS family genes in prostate cancer: frequency and transcript variant analysis by RT-PCR and FISH on paraffin-embedded tissues. Mod Pathol. 2007;20(9):921-8. Doi:10.1038/modpathol.3800903.
URI Genomics & Sequencing Center. Calculator for determining the number of copies of a template (29 de ene 2004). [Cited 6 may 2015]. Available at: http://cels.uri.edu/gsc/cndna.html.
Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 2002;3(7):RESEARCH0034.
Varambally S, Dhanasekaran SM, Zhou M, Barrette TR, Kumar-Sinha C, Sanda MG, et al. The polycomb group protein EZH2 is involved in progression of prostate cancer. Nature. 2002;419(6907):624-629. Doi:10.1038/nature01075.
Wacker MJ y Godard MP. Analysis of one-step and two-step real-time RT-PCR using SuperScript III. J Biomol Tech. 2005;16(3):266-271.
Wang J, Cai Y, Ren C, Ittmann M. Expression of Variant TMPRSS2/ERG Fusion Messenger RNAs Is Associated with Aggressive Prostate Cancer. Cancer Res. 2006;66(17):8347-8351. Doi:10.1158/0008-5472.CAN-06-1966.
Wen G, Yang J, Luo Q, Hu Z, Song N, Zhang R, et al. A one-step real-time reverse transcription-polymerase chain reaction detection of classical swine fever virus using a minor groove binding probe. Vet Res Commun. 2010;34(4):359-369. Doi:10.1007/s11259-010-9363-8.
Yoshimoto M, Joshua AM, Chilton-Macneill S, Bayani J, Selvarajah S, Evans AJ, et al. Three-color FISH analysis of TMPRSS2/ERG fusions in prostate cancer indicates that genomic microdeletion of chromosome 21 is associated with rearrangement. Neoplasia 2006;8(6):465-469. Doi:10.1593/neo.06283.
Referencias
Al-Shanti N, Saini A, Stewart CE. Two-Step versus One-Step RNA-to-CT 2-Step and One-Step RNA-to-CT 1-Step: validity, sensitivity, and efficiency. J Biomol Tech. 2009;20(3):172–179.
Alumkal JJ y Herman JG. Distinct epigenetic mechanisms distinguish TMPRSS2-ERG fusion-positive and -negative prostate cancers. Cancer Discov. 2012;2(11):979-981. Doi:10.1158/2159-8290.CD-12-0392
Arora R, Koch MO, Eble J N, Ulbright TM, Li L, Cheng L. Heterogeneity of Gleason grade in multifocal adenocarcinoma of the prostate. Cancer. 2004;100(11):2362-2366. Doi:10.1016/j.ccr.2010.05.026
Applied Biosystems. Creating Standard Curves with Genomic DNA or Plasmid DNA Templates for Use in Quantitative PCR (2003). [Cited 6 may 2015]. Available at: http://www6.appliedbiosystems.com/support/tutorials/pdf/quant_pcr.pdf
Ateeq B, Tomlins SA, Laxman B, Asangani IA, Cao Q, Cao X, et al. Therapeutic targeting of SPINK1-positive prostate cancer. Sci Transl Med. 2011;3(72):72ra17. Doi:10.1126/scitranslmed.3001498
Attard G, Clark J, Ambroisine L, Mills IG, Fisher G, Flohr P, et al. Heterogeneity and clinical significance of ETV1 translocations in human prostate cancer. Br J Cancer. 2008;99(2):314-320. Doi:10.1038/sj.bjc.6604472
Barbieri CE, Baca SC, Lawrence MS, Demichelis F, Blattner M, Theurillat JP, et al. Exome sequencing identifies recurrent SPOP, FOXA1 and MED12 mutations in prostate cancer. Nat Genet. 2012;44(6):685-689. Doi:10.1038/ng.2279
Barry M, Perner S, Demichelis F, Rubin MA. TMPRSS2-ERG fusion heterogeneity in multifocal prostate cancer: clinical and biologic implications. Urology. 2007;70(4):630-633. Doi:10.1016/j.urology.2007.08.032
Bastacky SI, Wojno KJ, Walsh PC, Carmichael MJ, Epstein JI. Pathological features of hereditary prostate cancer. J Urol. 1995;153(3 Pt 2):987-992.
Bowen C, Bubendorf L, Voeller HJ, Slack R, Willi N, Sauter G, et al. Loss of NKX3.1 expression in human prostate cancers correlates with tumor progression. Cancer Res. 2000;60(21):6111-6115.
Börno ST, Fischer A, Kerick M, Fälth M, Laible M, Brase JC, et al. Genome-wide DNA methylation events in TMPRSS2-ERG fusion-negative prostate cancers implicate an EZH2-dependent mechanism with miR-26a hypermethylation. Cancer Discov. 2012;2(11):1024-1035. Doi:10.1158/2159-8290.CD-12-0041
Clark J, Merson S, Jhavar S, Flohr P, Edwards S, Foster CS, et al. Diversity of TMPRSS2-ERG fusion transcripts in the human prostate. Oncogene 2007;26(18):2667-73. Doi:10.1038/sj.onc.1210070
Epstein JI, Allsbrook WC, Amin MB, Egevad LL, Committee IG. The 2005 International Society of Urological Pathology (ISUP) Consensus Conference on Gleason Grading of Prostatic Carcinoma. Am J Surg Pathol. 2005;29(9):1228-1242.
Fernández-Serra A, Rubio L, Calatrava A, Rubio-Briones J, Salgado R, Gil-Benso R. et. al. Molecular characterization and clinical impact of TMPRSS2-ERG rearrangement on prostate cancer: comparison between FISH and RT-PCR. Biomed Res Int. 2013;2013:465179. Doi:10.1155/2013/465179
Flavin RJ, Pettersson A, Hendrickson WK, Fiorentino M, Finn SP, Kunz L, et al. SPINK1 Protein Expression and Prostate Cancer Progression. Clin Cancer Res. 2014;20(18):4904-4911. Doi:10.1158/1078-0432.CCR-13-1341
Grupp K, Diebel F, Sirma H, Simon R, Breitmeyer K, Steurer S, et al. SPINK1 expression is tightly linked to 6q15- and 5q21-deleted ERG-fusion negative prostate cancers but unrelated to PSA recurrence. Prostate. 2013;73(15):1690-1698. Doi:10.1002/pros.22707
Guo Cc, Zuo G, Cao D, Troncoso P, Czerniak BA. Prostate cancer of transition zone origin lacks TMPRSS2-ERG gene fusion. Mod Pathol. 2009;22(7):866-871. Doi:10.1038/modpathol.2009.57
Gurel B, Ali TZ, Montgomery EA, Begum S, Hicks J, Goggins M, et al. NKX3.1 as a marker of prostatic origin in metastatic tumors: Am J Surg Pathol. 2010;34(8):1097-1105. Doi:10.1097/PAS.0b013e3181e6cbf3
Han B, Mehra R, Dhanasekaran SM, Yu J, Menon A, Lonigro RJ, Wang X, Gong Y, et al. A fluorescence in situ hybridization screen for E26 transformation-specific aberrations: identification of DDX5-ETV4 fusion protein in prostate cancer. Cancer Res. 2008;68(18): 7629-7637. Doi:10.1158/0008-5472.CAN-08-2014
IARC. International Agency for Research in Cancer. Globocan 2012: Estimated cancer incidence, mortality and prevalence worldwide in 2012. [Cited 6 may 2015]. Available at: http://globocan.iarc.fr/Pages/online.aspx.
Jhavar S, Brewer D, Edwards S, Kote-Jarai Z, Attard G, Clark J, et al. Integration of ERG gene mapping and gene-expression profiling identifies distinct categories of human prostate cancer. BJU Int. 2009;103(9):1256-1269. Doi:10.1111/j.1464-410X.2008.08200.x
Kunderfranco P, Mello-Grand M, Cangemi R, Pellini S, Mensah A, Albertini V, et al. ETS transcription factors control transcription of EZH2 and epigenetic silencing of the tumor suppressor gene Nkx3.1 in prostate cancer: PLoS One. 2010;5(5):e10547. Doi: 10.1371/journal.pone.0010547
Lapointe J, Li C, Giacomini CP, Salari K, Huang S, Wang P, et al. Genomic profiling reveals alternative genetic pathways of prostate tumorigenesis. Cancer Res. 2007;67(18): 8504-8510. Doi:10.1158/0008-5472.CAN-07-0673.
Leinonen KA, Saramäki OR, Furusato B. Loss of PTEN Is Associated with Aggressive Behavior in ERG-Positive Prostate Cancer, Cancer Epidemiol Biomarkers Prev. 2013;22(12):2333-44. Doi:10.1158/1055-9965
Leinonen KA, Tolonen TT, Bracken H, Stenman UH, Tammela TL, Saramäki OR, et al. Association of SPINK1 expression and TMPRSS2: ERG fusion with prognosis in endocrine-treated prostate cancer: Clin Cancer Res. 2010;6(10):2845-2851. Doi:10.1158/1078-0432.CCR-09-2505
Lippolis G, Edsjö A, Stenman UH, Bjartell A. A high-density tissue microarray from patients with clinically localized prostate cancer reveals ERG and TATI exclusivity in tumor cells. Prostate Cancer Prostatic Dis. 2013;16(2):145-150. Doi:10.1038/pcan.2013.7
Mehra R, Tomlins SA, Shen R, Nadeem O, Wang L, Wei JT, et al. Comprehensive assessment of TMPRSS2 and ETS family gene aberrations in clinically localized prostate cancer. Mod Pathol. 2007;20(5):538-544. Doi:10.1038/modpathol.3800769
Mertz KD, Setlur SR, Dhanasekaran SM, Demichelis F, Perner S, Tomlins SA, et al. Molecular characterization of TMPRSS2-ERG gene fusion in the NCI-H660 prostate cancer cell line: a new perspective for an old model. Neoplasia. 2007;9(3):200-206.
Mohamed N, Elfaitouri A, Fohlman J, Friman G, Blomberg J. A sensitive and quantitative single-tube real-time reverse transcriptase-PCR for detection of enteroviral RNA. J Clin Virol. 2004;30(2):150-156. Doi:10.1016/j.jcv.2003.08.016
Mori R, Wang Q, Danenberg KD, Pinski JK, Danenberg PV. Both beta-actin and GAPDH are useful reference genes for normalization of quantitative RT-PCR in human FFPE tissue samples of prostate cancer. Prostate. 2008;68(14):1555-60. Doi:10.1002/pros.20815
Paju A, Hotakainen K, Cao Y, Laurila T, Gadaleanu V, Hemminki A, et al. Increased expression of tumor-associated trypsin inhibitor, TATI, in prostate cancer and in androgen-independent 22Rv1 cells. Eur Urol. 2007;52(6):1670-1679. Doi: 10.1016/j.eururo.2007.01.096
Park K, Tomlins SA, Mudaliar KM, Chiu YL, Esgueva R, Mehra R, et al. Antibody-based detection of ERG rearrangement-positive prostate cancer. Neoplasia. 2010;12(7):590-598.
Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res. 2001;29(9):e45.
Pflueger D, Rickman DS, Sboner A, Perner S, Lafargue CJ, Svensson MA, et al. N-myc downstream regulated gene 1 (NDRG1) is fused to ERG in prostate cancer. Neoplasia. 2009;11(8):804-811.
Rose A, Xu Y, Chen Z, Fan Z, Stamey TA, McNeal JE, et al. Comparative gene and protein expression in primary cultures of epithelial cells from benign prostatic hyperplasia and prostate cancer. Cancer Lett. 2005;227(2):213-22. Doi:10.1016/j.canlet.2005.01.037
Shin YJ Y Kim JH. The role of EZH2 in the regulation of the activity of matrix metalloproteinases in prostate cancer cells. PLoS One. 2012;7(1):e30393. Doi: 10.1371/journal.pone.0030393
Thangapazham R, Saenz F, Katta S, Mohamed AA, Tan SH, Petrovics G, et al. Loss of the NKX3.1 tumor suppressor promotes the TMPRSS2-ERG fusion gene expression in prostate cancer. BMC Cancer. 2014;14:16. Doi:10.1186/1471-2407-14-16.
Tomlins SA, Bjartell A, Chinnaiyan AM, Jenster G, Nam RK, Rubin MA, et al. ETS gene fusions in prostate cancer: from discovery to daily clinical practice. Eur Urol. 2009;56(2):275-286. Doi:10.1016/j.eururo.2009.04.036
Tomlins SA, Laxman B, Dhanasekaran SM, Helgeson BE, Cao X, Morris DS, et al. Distinct classes of chromosomal rearrangements create oncogenic ETS gene fusions in prostate cancer. Nature. 2007;448(7153):595-599. Doi:10.1038/nature06024
Tomlins SA, Rhodes DR, Perner S, Dhanasekaran SM, Mehra R, Sun X-W, et al. Recurrent Fusion of TMPRSS2 and ETS Transcription Factor Genes in Prostate Cancer. Science. 2005;310(5748): 644-648. Doi:10.1126/science.1117679
Tomlins SA, Rhodes DR, Yu J, Varambally S, Mehra R, Perner S, et al. The role of SPINK1 in ETS rearrangement-negative prostate cancers. Cancer Cell. 2008;13(6):519-528. Doi:10.1016/j.ccr.2008.04.016
Tu JJ, Rohan S, Kao J, Kitabayashi N, Mathew S, Chen YT. Gene fusions between TMPRSS2 and ETS family genes in prostate cancer: frequency and transcript variant analysis by RT-PCR and FISH on paraffin-embedded tissues. Mod Pathol. 2007;20(9):921-8. Doi:10.1038/modpathol.3800903
URI Genomics & Sequencing Center. Calculator for determining the number of copies of a template (29 de ene 2004). [Cited 6 may 2015]. Available at: http://cels.uri.edu/gsc/cndna.html
Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol. 2002;3(7):RESEARCH0034.
Varambally S, Dhanasekaran SM, Zhou M, Barrette TR, Kumar-Sinha C, Sanda MG, et al. The polycomb group protein EZH2 is involved in progression of prostate cancer. Nature. 2002;419(6907):624-629. Doi:10.1038/nature01075
Wacker MJ y Godard MP. Analysis of one-step and two-step real-time RT-PCR using SuperScript III. J Biomol Tech. 2005;16(3):266-271.
Wang J, Cai Y, Ren C, Ittmann M. Expression of Variant TMPRSS2/ERG Fusion Messenger RNAs Is Associated with Aggressive Prostate Cancer. Cancer Res. 2006;66(17):8347-8351. Doi:10.1158/0008-5472.CAN-06-1966
Wen G, Yang J, Luo Q, Hu Z, Song N, Zhang R, et al. A one-step real-time reverse transcription-polymerase chain reaction detection of classical swine fever virus using a minor groove binding probe. Vet Res Commun. 2010;34(4):359-369. Doi:10.1007/s11259-010-9363-8
Yoshimoto M, Joshua AM, Chilton-Macneill S, Bayani J, Selvarajah S, Evans AJ, et al. Three-color FISH analysis of TMPRSS2/ERG fusions in prostate cancer indicates that genomic microdeletion of chromosome 21 is associated with rearrangement. Neoplasia 2006;8(6):465-469. Doi:10.1593/neo.06283
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Licencia
Derechos de autor 2016 Acta Biológica Colombiana

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
1. La aceptación de manuscritos por parte de la revista implicará, además de su edición electrónica de acceso abierto bajo licencia Attribution-NonCommercial-ShareAlike 4.0 (CC BY NC SA), la inclusión y difusión del texto completo a través del repositorio institucional de la Universidad Nacional de Colombia y en todas aquellas bases de datos especializadas que el editor considere adecuadas para su indización con miras a incrementar la visibilidad de la revista.
2. Acta Biológica Colombiana permite a los autores archivar, descargar y compartir, la versión final publicada, así como las versiones pre-print y post-print incluyendo un encabezado con la referencia bibliográfica del articulo publicado.
3. Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
4. Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos institucionales, en su página web o en redes sociales cientificas como Academia, Researchgate; Mendelay) lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).