Identificación de una colección de microalgas aisladas de Costa Rica mediante secuenciación de ADNr 18S
Identification of a Microalgae Collection Isolated from Costa Rica by 18S rDNA Sequencing
DOI:
https://doi.org/10.15446/abc.v23n2.68088Palabras clave:
biotecnología microalgal, filogenia, identificación molecular. (es)microalgal biotechnology, molecular identification, phylogeny. (en)
Descargas
Las microalgas componen un diverso grupo polifilético de microorganismos fotosintéticos. Debido a su potencial biotecnológico, los estudios para aislar e identificar nuevas cepas han incrementado, por lo que es necesario el desarrollo de nuevas técnicas para su correcta identificación y clasificación. Utilizando herramientas de biología molecular, en este estudio se analizó el gen del ADNr 18S de 12 cepas microalgales aisladas de diferentes regiones de Costa Rica, resultando seis pertenecientes a la clase Trebouxiophyceae, tres a Chlorophyceae, dos a Prymnesiophyceae y una a Cyanidiophyceae. Este estudio reporta por primera vez la identificación molecular de cepas microalgales aisladas de Costa Rica, resaltando la diversidad de estos microorganismos en el país.
Microalgae integrate a diverse polyphyletic group of photosynthetic microorganisms. Due to its biotechnological potential, studies to isolate and identify new strains have increased, hence, it is necessary to develop new techniques for their correct identification and classification. Using molecular biology tools, this study analyzed the 18S rDNA gene from 12 microalgal strains isolated from different regions of Costa Rica, resulting in six strains belonging to the class Trebouxiophyceae, three to Chlorophyceae, two to Prymnesiophyceae and one to Cyanidiophyceae. This study reports for the first time the molecular identification of microalgal strains isolated from Costa Rica, highlighting the diversity of these microorganisms in the country.
Recibido: 2 de octubre de 2017; Revisión recibida: 6 de diciembre de 2017; Aceptado: 16 de marzo de 2018
RESUMEN
Las microalgas componen un diverso grupo polifilético de microorganismos fotosintéticos. Debido a su potencial biotecnológico, los estudios para aislar e identificar nuevas cepas han incrementado, por lo que es necesario el desarrollo de nuevas técnicas para su correcta identificación y clasificación. Utilizando herramientas de biología molecular, en este estudio se analizó el gen del ADNr 18S de 12 cepas microalgales aisladas de diferentes regiones de Costa Rica, resultando seis pertenecientes a la clase Trebouxiophyceae, tres a Chlorophyceae, dos a Prymnesiophyceae y una a Cyanidiophyceae. Este estudio reporta por primera vez la identificación molecular de cepas microalgales aisladas de Costa Rica, resaltando la diversidad de estos microorganismos en el país.
Palabras clave:
biotecnología microalgal, filogenia, identificación molecular.ABSTRACT
Microalgae integrate a diverse polyphyletic group of photosynthetic microorganisms. Due to its biotechnological potential, studies to isolate and identify new strains have increased, hence, it is necessary to develop new techniques for their correct identification and classification. Using molecular biology tools, this study analyzed the 18S rDNA gene from 12 microalgal strains isolated from different regions of Costa Rica, resulting in six strains belonging to the class Trebouxiophyceae, three to Chlorophyceae, two to Prymnesiophyceae and one to Cyanidiophyceae. This study reports for the first time the molecular identification of microalgal strains isolated from Costa Rica, highlighting the diversity of these microorganisms in the country.
Keywords:
microalgal biotechnology, molecular identification, phylogeny.Las microalgas componen un diverso grupo polifilético de microorganismos (eucariotas y procariotas) que se caracterizan por realizar fotosíntesis (Andersen, 2013). Recientemente, la amplia gama de biomoléculas que sintetizan (carbohidratos, lípidos, proteínas y pigmentos) las ha convertido en organismos comercialmente atractivos (Bux, 2013). Se estima que se han descrito alrededor de 44000 especies de microalgas a nivel mundial, aisladas de diversos ambientes como agua dulce, agua marina y fuentes hidrotermales (Barsanti y Gualtieri, 2014). Costa Rica posee numerosos hábitats para el crecimiento microalgas, sin embargo, los grupos taxonómicos de microalgas nunca han sido examinados en aguas costarricenses y sólo se dispone de observaciones anecdóticas ocasionales (Werthmann, 2009). Uno de los primeros reportes realizado por Silva-Benavides et al., (2008) describió e identificó 4 géneros y 52 especies aisladas de 17 muestras de ríos de Costa Rica utilizando microscopio óptico, evidenciando una alta diversidad de especies microalgales. A medida que los estudios en biodiversidad microalgal continúan y las colecciones de especies de microalgas se tornan más amplias, la correcta identificación y clasificación de microalgas se vuelve una tarea más difícil (Soylu y Gõnülol, 2012), especialmente cuando la caracterización es basada en características morfológicas, debido a la presencia de especies crípticas y la plasticidad fenotípica que presentan algunas microalgas (Hoshina y Fujiwara, 2013). La identificación molecular de microalgas utilizando secuencias de ADN podría proporcionar los medios para identificarlas de forma consistente y rápida, independientemente de la etapa de la vida y fenotipo (Hadi et al., 2016). Recientemente se han desarrollado protocolos eficaces para la identificación de cepas de microalgas utilizando varios marcadores moleculares, entre los que destacan los genes de la subunidad mayor de la ribulosa-1,5-bisfosfato carboxilasa/oxigenasa (rbcL), el espaciador interno transcrito 2 (ITS-2) y el gen del ARNr 18S (Radha et al., 2013). En el presente estudio, se revisó la clasificación de algunas cepas de microalgas de la colección del Centro de Investigación en Biotecnología del Instituto Tecnológico de Costa Rica (CIB-ITCR) utilizando datos de la secuencia de ADN codificante para la subunidad pequeña del ARN ribosomal (SSU rDNA) y se reportó las primeras anotaciones moleculares sobre la diversidad de microalgas de Costa Rica.
Se seleccionaron 12 cepas microalgales, correspondientes a una fracción de la colección de microalgas del CIB-ITCR, las cuales fueron aisladas y purificadas a partir de diferentes ambientes acuáticos como agua dulce, agua marina, aguas residuales y manantiales hidrotermales y representan distintas regiones del país (Tabla 1), siguiendo lo establecido por la Ley de Biodiversidad de Costa Rica y contando con los permisos de colecta e investigación de la entidad a cargo. Con una muestra de cultivo de 50 mL de cada cepa se realizó extracción de ADN con el kit MagJET Plant Genomic DNA Kit (Thermo Fisher Scientific, EE.UU). La identificación taxonómica de las cepas se realizó mediante la amplificación por PCR y secuenciación de la región 18S del ADNr. La reacción de PCR se llevó a cabo con los iniciadores NS1 (GTAGTCATATGCTTGTCTC) y NS8 (TCCGCAGGTTCACCTACGGA) (Liu, 2011), en un volumen total de 25 ML conteniendo 0,2 mM dNTPs, 0,2 mM de cada iniciador, 2,0 mM MgCl2 , 0,5 U de DreamTaq polimerasa (Life Technologies) y 5 mL de ADN en un termociclador Veriti® Thermal Cycler (Applied Biosystems) con el perfil térmico: 10 min desnaturalización a 95 °C, 35 ciclos de amplificación a 95 °C por 30 seg, 55 °C por 30 seg, 72 °C por 1 min 30 seg y una extensión final por 10 min a 72 °C. Con el fin de confirmar la naturaleza de los amplicones, se utilizó secuenciación en ambas direcciones con la metodología de Big Dye Terminator Cycle (Applied Biosystems, EE. UU.), en un secuenciador ABI Prism 3730XL (PE Applied Biosystems) de la compañía Macrogen (Corea del Sur).
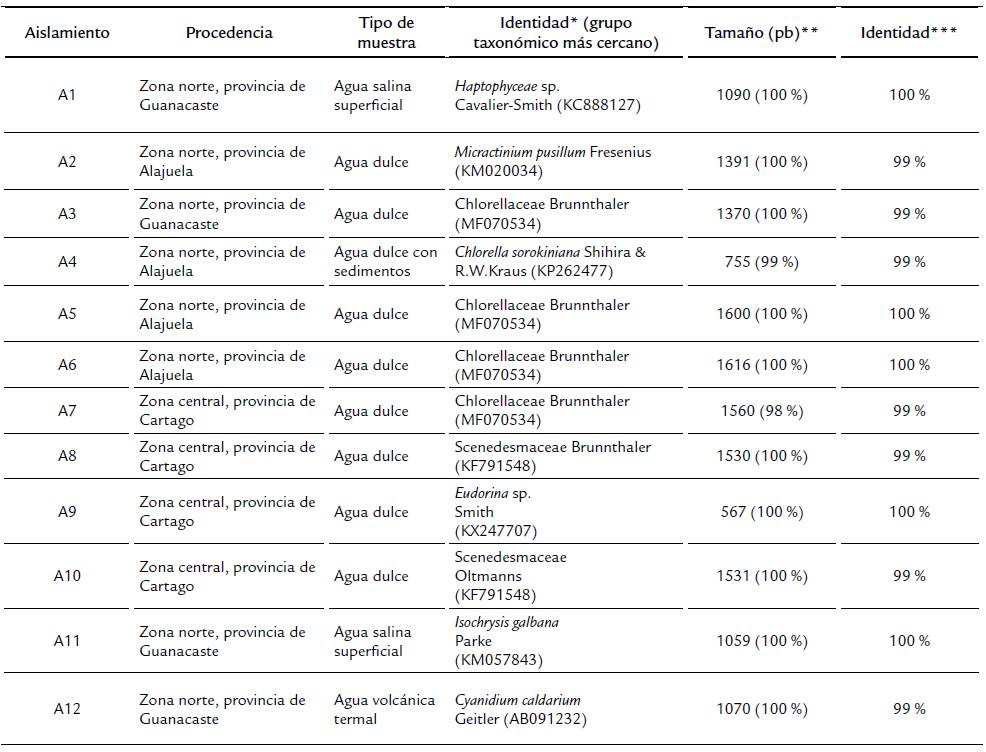
* Entre paréntesis se presenta el código GenBank de la secuencia **Entre paréntesis se presenta el porcentaje de cobertura. ***Valor (e) para todas las comparaciones: 0,0.Tabla 1: Descripción de doce cepas aisladas, purificadas y almacenadas en una colección de microalgas del CIB-ITCR, así como la identidad obtenida mediante comparación BLASTN de sus secuencias ADNr 18S.
Las secuencias obtenidas con cada cebador fueron analizadas según (Alvarez et al., 2013), se editaron y ensamblaron mediante la construcción de secuencias consenso utilizando la herramienta ClustalW Multiple Alignment (parámetros por defecto) y Create Cosensus Sequence del software BioEdit 7.2.6.1 (Hall, 1999). Se confirmó la identidad por comparación de la secuencia consenso obtenida de cada cepa con la base de datos "Nucleotide collection (nr/nt)" del NCBI mediante BLASTn (Altschul et al., 1990), utilizando los parámetros de búsqueda por defecto con un threshold e-value de 1e-3 y optimizado para "Highly similar sequences (megablast)".
Las secuencias consenso obtenidas fueron empleadas para realizar análisis filogenéticos basados en el algoritmo de Máxima verosimilitud utilizando un modelo de sustitución tipo nucleótido con cálculo de las distancias genéticas por el método de Kimura 2-parámetros, deleción completa para tratamiento de gaps y NNI (Nearest-Neighbor-Interchange) como método de búsqueda ML, utilizando el software Mega 7.0 (Kumar et al., 2016). El soporte de la topología interna del árbol filogenético se realizó mediante análisis de bootstrap con 1000 iteraciones (Vanega-Berrouet et al., 2014). Adicionalmente, se realizó una red de haplotipos mediante el software PopART (Leigh y Bryant, 2015), utilizando como método de inferencia la Red de Expansión Mínima (MSN).
El análisis por BLASTn para las secuencias de las microalgas correspondió a un alineamiento local que condujo a identificaciones putativas, de esta manera fue posible asociar consistentemente género y especie para cuatro cepas (A2, A4, A11 y A12), solamente género para dos cepas (A1 y A9) y las restantes seis pudieron ser clasificadas a nivel de clase (A3, A5, A6, A7, A8 y A10) (Tabla 1, Fig. 1a). Por esta razón, con el fin de tener una mayor confiabilidad en la identificación se realizó un análisis filogenético de máxima verosimilitud con las secuencias obtenidas en este estudio, de esta manera, fue posible la inferencia taxonómica mediante el agrupamiento de las cepas en clados a nivel de clase. El dendrograma resultante (Fig. 1b) se dividió en dos clados principales (I y II). El primer clado se subdivió a su vez en dos subclados (IA y IB), representando las clases Trebouxiophyceae y Chlorophyceae de la división Chlorophyta (algas verdes).
El subclado IA representaría a dos especies de microalgas diferentes; A6, A7, A5, A2 y A3 se agrupan dentro de una misma especie o nodo con valores de bootstrap de 99 %; mientras que A4 representaría una especie diferente. La clase Trebouxiophyceae es rica en especies de microalgas de vida libre, los miembros de este grupo exhiben notables variaciones morfológicas y habitan diversos ambientes terrestres y acuáticos (Lemieux et al., 2014). Engloba además muchas microalgas verdes, esféricas, unicelulares y no móviles de agua dulce difíciles de clasificar mediante características morfológicas (Stenclová et al., 2017).
Los resultados de la identificación molecular concuerdan con las descripciones morfológicas de las cepas analizadas, ya que presentan una morfología unicelular esférica, no móvil, con un diámetro de 2-7 Mm; con una pared celular gruesa, pero que varía según su estado de desarrollo (Fig. 1a, A2-A7). En la micrografía es posible también observar diferentes fases de la formación de la pared celular de las células hijas (reproducción por autosporulación).
Figura 1: Cepas de microalgas de la colección del CIB-ITCR codificadas de A1 a A12. a) Micrografía que muestra la morfología de doce aislamientos analizados (Barra= 10um para todos los aislamientos). b) Arbol filogenético basado en secuencias de la región 18S del ADNr de cada cepa (los números en las ramas indican los valores de bootstrap). El análisis filogenético se obtuvo utilizando el método de máxima verosimilitud, se muestra el árbol con mayor log de verosimilitud (-2693.56), los árboles iniciales para la búsqueda heurística se obtuvieron automáticamente aplicando los algoritmos Neighbor-Join y BioNJ a una matriz de distancias por pares estimadas utilizando el enfoque MCL (Maximum Composite Likelihood) y luego seleccionando la topología con un valor de log verosimilitud superior. El árbol está dibujado a escala, con longitudes de rama medidas en el número de sustituciones por sitio. El análisis involucró 12 secuencias de nucleótidos y se eliminaron todas las posiciones que contenían gaps y datos faltantes. Hubo un total de 874 posiciones en el conjunto de datos final. c) Diagrama de análisis de Red de Expansión Mínima (Minimun Spanning Network) de las secuencias consenso. El tamaño de los círculos representa las frecuencias de cada uno de los haplotipos. Se interconectan de acuerdo a sus relaciones filogenéticas y la longitud de las ramas indica el número de mutaciones (entre paréntesis).
El subclado IB representaría a dos especies de microalgas, A8 yA10 se agrupan dentro de una misma especie o nodo con valores de bootstrap de 100 %; mientras que A9 representaría una especie diferente. Las especies de la clase Chlorophyceae son microalgas verdes móviles con uno o dos pares de flagelos, sin mastigonemas. Las células pueden estar desnudas o cubiertas por una pared celular calcificada en mayor (Fig. 1a-A8, A10) o menor grado (Fig. 1a-A9) (Barsanti y Gualtieri, 2014).
El segundo clado se subdivió a su vez en dos subclados (IIA y IIB) representando las clases Cyanidiophyceae y Prymnesiophyceae, del filo Rhodophyta y Haptophyta respectivamente. El subclado IIA representa solamente una especie de microalga de la clase Cyanidiophyceae, aislada de un manantial de agua termal, de igual manera consistente con las características de la clase, microalgas termoacidófilas unicelulares de color verde a verde azulado (Fig. 1a-A12), las cuales soportan pH entre los 0,05-5 y temperaturas entre 35-56 °C (Lowell y Castenholz, 2013). Finalmente, las dos cepas del subclado IIB se agrupan dentro de una misma especie con valores de bootstrap de 100 %, y corresponden a microalgas de la clase Prymnesiophyceae, lo cual es consistente con su fenotipo, ya que corresponden a microalgas aisladas de agua marina y presentan características morfológicas y fisiológicas de especies halófitas, como flagelo, pigmentos del tipo carotenoides (Fig. 1a-A11) y tolerancia a la salinidad (Lee, 2008).
Adicionalmente, la red de haplotipos reflejó un resultado concordante al obtenido en el árbol filogenético, distribuyendo las secuencias analizada en seis grupos taxonómicos diferentes. En la red, cada esfera representa un haplotipo y su diámetro la frecuencia, por tanto es posible derterminar que las cepas A2, A3, A5, A6 y A7 corresponden a un mismo haplotipo. Filogenéticamente, este haplotipo representa el mas ancestral, ya que tiene a mayor cantidad de conexiones y es el más abundante, lo cual concuerda en ambos análisis realizados (Fig. 1b y Fig. 1c).
Estos resultados vislumbran el potencial biotecnológico de la colección de cepas microalgales del CIB-ITCR. Por ejemplo, las microalgas de la clase Trebouxiophyceae han llamado la atención de la comunidad científica debido a su utilidad en diversas aplicaciones biotecnológicas, como el tratamiento de aguas residuales, la generación de biocombustibles, la producción de nutracéuticos y otras moléculas de alto valor económico (Safi et al., 2014), dentro de este grupo destacan las microalgas del género Chlorella spp. En cuanto a las microalgas marianas, como Isochrysis galbana, estas han sido utilizadas ampliamente en la acuicultura por ser ricas en ácidos grasos poliinsaturados (PUFAs), se producen comercialmente como alimento para los primeros estadios larvarios de moluscos, peces y crustáceos; además, son una valiosa fuente de proteínas, carbohidratos y pigmentos fotosintéticos como la clorofila a y fucoxantina (Gilbert-López et al., 2015). Por su parte, las microalgas termoacidófilas de la clase Cyanidiophyceae podrían proveer una nueva variedad de moléculas y enzimas termostables, ya que han desarrollado mecanismos de adaptación que les permite soportar condiciones extremas que las microalgas de agua dulce no podrían tolerar, un ejemplo de molécula atractiva es el pigmento ficocianina (Rahman et al., 2017).
Esta investigación evidencia y comprueba que existe una gran diversidad de especies de microalgas en distintos ambientes de Costa Rica, de las cuales 12 han sido exitosamente aisladas, purificadas, clasificadas taxonómicamente y almacenadas en una colección nacional de cepas microalgales. Es necesario, intensificar la búsqueda de nuevas cepas de microalgas en Costa Rica, de manera que se aumente la cantidad de especies disponibles en colecciones con potencial para aplicaciones industriales y biotecnológicas, con el fin de facilitar la explotación de este recurso en el país. Este alcance fomentaría además el cultivo de microalgas a gran escala como iniciativa de crecimiento científico-tecnológico ambientalmente favorable y acorde con los esfuerzos para la mitigación del cambio climático, debido al potencial para producción de energía, alimento y captura de dióxido de carbono de estos microorganismos (Greene et al., 2016).
Adicionalmente, es importante mejorar las metodologías para identificación molecular de microalgas, ya que nuestros resultados demuestran que aún el análisis de la secuencia ADNr 18S completa, no es suficiente para lograr definir la identidad específica de algunas cepas. Este es el caso particular de las familias Scenedesmaceae y Chlorellaceae, cuyos miembros presentan similitudes mayores al 99 % en la secuencia estudiada, independientemente de su género y especie, resultado que ha sido reportado por otros autores para algas verdes (Tragin et al., 2016, Temraleeva et al., 2017). Es indispensable encontrar nuevas secuencias genéticas variables en microalgas que puedan ser utilizadas en conjunto con otras regiones conocidas (ITS, ADNr18S, rbcL, etc) para mejorar la capacidad de discriminación y lograr una identificación molecular más precisa. Para alcanzar este propósito es imprescindible aumentar la cantidad de genomas microalgales en las bases de datos, con lo cual se logrará realizar mayor cantidad de comparaciones genéticas que permitan definir diferencias entre especies cercanas.
AGRADECIMIENTOS
A la Vicerrectoría de Investigación y Extensión del Instituto Tecnológico de Costa Rica por el financiamiento de esta investigación.
REFERENCIAS
Referencias
Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. J Mol Biol. 1990; 215(3):403-410. Doi:10.1016/S0022-2836(05)80360-2
Álvarez C, Osorio N, Marín M. Molecular identification of microorganisms associated to the rhizosphere of vanilla plants in Colombia. Acta Biol Col. 2013;18(2):293-305.
Andersen R. The Microalgal Cell. En: Richmond A, editor. Handbook of microalgal culture: biotechnology and applied phycology. 2 ed. Oxford: Wiley-Blackwell. 2013. p. 3-20. Doi:10.1002/9781118567166.ch1
Barsanti L, Gualtieri P. Algae: anatomy, biochemistry, and biotechnology. 2 ed. Boca Raton: CRC Press/Taylor and Francis Group; 2014. p. 325. Doi:10.1201/b16544
Vanega-Berrouet KM, Gutiérrez-Sánchez PA, Marín-Montoya MA. Identificación molecular de hongos aislados de tejidos de fríjol con síntomas de antracnosis. Acta Biol Col. 2014;19(2):143-153. Doi:10.15446/abc.v19n2.39154
Bux F. Biotechnological Applications of Microalgae. 1 ed. Boca Raton: CRC Press/Taylor and Francis Group; 2013. p. 201.
Gilbert-López B, Mendiola JA, Fontecha J, van den Broek LAM, Sijtsma L, Cifuentes A, et al. Downstream processing of Isochrysis galbana: a step towards microalgal biorefinery. Green Chem. 2015;17(9):4599-4609. Doi:10.1039/C5GC01256B
Greene CH, Huntley ME, Archibald I, Gerber LN, Sills DL, Granados J, et al. Marine Microalgae: climate, energy, and food security from the sea. Oceanography. 2016; 29(4):10-15. Doi:10.5670/oceanog.2016.91
Hadi S, Santana H, Brunale PPM, Gomes TG, Oliveira MD, Matthiensen A, et al. DNA barcoding green microalgae isolated from Neotropical inland waters. PloS one. 2016;11(2):e0149284. Doi:10.1371/journal.pone.0149284
Hall T. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl Acid S. 1999;41(1):95-98.
Hoshina R, Fujiwara Y. Molecular characterization of Chlorella cultures of the National Institute for Environmental Studies culture collection with description of Micractinium inermum sp. nov., Didymogenes sphaerica sp. nov., and Didymogenes soliella sp. nov. (Chlorellaceae, Trebouxiophyceae). Phycol Res. 2013;61(2):124-132. Doi:10.1111/pre.12010
Kumar S, Stecher G, Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets. Mol Biol Evol. 2016;33(7):1870-1874. Doi:10.1093/molbev/msw054
Lee RE. Phycology. 4 ed. Cambridge: Cambridge Univ. Press; 2008. p. 521. Doi:10.1017/CBO9780511812897.029
Leigh JW, Bryant D. Popart: full-feature software for haplotype network construction. Methods Ecol Evol. 2015;6(9):1110–1116. Doi:10.1111/2041-210X.12410
Lemieux C, Otis C, Turmel M. Chloroplast phylogenomic analysis resolves deep-level relationships within the green algal class Trebouxiophyceae. BMC Evol Biol. 2014;14:211. Doi:10.1186/s12862-014-0211-2
Liu D. Molecular detection of human fungal pathogens. Boca Raton: CRC Press/Taylor and Francis Group; 2011. p.856-857. Doi:10.1201/b11375
Lowell C, Castenholz RW. The lowering of external pH in confined environments by thermo-acidophilic algae (class: Cyanidiophyceae). Environ Microbiol Rep. 2013;5(5):660-664. Doi: 10.1007/s00338-014-1127-4
Radha S, Fathima A, Iyappan S, Ramya M. Direct colony PCR for rapid identification of varied microalgae from freshwater environment. J Appl Phycol. 2013;25(2):609-613. Doi:10.1007/s10811-012-9895-0
Rahman DY, Sarian FD, van Wijk A, Martinez-Garcia M, van der Maarel, MJEC. Thermostable phycocyanin from the red microalga Cyanidioschyzon merolae, a new natural blue food colorant. J Appl Phycol. 2017;29(3):1233-1239. Doi:10.1007/s10811-016-1007-0
Safi C, Zebib B, Merah O, Pontalier P, Vaca-Garcia C. Morphology, composition, production, processing and applications of Chlorella vulgaris: A review. Renew Sust Energ Rev. 2014;35:265-278. Doi:10.1016/j.rser.2014.04.007
Silva-Benavides A, Sili C, Torzillo G. Cyanoprocaryota y microalgas (Chlorophyceae y Bacillariophyceae) bentónicas dominantes en ríos de Costa Rica. Revi Biol Trop. 2008;56(4):221-235. Doi:10.15517/rbt.v56i4.27229
Soylu E, Gönülol A. Morphological and 18S rRNA analysis of coccoid green algae isolated from lakes of Kızılırmak Delta. Turk J Biol. 2012;36:247-254.
Štenclová L, Fučíková K, Kaštovský J, Pažoutová M. Molecular and morphological delimitation and generic classification of the family Oocystaceae (Trebouxiophyceae, Chlorophyta). J Phycol. 2017;53(6):1263-1282. Doi:10.1111/jpy.12581
Temraleeva AD, Moskalenko SV, Bachura YM. Morphology, ecology, and 18S rDNA phylogeny of the green microalgal order Protosiphonales (Chlorophyceae, Chlorophyta). Microbiology. 2017;86(2):159-169. Doi:10.1134/S0026261717020205
Tragin M, Lopes dos Santos A, Christen R, Vaulot D. Diversity and ecology of green microalgae in marine systems: an overview based on 18S rRNA gene sequences. Perspect Phycol 2016;3(3):141-154. Doi:10.1127/pip/2016/0059
Werthmann IS. Marine biodiversity of Costa Rica, Central America. New York: Science and business media; 2009. p. 583. Doi:10.1007/978-1-4020-8278-8
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
CrossRef Cited-by
1. Hongwei Ning, Rui Li, Teng Zhou. (2022). Machine learning for microalgae detection and utilization. Frontiers in Marine Science, 9 https://doi.org/10.3389/fmars.2022.947394.
Dimensions
PlumX
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2018 Acta Biológica Colombiana

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
1. La aceptación de manuscritos por parte de la revista implicará, además de su edición electrónica de acceso abierto bajo licencia Attribution-NonCommercial-ShareAlike 4.0 (CC BY NC SA), la inclusión y difusión del texto completo a través del repositorio institucional de la Universidad Nacional de Colombia y en todas aquellas bases de datos especializadas que el editor considere adecuadas para su indización con miras a incrementar la visibilidad de la revista.
2. Acta Biológica Colombiana permite a los autores archivar, descargar y compartir, la versión final publicada, así como las versiones pre-print y post-print incluyendo un encabezado con la referencia bibliográfica del articulo publicado.
3. Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
4. Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos institucionales, en su página web o en redes sociales cientificas como Academia, Researchgate; Mendelay) lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).