Variabilidad interspecífica de duraznos (Prunus pérsica L. Batsch.) y ciruelos (Prunus domestica) usando RAMs
Intraespecific variability of peach (Prunus persica L. Batsch.) and plums (Prunus domestica) using RAMs
DOI:
https://doi.org/10.15446/rev.colomb.biote.v17n1.44644Palabras clave:
Prunus, Diversidad genética, Microsatélites RAMs, caducifolios (es)Resumen: Se seleccionó una muestra de 41 materiales de Prunus de la colección de caducifolios de la Universidad Pedagógica y Tecnológica de Colombia, para evaluar su variabilidad genética usando ocho marcadores Microsatélites Amplificados al Azar (RAMs). Se generaron un total de 121 alelos con pesos moleculares entre 260 y 1000 Kb. Se formaron tres grupos, a un coeficiente de similitud de 0.75, de acuerdo a las características del fruto y a la especie, encontrándose en el grupo II a los materiales de ciruelo. El porcentaje de loci polimórficos varío entre 71 y 99% para los cebadores CGA y CCA, respectivamente. El valor promedio de heterocigosidad fue de 0.22, mucho más bajo que los encontrados en otros estudios de diversidad genética en el género Prunus. La técnica RAMs mostró ser una herramienta útil para evaluar la diversidad genética en frutales caducifolios, al discriminar a los materiales en tres grupos e identificar el alto grado de consanguinidad que existe entre las diferentes especies de Prunus lo cual debe ser aprovechado dentro de las estrategias de hibridación que busquen la obtención de nuevos y mejores materiales.
Palabras claves: Prunus, Diversidad genética, Microsatélites RAMs, caducifolios.
Key words: Prunus, Genetic diversity, Microsatellites RAMs, deciduous.
A sample of 41 Prunus materials from the deciduous collection of the Pedagogical and Technological University of Colombia was selected to evaluate its genetic diversity using eight primers for Random Amplified Microsatellite (RAMs). A total of 121 alleles were generated with molecular weights ranging between 260 and 1000 Kb. Three groups were formed, a similarity coefficient of 0.75, according to fruit characteristics and specie, found plum materials in group II. The percentage of polymorphic loci ranged from 71 to 99% for the primers CGA and CCA, respectively. The average value of heterozygosity was 0.33, much lower than values found in other genetic diversity studies in the genus Prunus. The RAMs technique showed to be a useful tool for assessing genetic diversity in deciduous fruit, discriminate the materials into three groups and identify the high degree of consanguinity between different Prunus species which should be exploited in hybridization strategies looking for obtaining new and improved materials.
Key words: Prunus, Genetic diversity, Microsatellites RAMs, deciduous.
DOI: https://doi.org/10.15446/rev.colomb.biote.v17n1.44644
ARTÍCULO DE INVESTIGACIÓN
Variabilidad interspecífica de duraznos (Prunus pérsica L. Batsch.) y ciruelos (Prunus domestica) usando RAMs
Intraespecific variability of peach (Prunus persica L. Batsch.) and plums (Prunus domestica) using RAMs
Ana Cruz Morillo C.*, Yacenia Morillo C.**, Leonardo González M.***, Iván Adiel A.****
* Ing. Agrónomo. PhD en Ciencias Agropecuarias con énfasis en Fitomejoramiento. Docente Universidad Pedagógica y Tecnológica de Colombia, Tunja (Boyacá, Colombia), balmoregc@gmail.com.
** Ing. Agrónomo. PhD en Ciencias Agropecuarias con énfasis en Fitomejoramiento. Docente Universidad de los Llanos, Villavicencio (Meta, Colombia), ymorillo@unillanos.edu.co
*** Estudiante de Maestría en Fisiología Vegetal de la Universidad Pedagógica y Tecnológica de Colombia, Tunja (Boyacá, Colombia), Leonardo.gonzalez@uptc.edu.co.
**** Ingeniero agrónomo, Universidad de los Llanos, ivanavilamorales@hotmail.com. Autor para correspondencia: ana.morillo@uptc.edu.co.
Recibido: septiembre 18 de 2014 Aprobado: abril 27 de 2015
Resumen
Se seleccionó una muestra de 41 materiales de Prunus de la colección de caducifolios de la Universidad Pedagógica y Tecnológica de Colombia, para evaluar su variabilidad genética usando ocho marcadores Microsatélites Amplificados al Azar (RAMs). Se generaron un total de 121 alelos con pesos moleculares entre 260 y 1000 Kb. Se formaron tres grupos, a un coeficiente de similitud de 0.75, de acuerdo a las características del fruto y a la especie, encontrándose en el grupo II a los materiales de ciruelo. El porcentaje de loci polimórficos varío entre 71 y 99% para los cebadores CGA y CCA, respectivamente. El valor promedio de heterocigosidad fue de 0.22, mucho más bajo que los encontrados en otros estudios de diversidad genética en el género Prunus. La técnica RAMs mostró ser una herramienta útil para evaluar la diversidad genética en frutales caducifolios, al discriminar a los materiales en tres grupos e identificar el alto grado de consanguinidad que existe entre las diferentes especies de Prunus lo cual debe ser aprovechado dentro de las estrategias de hibridación que busquen la obtención de nuevos y mejores materiales.
Palabras claves: Prunus, Diversidad genética, Microsatélites RAMs, caducifolios
Abstract
A sample of 41 Prunus materials from the deciduous collection of the Pedagogical and Technological University of Colombia was selected to evaluate its genetic diversity using eight primers for Random Amplified Microsatellite (RAMs). A total of 121 alleles were generated with molecular weights ranging between 260 and 1000 Kb. Three groups were formed, a similarity coefficient of 0.75, according to fruit characteristics and specie, found plum materials in group II. The percentage of polymorphic loci ranged from 71 to 99% for the primers CGA and CCA, respectively. The average value of heterozygosity was 0.33, much lower than values found in other genetic diversity studies in the genus Prunus. The RAMs technique showed to be a useful tool for assessing genetic diversity in deciduous fruit, discriminate the materials into three groups and identify the high degree of consanguinity between different Prunus species which should be exploited in hybridization strategies looking for obtaining new and improved materials.
Key words: Prunus, Genetic diversity, Microsatellites RAMs, deciduous.
Introducción
La familia Rosaceae, comprende más de 3000 especies, muchas de las cuales son frutales de importancia económica el albaricoquero (P. armeniaca L.), el almendro (P. dulcis Mill), el cerezo (P. avium L.), y el ciruelo europeo (P. domestica L.) y japonés (P. salicina L.). (Fan et al., 2013). La producción anual de las especies más importantes de Prunus fue para manzanas 76.378.738 (Ton), peras 23.580.845 (Ton), duraznos y nectarinas 21.083.151 (Ton), albaricoque 3.956.639 (Ton), cerezas 2.256.519 (Ton), y ciruelos 10.702.744 (Ton) (FAOSTAT, 2014). En el género Prunus, además del durazno (Prunus persicae (L.) Batsch), también llamado melocotonero, existen otras cuatro especies distintas de melocotonero, P. davidiana (Carr.) Franch., P. mira Koehne, P. kansuensis Rehd. Y P. ferganensis, pero solo la especie P. persica se cultiva por su fruta (Hancock et al., 2008). Al igual que el ciruelo (Prunus domestica), constituyen las especies caducifolias más populares que se cultivan en las zonas templadas de todo el mundo al igual que el durazno, son un grupo diverso de plantas con muchas especies adaptadas a un amplio rango de condiciones ecogeográficas y que han sido cultivadas por siglos (Okie y Hancock, 2008).
La identificación de cultivares y el estudio de relaciones genéticas en estas especies tradicionalmente se basó en características morfológicas, sin embargo en los programas de mejoramiento de Prunus, su evaluación es un proceso tedioso debido al período juvenil tan largo, la influencia del estado fenológico en la expresión de las características y la existencia de factores climáticos que afectan su expresión, por estas razones la selección asistida por marcadores moleculares es particularmente útil en estos casos (Ercisli et al., 2011). En Prunus, los microsatélites (SSRs) han sido los marcadores más usados para la caracterización de germoplasma (Guarino et al., 2009), determinación de la diversidad genética (Rogatis et al., 2013), manejo de germoplasma (Kamm et al., 2011), identificación de cultivares y fingerprinting (Gulen et al., 2010), determinación de genes de interés (Bouvier et al., 2012) estudios de autoincompatibilidad y poblacional (Mariette et al., 2010) y mapeo genético (Dhanapal et al., 2012). En Prunus, los microsatélites desarrollados en una especie han sido usados en diferentes especies, demostrando su transferibilidad y habilidad para detectar polimorfismos (Fan et al., 2013).
Zietkiewick et al. (1994), propusieron un método para medir la diversidad genética en plantas y animales usando cebadores basados en microsatélites. Esta técnica combina los beneficios de los microsatélites y los RAPD (Random Amplified Polymorphic DNA). Hantula et al. (1996) seleccionaron cuatro cebadores (GT, ACA, CCA y CGA) con una longitud de 18 bases que incluyen un extremo 5´"degenerado" de tres nucleótidos el cual sirve de anclaje para asegurar la unión del cebador al inicio del microsatélite. Ellos denominaron a esta técnica RAM (Random Amplified Polymorphic DNA). Los RAMs se basan en la reacción en cadena de la polimerasa (PCR), el método es altamente reproducible y permite la detección de polimorfismos en el ADN intra e interespecífico (Muñoz et al., 2008).
Con los marcadores RAMs se estudian poblaciones, se mide la diversidad genética en plantas y animales, se muestra la base de la variación de los individuos, se permite seleccionar regiones del genoma, analizar la información que se expresa ya que el número de polimorfismos detectables es teóricamente ilimitado (Mahuku et al., 2002). La metodología se ajusta para pequeños laboratorios en términos de equipos y facilidades de costo, no requiere conocimiento previo de secuencias (Muñoz et al., 2008).
Trabajos sobre caracterización de diversidad genética vegetal sugieren que la técnica RAMs es útil para identificar duplicados en bancos de germoplasma, para establecer grupos o asociaciones entre individuos de una población de acuerdo con la especie, localización geográfica, relaciones filogenéticas, entre otras (Muñoz et al., 2008). Ampliamente utilizado en frutales como la mora (Rubus glaucus) en donde diferenció las especies R. glaucus, R. robustus y R. urticifolius, detectó duplicados y encontró alta variabilidad genética en R. glaucus, la especie más importante (Morillo et al., 2005); en uchuva estos marcadores encontraron alta diversidad y dos accesiones de fruto rojo se diferenciaron genéticamente de las amarillas (Bonilla et al., 2008). En guayaba estos cebadores fueron altamente polimórficos y se encontró alta variabilidad en el Valle del Cauca (Sanabria et al.., 2006); en mandarina permitió agrupar las accesiones según los tipos mandarino Satsuma, mandarinos Clementinos e híbridos, por lo cual es una herramienta útil para evaluar la diversidad genética en el género Citrus (Morillo et al., 2009; Mora et al., 2013).
Morillo et al. (2014), utilizando ocho cebadores Microsatélites Amplificados al Azar (RAMs) evaluaron la diversidad genética de 31 materiales de Prunus de la colección de caducifolios de la Universidad Pedagógica y Tecnológica de Colombia, con un coeficiente de similaridad de 0.75 se formaron tres grupos de acuerdo principalmente con las características del fruto, con un valor de heterocigosidad de 0.22. La técnica RAMs fue útil para evaluar la diversidad genética en especies caducifolias.
Dentro de este contexto, este trabajo de investigación tuvo como objetivo principal hacer un análisis de la variabilidad interespecífica con marcadores microsatélites RAMs de los materiales de durazno y ciruelo, de la colección de caducifolios que tiene la Universidad Pedagógica y Tecnológica de Colombia, como una aproximación hacia el planteamiento de estrategias de hibridación e identificación de materiales élite.
Materiales y métodos
Se evaluaron un total de 28 accesiones de durazno, 11 de ciruelo, 2 de albaricoque (Prunus armeniaca) y un almendro (Prunus dulcis) pertenecientes a la colección de caducifolios de la Universidad Pedagógica y Tecnológica de Tunja, establecido en la granja experimental de Tunguavita, Paipa localizada a 36 Km de la ciudad de Tunja con una latitud Norte de 5° 47´04¨ y longitud Oeste de 73°07´17¨ con una temperatura promedia de 15°C (tabla 1).
Caracterización molecular
La caracterización molecular se hizo en los laboratorios de investigación en Biología Molecular, BIOPLASMA y GEBIMOL, de la Universidad Pedagógica de Colombia, Tunja. Para la extracción de ADN se utilizó el protocolo de Dellaporta et al. (1983). Los ADN totales se visualizaron en geles de agarosa al 0.8%, en una cámara Maxicell Primo EC-340 Electroforesis Gel System. Para determinar la concentración de ADN se utilizó el fluorómetro Hoefer Dyna Quant 200 y se diluyó en agua tipo HPLC a un volumen total de 100 μl a 10 ng/μl y se almacenó a -20 °C. Para el análisis RAMs se utilizaron ocho cebadores sintetizados por Technologies Inc. Bioneer (tabla 2). Para la reacción de amplificación con RAMs se preparó el cóctel en un tubo estéril de microcentrífuga (1.5 ml) para un volumen final de 25 μl. La mezcla de reacción se preparó con buffer 1X, MgCl2 1.5 mM, dNTPs 0.2 mM, Taq Polimerasa 1U, cebador 2 μM y ADN genómico 10ng.
Las siguientes designaciones se usan para los sitios degenerados: H (A ó T ó C); B (G ó T ó C); V (G ó A ó C) y D (G ó A ó T).
La amplificación se llevó a cabo en un termociclador PTC 100 Programmable Termal Controller (MJ. Research, Inc). La desnaturalización inicial fue a 95 °C durante 5 min; desnaturalización a 95 °C por 30 seg, hibridación a una temperatura de 50 °C (cebador AG ACA y CA), 55 °C (cebador CCA-TG-CT) y 58 °C (cebador GT-CGA) durante 45 seg, una extensión de 72 °C por 2 min, 37 ciclos desde la desnaturalización a extensión y por último una extensión a 72 °C durante 7 min. Los productos amplificados fueron separados por electroforesis en geles de poliacrilamida 37:1 (acrilamida: bisacrilamida) al 7 % a 150 v por 1 h en una cámara pequeña de DNA Sequencing System, FB-SEQ-3545 de Fisher Biotechnologies. La tinción se realizó usando sales de plata siguiendo el protocolo de Cres et al. (2001).
Análisis estadístico
Se generó una matriz binaria de presencia (uno) y ausencia (cero). Para la selección de bandas polimórficas se consideró como locus polimórfico aquel en el cual la frecuencia del alelo más común fue menor que el 95%. La similitud genética entre los individuos se calculó utilizando el coeficiente de similitud de Nei y Li (1979). El análisis de agrupamiento se realizó con el programa SAHN de NTSYS-pc (versión 2.02g, 98) utilizando el UPGMA, método gráfico de agrupamiento por parejas que usa el promedio aritmético no ponderado. El dendrograma se construyó con el programa TREE de NTSYS-pc (versión 2.02g, 98). Para evaluar la diversidad genética se estimó la heterocigosidad insesgada y el porcentaje de loci polimórficos utilizando el paquete estadístico TFPGA (Tools For Population Genetic Analices, versión 1.3, 1997). Se utilizó el paquete estadístico AMOVA®, por medio del programa AMOVA PREP para llevar a cabo el análisis de varianza molecular (AMOVA) y así poder determinar las diferencias entre y dentro de los grupos formados. Se determinó el f estadístico insesgado con un intervalo de confianza del 95 %.
Resultados y Discusión
El análisis mediante el coeficiente de Nei-Li, a un nivel de similitud de 0.75, diferenció a los materiales en 3 grupos (figura 1).
Los duraznos se clasifican por la presencia de tricomas en la piel y por la adherencia de la pulpa al hueso en duraznos propiamente dichos (Prunuspérsica L.) son aquellos que tienen la piel aterciopelada y la pulpa unida al hueso; el durazno-melocotón (Prunus pérsica l.) es de piel aterciopelada, pero la pulpa no está unida al hueso, se llaman duraznos abridores o priscos y los duraznos-nectarinos (Prunus pérsica var. Nectarino) es de piel lisa y la pulpa no unida al hueso, se les denomina duraznos calvos (Campos et al., 2013). Teniendo en cuenta esta clasificación en el Grupo I, se encuentran las variedades de durazno propiamente dichas como el Conservero, de pulpa amarilla con coloración rojiza alrededor del hueso, tamaño mediano fue introducido del Ecuador y el Camuezo, de pulpa amarilla, producción prolífica, se utiliza como patrón para todas las variedades de durazno y algunas de ciruelo. El Jarillo es una variedad que se importa de Venezuela, de piel y pulpa amarilla, en muchos viveros se usa como patrón (Campos et al., 2013).
A nivel mundial, existen más de 15,000 variedades de durazno (Castro et al., 1998). En Colombia se destacan las variedades fresa morado, Pezón de Venus, Fresa blanco, Elberta gigante, Rubidoux, Frank, Ventura, Diamante, Riograndense, Dorado, Conservero amarillo y Zapallo (Ordoñez, 2007), algunas de las cuales se encuentran dentro de este grupo.
La mayor parte de los ciruelos evaluados en este estudio se encuentran conformando el grupo II. De acuerdo con el color de la piel del fruto, se han clasificado en amarillos como la variedad Ecuatoriana, que tiene bajo contenido de pruina y se propaga vegetativamente, para obtener patrones e injertar variedades de ciruelo como Kelsey, Sangre Toro, Duarte y Beauty, que no son compatibles con el durazno común, que es el patrón usado comúnmente; y rojos como las variedades Beauty, con pruina, forma acorazonada, comercialmente es la más abundante en el mercado, aunque es muy susceptible a la manipulación poscosecha; Methley, de pulpa roja con sabor dulce, muy prolífica y de producción temprana, a pesar de su tamaño no tiene problemas de mercado; Horvin, piel roja con pruina, prolífica y de cosecha temprana, se puede llegar a obtener dos cosechas por año, principalmente en zonas de régimen bimodal de lluvias (Campos et al., 2013).
El patrón más utilizado en la injertación de ciruelo es el Durazno Blanco Común, sin embargo, presenta incompatibilidades con algunas variedades de ciruelo. El Flor Blanca también se utiliza pero con los mismos problemas anteriores. Por lo cual se reemplazan con ciruelos Ecuatoriano, Rojo Japonés o mirabolano, éste último se caracteriza por ser rústico, de fruta de color rojo, pequeña y hoja pequeña sin ningún valor comercial con fruta fresca. El almendro es una especie que presenta buena compatibilidad en la injertación con las diferentes variedades de ciruelo, por lo cual se encuentra haciendo parte de este grupo (Campos et al., 2013).
En el grupo III, encontramos entre las variedades más importantes de durazno a la variedad Florida que se ha adaptado a las condiciones del Cauca, y es sembrada a los 2400 m.s.n.m en los municipios de Silvia y Sotará. Sus frutos son de buena calidad, muy jugosos de color rojo y pulpa amarilla, la producción empieza a los tres años después de la siembra y puede producir durante 20 años (Serna y Oliva, 1999). Las variedades Dorado y Diamante se caracterizan por tener una piel amarilla con ligera pigmentación roja, pulpa amarilla oro con coloración rojiza alrededor del hueso. La primera presenta buenas características para la industrialización, y por su buen contenido de azúcar, es excelente para el consumo en fresco y la segunda se caracteriza porque la planta es muy vigorosa y productiva. La variedad Oro con un color amarillo atractivo presenta buenas propiedades organolépticas (Baiza, 2004).
Dentro de las variedades de durazno-melocotón, tenemos a la Rubidoux, por ser resistente a la manipulación poscosecha, tener una pulpa amarilla con coloración rojiza y una forma ligeramente redondeada; y al Rey Negro de piel amarilla con manchas rojas opacas, presencia de un pezón muy pronunciado, el cual le trae problemas en la manipulación poscosecha (Campos, 2013). El albaricoque canino, fue material que presentó menor similitud con el resto de los materiales evaluados, aunque se ha reportado mayor afinidad con otras especies del género Prunus (Gallego, 2010).
En términos generales, los agrupamientos de los materiales de duraznos evaluados, con los marcadores RAMs correspondieron de acuerdo a la especie evaluada, y a las características del fruto al igual que en otros estudios de diversidad genética usando marcadores microsatélites en Prunus (Stanys et al., 2008). Sin embargo también se observa el alto grado de similitud entre especies como el durazno y el ciruelo y de este último con el albaricoque, lo cual puede ser útil para el establecimiento de estrategias de hibridación dentro de un programa de mejoramiento genético de Prunus.
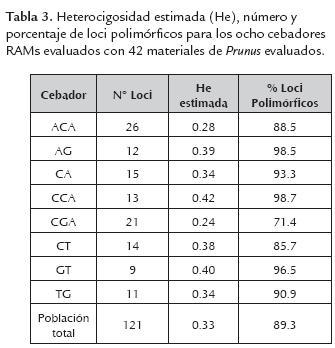
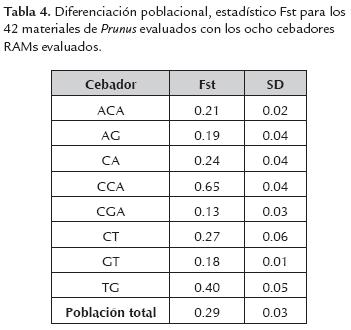
Los ocho cebadores RAMs utilizados para la caracterización molecular en durazno, generaron un total de 121 alelos que fluctuaron entre 9 para el cebador GT y 26 para el cebador ACA, con pesos moleculares entre 260 y 1000 kb. El porcentaje de loci polimórficos varió entre 71 a 98.7% para los cebadores CGA y CCA, respectivamente (tabla 3). El número de alelos se considera adecuado para estimar parámetros genéticos al compararlo con los resultados obtenidos en otras especies de Prunus (Stanys et al., 2012; Fernández et al., 2009). El cebador CCA fue el que mayor aporte hizo a la variación genética observada, Fst de 0.65 (tabla 4) lo que significa que puede ser útil para lograr mayor diferenciación entre los materiales del género Prunus.
Los valores de heterocigosidad estuvieron comprendidos entre 0.24 para el cebador CGA y 0.42 para el CCA. El valor promedio de heterocigosidad para todos los materiales evaluados en general fue de 0.33 mucho más bajo que los encontrados en estudios de diversidad genética usando SSRs realizados por, Carrasco et al. (2012) en 29 cultivares élites de ciruelo Japonés (Prunus salicina) y 4 cultivares de Prunus (0.80); Barac et al. (2014) en 35 accesiones de Prunus cerasus (0.39); Fon et al. (2013) en 94 cultivares de durazno y nectarinas (0.45). Sin embargo fue más alto que los valores encontrados por Barac et al. (2014) al evaluar 7 accesiones de P.mahaleb (0.28), 22 de P. avium (0.31), 4 de P. fruticosa (0.31), 4 de P. serrulata (0.28) y 4 de P. persica (0.19).
Los bajos niveles de heterocigosidad encontrados se atribuyen a los sistemas de apareamiento ya que la mayoría de los cultivares de durazno son autocompatibles lo que favorece los procesos de autofecundación (Sassa et al., 1992). Bajos niveles de polimorfismos también se han encontrado en materiales de cereza dulce (Boskovic y Tobutt, 1997). Esto probablemente refleja una estrecha base genética en el germoplasma de ciruela dulce y de durazno. Sin embargo concuerda con las afirmaciones de Imbert y Lefèvre (2003) quienes consideran que un exceso de heterocigosidad no es muy usual en especies arbóreas y que generalmente esta se ve favorecida por la autoincompatibilidad y por los cruzamientos interespecíficos (Stoeckel et al., 2006). La estrecha base genética utilizada para el desarrollo de cultivares y la existencia de ancestros comunes en los pedigree de duraznos y ciruelas, podrían explicar los resultados encontrados (Okie y Hancock, 2008). En general los bajos niveles de variabilidad son consistentes con los obtenidos usando diferentes marcadores moleculares en especies del género Prunus (Aranzana et al., 2010).
El coeficiente de diferenciación genética (Fst) obtenido al evaluar los 42 materiales de Prunus, con los ocho marcadores RAMs fue de 0.29, con una desviación estándar de 0.03 (tabla 4). Según Wright (1978), valores de 0.25 muestran gran diferenciación genética, lo cual puede estar reflejado en el alto grado de domesticación que han sufrido estos materiales, ya que la mayoría de ellos corresponden a variedades comerciales. Este valor es más alto que lo encontrado por Carrasco et al. (2012) en ciruelo (-0.13) y mucho más alto que en otras especies de polinización cruzada como cereza (0.092; Stoeckel et al., 2006), almendro (0.15; Fathi et al., 2008) y albaricoque (0.04; Bourguiba et al., 2010). Lo anterior muestra que las variedades de durazno y ciruelo son genéticamente menos diversas, contrario a los resultados obtenidos por Fon et al. (2013), esto se puede explicar teniendo en cuenta la historia evolutiva de las diferentes especies en su proceso de domesticación y su sistema de propagación clonal (McKey et al., 2010), pero también debido a que el durazno es la especie menos polimórfica del género Prunus por su condición de autogamia (Font et al., 2014).
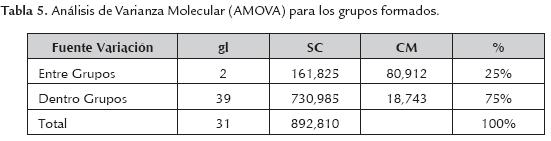
Los marcadores RAMs discriminaron a los materiales en tres grupos a unas distancias genéticas muy reducidas (0.75) mostrando así el grado consanguinidad que existe entre ellos. Carrasco et al. (2012) encontró valores de similitud comprendidos entre 0.57 a 0.99 al caracterizar con marcadores SSR e ISSR 29 cultivares de ciruelo Japonés (Prunus salicina) y 4 de durazno. También se han encontrado alto valores de similitud entre y dentro de las diferentes especies del género Prunus (Yoon et al., 2006; Fernández et al., 2009). El Análisis de Varianza Molecular (AMOVA) para los tres grupos mostró que la variación se encuentra entre los grupos, que correspondió a una varianza de 75% (tabla 5), el 25% restante se debió al componente varianza genética dentro de los grupos.
La similitud existente entre los diferentes materiales evaluados soporta la idea que existe una alta capacidad para la hibridación y el flujo genético entre las especies de Prunus evaluadas. La estrecha base genética usada para el desarrollo de variedades podría explicar estos resultados, pues los materiales estudiados pueden tener un ancestro común entre su pedigree (Okie y Hancock, 2008).
El alto grado de sintenia que se presenta entre los genomas de durazno y ciruelo, como se pudo observar en este estudio, puede ser aprovechado para producción de híbridos ya que hay estudios que muestran como estas dos especies son utilizadas para la generación de nuevos y mejores materiales dentro de estrategias de mejoramiento del género Prunus (Frecon, 2006; Reales et al., 2010). El uso de marcadores moleculares, el análisis de secuencias (Scalabrin et al., 2013; Vicedomini et al., 2013) permitirán un estudio más detallado del genoma de las especies de Prunus y al vez serán una herramienta útil para complementar y hacer más eficientes las estrategias de hibridación entre las especies del género Prunus.
Conclusión
La caracterización molecular con marcadores RAMs de la colección de duraznos y ciruelos de la Universidad Pedagógica y Tecnológica de Colombia, mostro que hay un alto grado de similitud entre los materiales evaluados, por lo cual la diversidad fue baja, sin embargo, esto muestra el alto grado de consanguinidad de las especies evaluadas y que puede ser usada eficientemente dentro de esquemas de hibridación que conduzcan hacia la obtención de nuevos y mejores materiales de Prunus.
Agradecimientos
Los autores agradecen la colaboración al grupo de investigación CIDE, a los laboratorios de investigación BIOPLASMA y GEBIMOL, a la Granja Experimental de Tunguavita y la Dirección de Investigaciones, DIN, de la Universidad Pedagógica y Tecnológica de Tunja.
Referencias bibliográficas
Aranzana, M. J., Abbassi, E. K., Howad, W., & Arus, P. (2010). Genetic variation, population structure and linkage disequilibrium in peach commercial varieties. BMC Genetics, 11(1), 69-79.
Baíza, V. (2004). Guía técnica del cultivo del melocotón. Programa Nacional de Frutas del Salvador. 46 p.
Barac, G., Ognjanov, V., Obreht, D., Ljubojevic, M., Bosnjakovic, D., Pejic, I., & Gasic, K. (2014). Genotypic and phenotypic diversity of cherry species collected in Serbia. Plant Molecular Biology Reporter, 32(1), 92-108.
Bonilla, M., Espinosa, K., Posso, A., & Muñoz, J. (2008). Caracterización molecular de 43 accesiones de uchuva de seis departamentos de Colombia. Acta Agronómica, 57 (2), 109-115.
Boskovic, R., Russell, K., & Tobutt, K. (1997). Inheritance of stylar ribonucleases in cherry progenies, and reassignment of incompatibility alleles to two incompatibility groups. Euphytica, 95(2), 221-228.
Bourguiba, H., Krichen, L., Audergon, J., Khadari, B., & Farah, N. (2010). Impact of mapped SSR markers on the genetic diversity of apricot (Prunus armeniaca L.) in Tunisia. Plant Molecular Biology Reporter, 28(4), 578-587.
Bouvier, L., Bourcy, M., Boulay, M., Tellier, M., Guérif, P., Denancé, C., Durel, C., & Lespinasse, Y. (2012). A new pear scab resistance gene Rvp1 from the European pear cultivar 'Navara' maps in a genomic region syntenic to an apple scab resistance gene cluster on linkage group 2. Tree Genetics & Genomes, 8(1), 53-60.
Campos, E., Miranda, D., Fischer, G., & Carranza, C. (2013). Los frutales caducifolios en Colombia. Especies y Variedades de hoja caduca en Colombia. 47-65 pp.
Carrasco, B., C., Moya, M., Gebauer, M., & García, R. (2012). Genetic characterization of Japanese plum cultivars (Prunus salicina) using SSR and ISSR molecular markers. Ciencia e Investigación Agraria, 39(3), 533-543.
Castro, S., Delgado, Z., Ramírez, F., & Puentes, M. (1998). Manejo Post-cosecha y Comercialización de Durazno (Prunus pérsica (L.) Batsch). Serie de paquete de Capacitación sobre Manejo de Post-cosecha de frutas y Hortalizas. NRI, DFID, SENA, Convenio SENA Reino Unido. OP Gráficas, Bogotá, Colombia. 372 p.
Crest, S., Tulman-Neto, A., & Figueira, A. (2001). Detection off single sequence repeat polymorphisms in denaturing polyacrilamide sequencing gels by silver staining. Plant Molecular Biology Reporter, 19(4), 299-306.
Dellaporta, S., Wood, J., & Hicks, J. (1983). A plant DNA minipreparation: Versión II. Plant Molecular Biology Reporter, 1(4), 19-21.
Dhanapal, A., Martínez, P., Gradziel, T., & Crisosto, H. (2012). First genetic linkage map chilling injury susceptibility in peach (Prunus persica L. Batsch) fruit with SSR and SNP markers. Journal of Plant Science & Molecular Breeding, 34(1), 1-13.
Ercisli, S., Agar, G., Yildirim, N., Duralija, B., Vokurka, A., & Karlidag H. (2011).Genetic diversity in wild sweet cherries (Prunus avium) in Turkey revealed by SSR markers. Genetics and Molecular Research, 10(2), 1211-1219.
Fan, L., Zhang, M., Liu, Q., Li, T., Song, Y., Wang, L., Zhang, S., & Wu, L. (2013). Transferability of newly developed pear SSR markers to other Rosaceae species. Plant Molecular Biology Reporter, 31(6), 1271-1282.
FAOSTAT (2014). Estadísticas de producción cultivos caducifolios. Consultado en: http://faostat.fao.org/.
Fathi, A., Ghareyazi, B., Haghnazari, A., Ghaffari, M., Pirseyedi, S., Kadkhodaei, S., Naghavi, M., & Mardi, M. (2008). Assessment of the genetic diversity of almond (Prunus dulcis) using microsatellite markers and morphological traits. Iranian Journal of Biotechnology, 6(2), 98-106.
Fernández, A., Martí, J., Alonso, M., Espiau, M., Rubio, M., & Socias, R. (2009). Genetic Diversity in Spanish and Foreign Almond Germoplasm Assessed by Molecular Characterization with Simple Sequence Repeats. Journal of the American Society for Horticultural Science, 134(5), 535-542.
Font, C., Gradziel, T., Gogorcena, y., & Moreno, M. (2014). Phenotypic diversity among local Spanish and foreign peach and nectarine (Prunus persica L.) Batsch accessions. Euphytica, 197(2), 261-277.
Frecon, J. (2006). Fruit register list 43: Peach. HortScience, 41(5), 1116-1119.
Gallego, A. (2010). El cultivo del almendro en producción ecológica. Asociación para el Desarrollo Sostenible del Poniente Granadino. 16p.
Guarino, C., Santoro, S., Simone, L., & Cipriani, G. (2009). Prunus avium: nuclear DNA study in wild populations and sweet cherry cultivars. Genome, 52(4), 320-337.
Gulen, H., Ipek, A., Ergin, S., Ackay, M.E. & Eris, A. (2010). Assessment of genetic relationships among 29 introduced and 49 local sweet cherry accessions in Turkey using AFLP and SSR markers. Journal of Horticultural Science & Biotechnology, 85(5), 427-431.
Hancock, J., Scorza, R., & Lobos, G. (2008). Peaches. In: J.F. Hancock (ed), Temperature Fruit Crop Breeding: Germoplasm to Genomics. Springer, East Lansing. 455p.
Hantula, J., Dusabenyagasani, M., & Hamelin, R. (1996). Random Amplified Microsatellites (RAMs) a novel method for characterizing genetic variation within fungi. European Journal of Forest Pathology, 26(3), 159-166.
Imbert, E., & Lefévre, F. (2003). Dispersal and gene flow of Populus nigra (Salicaceae) along a dynamic river system. Journal Ecology, 91(3), 447-456.
Kamm, U., Gugerli, F., Rotach, P., Edwards, P., & Holderegger, R. (2011). Genetic consequences of low local tree densities - Implications for the management of naturally rare, insect pollinated species in temperate forests. Forest Ecology and Management, 262(6), 1047-1053.
Mahuku, G., Henríquez, M., Muñoz, J., & Buruchara, R. (2002). Molecular markers dispute the existence of the Afro-Andean group of the Bean Angular Leaf Spot pathogen, Phaeoisariopsis griseola. Phytopahology, 92(6), 580-589.
Mariette, S., Tavaud, M., Arunyawat, U., Capdeville, G., Millan, M., & Salin, F. (2010). Population structure and genetic bottleneck in sweet cherry estimated with SSRs, and the gametophytic self-incompatibility locus. BMC Genetics, 11, 77-93.
McKey, D., Elias, M., Pujol, B., & Duputié, A. (2010). Ecological approaches to crop domestication. In: Gepts P, Bettinger R, Brush SB, Famula T, McGuire PE, Qualset CO, eds. Biodiversity in agriculture: domestication, evolution and sustainability. Cambridge, UK: Cambridge University Press. 456p.
Mora, S., Morillo, Y., Morillo, A., Caicedo, A., y Muñoz, J. (2013). Caracterización molecular con microsatélites aleatorios RAMs de 30 accesiones de mandarina (Citrus reticulata) del banco de germoplasma de Corpoica-Palmira. Investigación Agropecuaria, 10(2), 161-172.
Morillo, A., Morillo, Y., Chagüeza, Y., Caicedo, A., Jaramillo, J., Muñoz, O., Arcos, A., Vásquez, E., y Muñoz, J. (2009). Caracterización de la diversidad genética en naranja y comparación del polimorfismo de microsatélites amplificados al azar (RAMs) usando electroforesis de poliacrilamida y agarosa. Acta Agronómica, 58(4), 234-244.
Morillo, A., Morillo, Y., y Pinzón, E. (2014). Caracterización con RAMs de la colección de durazno (Prunus pérsica (L.) Batsch existente en la Universidad Pedagógica y Tecnológica de Colombia. Acta Agronómica, 63(4), 367-376.
Morillo, A., Morillo Y., Zamorano, A., Vásquez, E., y Muñoz, J. (2005). Caracterización molecular con microsatélites aleatorios RAM de la colección de mora Rubus spp, de la Universidad Nacional de Colombia, Sede Palmira. Acta Agronómica, 54(2), 15-24.
Muñoz, J., Morillo, A., & Morillo, Y. (2008). . Microsatélites Amplificados al Azar (RAMs) en estudios de diversidad genética vegetal. Acta Agronómica, 57(4), 219-226.
Nei, M., & Li, W. (1979). Mathematical model for studying genetic variation in terms of restricción endonucleasa. Proceedings of the National Academy of Sciences, 79(5), 5267-5273.
Okie, W., & Hancock J. (2008). Plums. In: Hancock J.F. (ed.). Temperate fruit crop breeding: germoplasm to genomics. Kluwer Academic Publisher. Dordrecht, Holland. 337-358 pp.
Ordóñez, O. (2007). Monografía del durazno, Secretaria de desarrollo Rural. Estado de Puebla. Disponible en: http://www.sdr.gob.mx/Contenido/CadenasProductivas.pdf.
Reales, A., Sargent, D., Tobutt, k., & Rivera, D. (2010). Phylogenetics of Eurasian plums, Prunus L. section Prunus (Rosaceae), according to coding and non-coding chloroplast DNA sequences. Tree Genetics & Genomes, 6(1), 37-45.
Rogatis, A., Ferrazzini, D., Ducci, F., Guerri, S., & Belleti, P. (2013). Genetic variation in Italian wild cherry (Prunus avium L.) as characterized by nSSR markers. Forestry, 86(1), 391-400.
Sanabria, H., García, H., y Muñoz, J. (2006).Caracterización molecular con marcadores RAM de árboles nativos de Psidium guajava (guayaba) en el Valle del Cauca. Acta Agronómica, 55(1), 23-30.
Sassa, H., Hirano, H., & Ikehashi, H. (1992).Self-incompatibility-related RNases in styles of Japanese pear (Pyrus serotina Rehd.). Plant Cell Physiology, 33(6), 811-814.
Serna, D., y Oliva, L. (1999). El cultivo del durazno. Cartilla divulgativa elaborada por CorpoTunía, Corporación para el Desarrollo de Tunía. 25 p.
Scalabrin, S., Policriti, A., Nadalin, F., Pinosio, S., Cattonaro, F., Vendramin, E., Aramini, V., Verde, I., Bassi, D., Pirona, R., Rossini, L., Cipriani, G., Testolin, R., & Morgante, M. (2013). A catalog of molecular diversity of Prunus germoplasm gathered from aligning NGS reads to the peach reference sequence: Bioinformatic approaches and challenges. Acta Horticulturae, 976 (3), 169-176.
Stanys, V., Frercks, B., Siksnianiene, B., Stepulaitiene, I., Gelvonauskiene, D., Staniene, G., & Bobinas, C. (2012). Identification of sweet cherry (Prunus avium L.) cultivars using AFLP and SSR markers. emdirbyst - Agriculture, 99(4), 437-444.
Stanys, V., Stanyt, R., Stanien, G., & Vinskien, J. (2008). S-allele identification by PCR analysis in Lithuanian sweet cherries. Biologija, 54(1), 22-26.
Stoeckel, S., Grange, J., Fernández, J., Bilger, I., Frascaria, N., & Mariette, S. (2006). Heterozygote excess in a self-incompatible and partially clonal forest tree species Prunus avium L. Molecular Ecology, 15(8), 2109-2118.
Vicedomini, R., Vezzi, F., Scalabrin, S., Arvestad, L., & Policriti, A. (2013). GAM-NGS: genomic assemblies merger for next generation sequencing. BMC Bioinformatics, 14(7), 1471-1481.
Wright, S. (1978). Evolution and the genetics of populations, variability within and among natural populations, Vol 4. University of Chicago Press. Chicago. 123p.
Yoon, J., Dongcheng, L., Wonseob, S., Weisheng, L., Aimin, Z. & Shaohua, L. (2006). Genetic diversity and ecogeographical phylogenetic relationships among Peach and Nectarine cultivars based on Simple Sequence Repeat (SSR) Markers. Journal of American Society for Horticultural Science, 131(4), 513-521.
Zietkiewicz, E., Rafalski, A., & Labuda, D. (1994). Genome fingerprinting by Simple Sequence Repeat (SSR)-anchored Polymerase Chain Reaction Amplification. Genomics, 20(2), 176-183.
Referencias
Aranzana, M. J., Abbassi, E. K., Howad, W., & Arus, P. (2010). Genetic variation, population structure and linkage disequilibrium in peach commercial varieties. BMC Genetics, 11(1), 69-79.
Baíza, V. (2004). Guía técnica del cultivo del melocotón. Programa Nacional de Frutas del Salvador. 46 p.
Barac, G., Ognjanov, V., Obreht, D., Ljubojevic, M., Bosnjakovic, D., Pejic, I., & Gasic, K. (2014). Genotypic and phenotypic diversity of cherry species collected in Serbia. Plant Molecular Biology Reporter, 32(1), 92-108.
Bonilla, M., Espinosa, K., Posso, A., & Muñoz, J. (2008). Caracterización molecular de 43 accesiones de uchuva de seis departamentos de Colombia. Acta Agronómica, 57 (2), 109-115.
Boskovic, R., Russell, K., & Tobutt, K. (1997). Inheritance of stylar ribonucleases in cherry progenies, and reassignment of incompatibility alleles to two incompatibility groups. Euphytica, 95(2), 221–228.
Bourguiba, H., Krichen, L., Audergon, J., Khadari, B., & Farah, N. (2010). Impact of mapped SSR markers on the genetic diversity of apricot (Prunus armeniaca L.) in Tunisia. Plant Molecular Biology Reporter, 28(4), 578-587.
Bouvier, L., Bourcy, M., Boulay, M., Tellier, M., Guérif, P., Denancé, C., Durel, C., & Lespinasse, Y. (2012). A new pear scab resistance gene Rvp1 from the European pear cultivar ‘Navara’ maps in a genomic region syntenic to an apple scab resistance gene cluster on linkage group 2. Tree Genetics & Genomes, 8(1), 53-60.
Campos, E., Miranda, D., Fischer, G., & Carranza, C. (2013). Los frutales caducifolios en Colombia. Especies y Variedades de hoja caduca en Colombia. 47-65 pp.
Carrasco, B., C., Moya, M., Gebauer, M., & García, R. (2012). Genetic characterization of Japanese plum cultivars (Prunus salicina) using SSR and ISSR molecular markers. Ciencia e Investigación Agraria, 39(3), 533-543.
Castro, S., Delgado, Z., Ramírez, F., & Puentes, M. (1998). Manejo Post-cosecha y Comercialización de Durazno (Prunus pérsica (L.) Batsch). Serie de paquete de Capacitación sobre Manejo de Post-cosecha de frutas y Hortalizas. NRI, DFID, SENA, Convenio SENA Reino Unido. OP Gráficas, Bogotá, Colombia. 372 p.
Crest, S., Tulman-Neto, A., & Figueira, A. (2001). Detection off single sequence repeat polymorphisms in denaturing polyacrilamide sequencing gels by silver staining. Plant Molecular Biology Reporter, 19(4), 299-306.
Dellaporta, S., Wood, J., & Hicks, J. (1983). A plant DNA minipreparation: Versión II. Plant Molecular Biology Reporter, 1(4), 19-21.
Dhanapal, A., Martínez, P., Gradziel, T., & Crisosto, H. (2012). First genetic linkage map chilling injury susceptibility in peach (Prunus persica L. Batsch) fruit with SSR and SNP markers. Journal of Plant Science & Molecular Breeding, 34(1), 1-13.
Ercisli, S., Agar, G., Yildirim, N., Duralija, B., Vokurka, A., & Karlidag H. (2011).Genetic diversity in wild sweet cherries (Prunus avium) in Turkey revealed by SSR markers. Genetics and Molecular Research, 10(2), 1211-1219.
Fan, L., Zhang, M., Liu, Q., Li, T., Song, Y., Wang, L., Zhang, S., & Wu, L. (2013). Transferability of newly developed pear SSR markers to other Rosaceae species. Plant Molecular Biology Reporter, 31(6), 1271-1282.
FAOSTAT (2014). Estadísticas de producción cultivos caducifolios. Consultado en: http://faostat.fao.org/.
Fathi, A., Ghareyazi, B., Haghnazari, A., Ghaffari, M., Pirseyedi, S., Kadkhodaei, S., Naghavi, M., & Mardi, M. (2008). Assessment of the genetic diversity of almond (Prunus dulcis) using microsatellite markers and morphological traits. Iranian Journal of Biotechnology, 6(2), 98-106.
Fernández, A., Martí, J., Alonso, M., Espiau, M., Rubio, M., & Socias, R. (2009). Genetic Diversity in Spanish and Foreign Almond Germoplasm Assessed by Molecular Characterization with Simple Sequence Repeats. Journal of the American Society for Horticultural Science, 134(5), 535-542.
Font, C., Gradziel, T., Gogorcena, y., & Moreno, M. (2014). Phenotypic diversity among local Spanish and foreign peach and nectarine (Prunus persica L.) Batsch accessions. Euphytica, 197(2), 261-277.
Frecon, J. (2006). Fruit register list 43: Peach. HortScience, 41(5), 1116–1119.
Gallego, A. (2010). El cultivo del almendro en producción ecológica. Asociación para el Desarrollo Sostenible del Poniente Granadino. 16p.
Guarino, C., Santoro, S., Simone, L., & Cipriani, G. (2009). Prunus avium: nuclear DNA study in wild populations and sweet cherry cultivars. Genom.52: 320–337. Genome, 52(4), 320-337.
Gulen, H., Ipek, A., Ergin, S., Ackay, M.E. & Eris, A. (2010). Assessment of genetic relationships among 29 introduced and 49 local sweet cherry accessions in Turkey using AFLP and SSR markers. Journal of Horticultural Science & Biotechnology, 85(5), 427-431.
Hancock, J., Scorza, R., & Lobos, G. (2008). Peaches. In: J.F. Hancock (ed), Temperate Fruit crop Breeding. Germplasm to Genomics. Springer, East Lansing. 455p.
Hantula, J., Dusabenyagasani, M., & Hamelin, R. (1996). Random Amplified Microsatellites (RAMs) a novel method for characterizing genetic variation within fungi. European Journal of Forest Pathology, 26(3), 159-166.
Imbert, E., & Lefévre, F. (2003). Dispersal and gene flow of Populus nigra (Salicaceae) along a dynamic river system. Journal Ecology, 91(3), 447-456.
Kamm, U., Gugerli, F., Rotach, P., Edwards, P., & Holderegger, R. (2011). Genetic consequences of low local tree densities – Implications for the management of naturally rare, insect pollinated species in temperate forests. Forest Ecology and Management, 262(6), 1047-1053.
Mahuku, G., Henríquez, M., Muñoz, J., & Buruchara, R. (2002). Molecular markers dispute the existence of the Afro-Andean group of the Bean Angular Leaf Spot pathogen, Phaeoisariopsis griseola. Phytopahology, 92(6), 580-589.
Mariette, S., Tavaud, M., Arunyawat, U., Capdeville, G., Millan, M., & Salin, F. (2010). Population structure and genetic bottleneck in sweet cherry estimated with SSRs, and the gametophytic self-incompatibility locus. BMC Genetics, 11, 77-93.
McKey, D., Elias, M., Pujol, B., & Duputié, A. (2010). Ecological approaches to crop domestication. In: GeptsP, BettingerR, Brush SB, FamulaT, McGuirePE, QualsetCO, eds. Biodiversity in agriculture: domestication, evolution and sustainability. Cambridge, UK: Cambridge University Press. 456p.
Mora, S., Morillo, Y., Morillo, A., Caicedo, A., y Muñoz, J. (2013). Caracterización molecular con microsatélites aleatorios RAMs de 30 accesiones de mandarina (Citrus reticulata) del banco de germoplasma de Corpoica-Palmira. Investigación Agropecuaria, 10(2), 161-172.
Morillo, A., Morillo, Y., Chagüeza, Y., Caicedo, A., Jaramillo, J., Muñoz, O., Arcos, A., Vásquez, E., y Muñoz, J. (2009). Caracterización de la diversidad genética en naranja y comparación del polimorfismo de microsatélites amplificados al azar (RAMs) usando electroforesis de poliacrilamida y agarosa. Acta Agronómica, 58(4), 234-244.
Morillo, A., Morillo, Y., y Pinzón, E. (2014). Caracterización con RAMs de la colección de durazno (Prunus pérsica (L.) Batsch existente en la Universidad Pedagógica y Tecnológica de Colombia. Acta Agronómica, 63(4), 367-376.
Morillo, A., Morillo Y., Zamorano, A., Vásquez, E., y Muñoz, J. (2005). Caracterización molecular con microsatélites aleatorios RAM de la colección de mora Rubus spp, de la Universidad Nacional de Colombia, Sede Palmira. Acta Agronómica, 54(2), 15-24.
Muñoz, J., Morillo, A., & Morillo, Y. (2008). . Microsatélites Amplificados al Azar (RAMs) en estudios de diversidad genética vegetal. Acta Agronómica, 57(4), 219-226.
Nei, M., & Li, W. (1979). Mathematical model for studying genetic variation in terms of restricción endonucleasa. Proceedings of the National Academy of Sciences, 79(5), 5267-5273.
Okie, W., & Hancock J. (2008). Plums. In: Hancock J.F. (ed.). Temperate fruit crop breeding: germoplasm to genomics. Kluwer Academic Publisher. Dordrecht, Holland. 337-358 pp.
Ordóñez, O. (2007). Monografía del durazno, Secretaria de desarrollo Rural. Estado de Puebla. Disponible en: http://www.sdr.gob.mx/Contenido/CadenasProductivas.pdf.
Reales, A., Sargent, D., Tobutt, k., & Rivera, D. (2010). Phylogenetics of Eurasian plums, Prunus L. section Prunus (Rosaceae), according to coding and non-coding chloroplast DNA sequences. Tree Genetics & Genomes, 6(1), 37-45.
Rogatis, A., Ferrazzini, D., Ducci, F., Guerri, S., & Belleti, P. (2013). Genetic variation in Italian wild cherry (Prunus avium L.) as characterized by nSSR markers. Forestry, 86(1), 391-400.
Sanabria, H., García, H., y Muñoz, J. (2006).Caracterización molecular con marcadores RAM de árboles nativos de Psidium guajava (guayaba) en el Valle del Cauca. Acta Agronómica, 55(1), 23-30.
Sassa, H., Hirano, H., & Ikehashi, H. (1992).Self-incompatibility-related RNases in styles of Japanese pear (Pyrus serotina Rehd.). Plant Cell Physiology, 33(6), 811-814.
Serna, D., y Oliva, L. (1999). El cultivo del durazno. Cartilla divulgativa elaborada por CorpoTunía, Corporación para el Desarrollo de Tunía. 25 p.
Scalabrin, S., Policriti, A., Nadalin, F., Pinosio, S., Cattonaro, F., Vendramin, E., Aramini, V., Verde, I., Bassi, D., Pirona, R., Rossini, L., Cipriani, G., Testolin, R., & Morgante, M. (2013). A catalog of molecular diversity of Prunus germoplasm gathered from aligning NGS reads to the peach reference sequence: Bioinformatic approaches and challenges. Acta Horticulturae, 976 (3), 169-176.
Stanys, V., Frercks, B., Siksnianiene, B., Stepulaitiene, I., Gelvonauskiene, D., Staniene, G., & Bobinas, C. (2012). Identification of sweet cherry (Prunus avium L.) cultivars using AFLP and SSR markers. Žemdirbystė - Agriculture, 99(4), 437-444.
Stanys, V., Stanytė, R., Stanienė, G., & Vinskienė, J. (2008). S-allele identification by PCR analysis in Lithuanian sweet cherries. Biologija, 54(1), 22–26.
Stoeckel, S., Grange, J., Fernández, J., Bilger, I., Frascaria, N., & Mariette, S. (2006). Heterozygote excess in a self-incompatible and partially clonal forest tree species Prunus avium L. Molecular Ecology, 15(8), 2109-2118.
Vicedomini, R., Vezzi, F., Scalabrin, S., Arvestad, L., & Policriti, A. (2013). GAM-NGS: genomic assemblies merger for next generation sequencing. . BMC Bioinformatics, 14(7), 1471-1481.
Wright, S. (1978). Evolution and the genetics of populations, variability within and among natural populations, Vol 4. University of Chicago Press. Chicago. 123p.
Yoon, J., Dongcheng, L., Wonseob, S., Weisheng, L., Aimin, Z. & Shaohua, L. (2006). Genetic diversity and ecogeographical phylogenetic relationships among Peach and Nectarine cultivars based on Simple Sequence Repeat (SSR) Markers. Journal of American Society for Horticultural Science, 131(4), 513-521.
Zietkiewicz, E., Rafalski, A., & Labuda, D. (1994). Genome fingerprinting by Simple Sequence Repeat (SSR)-anchored Polymerase Chain Reaction Amplification. Genomics, 20(2), 176-183.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
CrossRef Cited-by
1. Germán Ortuño-Hernández, Paula Sandoval-Belmar, David Ruiz, Pedro Martínez-Gómez, Claudio Meneses, Juan A. Salazar. (2025). Insights into the Molecular Basis of Fruit Development in Prunus Species. Plant Molecular Biology Reporter, 43(3), p.1397. https://doi.org/10.1007/s11105-025-01548-8.
Dimensions
PlumX
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2015 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).