Aprendizaje de Clases de Equivalencia de Redes Bayesianas basado en Búsqueda Competitiva de Hormigas Artificiales
Learning of bayesian networks equivalence classes based on competitive search of artificial ants
DOI:
https://doi.org/10.15446/rev.colomb.biote.v16n2.47234Palabras clave:
redes bayesianas, aprendizaje estructural, clases de equivalencia, colonia de hormigas, búsqueda heurística (es)Título en español: Aprendizaje de clases de equivalencia de redes bayesianas basado en búsqueda competitiva de hormigas artificiales
Título en ingles: Learning of bayesian networks equivalence classes based on competitive search of artificial ants
Resumen: Este artículo propone un algoritmo de aprendizaje de clases de equivalencia de redes bayesianas basado en un algoritmo de búsqueda Greedy y modelos de búsqueda inspirados en hormigas competitivas. Específicamente para el algoritmo propuesto, se obtuvo una mejor aproximación entre la red predicha y la red bayesiana teórica de ejemplo ASIA, con respecto a algoritmos anteriores, para conjuntos de datos con 20 y 500 muestras. En promedio el algoritmo desarrollado obtuvo una aproximación con respecto a la distancia estructural de hamming de 10.7% y 5.3% menor comparada con la obtenida por los algoritmos Greedy y de colonia de hormigas (ACO-E) respectivamente para 20 muestras, y de hasta el 6.8% menor con respecto al algoritmo ACO-E para 500 muestras. Además, para 500 muestras el número de llamadas a la función de puntaje realizadas por el algoritmo propuesto fue menor que las realizadas por el algoritmo ACO-E en el 90% de las combinaciones, concluyendo que hubo una reducción de la complejidad computacional. Finalmente se presentan los resultados de la aplicación del algoritmo propuesto a un microarreglo obtenido por muestras de pacientes con Leucemia Mieloide Aguda (LMA) con 6 nuevas interacciones con dependencias estadísticas como potenciales interacciones biológicas con alta probabilidad.
Palabras clave: redes bayesianas, aprendizaje estructural, clases de equivalencia, colonia de hormigas, búsqueda heurística.
Abstract: This article proposes an algorithm for learning equivalence classes of Bayesian networks based on a Greed search algorithm and search patterns inspired by competitive ants. Specifically, for the proposed algorithm, we obtained a better approximation between the predicted network and the theoretical network ASIA with respect to previous algorithms for data sets with 20 and 500 samples. On average, the algorithm developed an approximation with respect to Structural Hamming Distance of 10.7% and 5.3% lower than Greedy algorithms and ACO-E respectively to 20 samples, and up to 6.8% lower tan ACO-E algorithm for 500 samples. Furthermore, for 500 samples the number of calls to the scoring function performed by the algorithm proposed was smaller than in the ACO-E algorithm in 90% of the combinations, concluding that there was a reduction in the computational complexity. Finally, we present the results of applying the proposed algorithm to a microarray samples obtained from patients with acute myeloid leukemia (AML) with 6 new interactions with statistical dependencies as potential biological interactions with high probability.
Key words: Bayesian networks, learning sructural equivalence classes, ant colony, heuristic search.
Recibido: mayo 15 de 2013 Aprobado: octubre 1 de 2014
This article proposes an algorithm for learning equivalence classes of Bayesian networks based on a Greed search algorithm and search patterns inspired by competitive ants. Specifically, for the proposed algorithm, we obtained a better approximation between the predicted network and the theoretical network ASIA with respect to previous algorithms for data sets with 20 and 500 samples. On average, the algorithm developed an approximation with respect to Structural Hamming Distance of 10.7% and 5.3% lower than Greedy algorithms and ACO-E respectively to 20 samples, and up to 6.8% lower tan ACO-E algorithm for 500 samples. Furthermore, for 500 samples the number of calls to the scoring function performed by the algorithm proposed was smaller than in the ACO-E algorithm in 90% of the combinations, concluding that there was a reduction in the computational complexity. Finally, we present the results of applying the proposed algorithm to a microarray samples obtained from patients with acute myeloid leukemia (AML) with 6 new interactions with statistical dependencies as potential biological interactions with high probability.
Key words: Bayesian networks, learning sructural equivalence classes, ant colony, heuristic search.
https://doi.org/10.15446/rev.colomb.biote.v16n2.47234
ARTÍCULO DE INVESTIGACIÓN
Aprendizaje de clases de equivalencia de redes bayesianas basado en búsqueda competitiva de hormigas artificiales
Learning of bayesian networks equivalence classes based on competitive search of artificial ants
E. Fabián Cardozo*, Henry Arguello Fuentes**
* MSc. Ingeniería de Sistemas e Informática, Universidad Industrial de Santander, Bucaramanga - Colombia. e-mail: fabiancardozo@gmail.com
* PhD, Profesor Asociado, Universidad Industrial de Santander. e-mail:henarfu@uis.edu.co
Recibido: mayo 15 de 2013 Aprobado: octubre 1 de 2014
Resumen
Este artículo propone un algoritmo de aprendizaje de clases de equivalencia de redes bayesianas basado en un algoritmo de búsqueda Greedy y modelos de búsqueda inspirados en hormigas competitivas. Específicamente para el algoritmo propuesto, se obtuvo una mejor aproximación entre la red predicha y la red bayesiana teórica de ejemplo ASIA, con respecto a algoritmos anteriores, para conjuntos de datos con 20 y 500 muestras. En promedio el algoritmo desarrollado obtuvo una aproximación con respecto a la distancia estructural de hamming de 10.7% y 5.3% menor comparada con la obtenida por los algoritmos Greedy y de colonia de hormigas (ACO-E) respectivamente para 20 muestras, y de hasta el 6.8% menor con respecto al algoritmo ACO-E para 500 muestras. Además, para 500 muestras el número de llamadas a la función de puntaje realizadas por el algoritmo propuesto fue menor que las realizadas por el algoritmo ACO-E en el 90% de las combinaciones, concluyendo que hubo una reducción de la complejidad computacional. Finalmente se presentan los resultados de la aplicación del algoritmo propuesto a un microarreglo obtenido por muestras de pacientes con Leucemia Mieloide Aguda (LMA) con 6 nuevas interacciones con dependencias estadísticas como potenciales interacciones biológicas con alta probabilidad.
Palabras clave: redes bayesianas, aprendizaje estructural, clases de equivalencia, colonia de hormigas, búsqueda heurística.
Abstract
This article proposes an algorithm for learning equivalence classes of Bayesian networks based on a Greed search algorithm and search patterns inspired by competitive ants. Specifically, for the proposed algorithm, we obtained a better approximation between the predicted network and the theoretical network ASIA with respect to previous algorithms for data sets with 20 and 500 samples. On average, the algorithm developed an approximation with respect to Structural Hamming Distance of 10.7% and 5.3% lower than Greedy algorithms and ACO-E respectively to 20 samples, and up to 6.8% lower tan ACO-E algorithm for 500 samples. Furthermore, for 500 samples the number of calls to the scoring function performed by the algorithm proposed was smaller than in the ACO-E algorithm in 90% of the combinations, concluding that there was a reduction in the computational complexity. Finally, we present the results of applying the proposed algorithm to a microarray samples obtained from patients with acute myeloid leukemia (AML) with 6 new interactions with statistical dependencies as potential biological interactions with high probability.
Key words: Bayesian networks, learning sructural equivalence classes, ant colony, heuristic search.
Introducción
El surgimiento de tecnologías para obtener muestras de elementos de una célula a nivel molecular, ha traído consigo interés en inferir conocimiento estructural acerca de sus interacciones (Pe'er, 2005). El enfoque de biología de sistemas supone que las muestras están relacionadas con una estructura, donde los elementos muestreados no están aislados sino que interactúan para formar una red con propiedades y funciones propias (Pe'er, 2005). Dentro de diferentes modelos matemáticos que interpretan de manera estructural un fenómeno, las redes bayesianas han sido utilizadas para representar el sistema de interacciones moleculares. En estas aproximaciones se asume que el nivel de expresión de los genes que conforman la red y sus relaciones tiene un grado de incertidumbre, por lo tanto pueden ser representados como variables aleatorias y probabilidades condicionales entre ellas. Dada esta incertidumbre, un estado propio de la red se puede describir por una distribución de probabilidad conjunta de las variables (Jensen, 2007). Así, el análisis e interpretación de las muestras se define como el problema de encontrar una red bayesiana que mejor se ajuste a ellas. Conocida una posible red, es posible hacer aproximaciones de diferentes escenarios de las moléculas (Needham et al., 2009). El proceso de encontrar una red bayesiana consiste en obtener los parámetros de dicha red (Heckerman, 1995), y su estructura. Esto último, sin embargo, es un problema NP-Duro (Chickering, 1996) y se formaliza de la siguiente manera: dado EB, el conjunto de todas las estructuras posibles de redes bayesianas con n nodos, Sf(G):EB→ la función de puntaje que mide cada estructura {G} en EB; y M un algoritmo de búsqueda, el objetivo del aprendizaje de redes bayesianas consiste en encontrar la mejor estructura G* de una red bayesiana tal que:

Este enfoque es llamado puntaje búsqueda, donde los algoritmos diseñados tienen en común operadores para moverse dentro del espacio de búsqueda, y su diferencia radica en como se utilizan dichos operadores. Entre ellos se han presentado en la literatura, el algoritmo K2 (Cooper, 1992), algoritmos evolutivos (Wong, 2004), colonias de hormigas (de Campos, 2002), enjambre de partículas (Du et al., 2009), sistemas inmunes artificiales (Castro, 2005) y uno de los más utilizados, el Hill Climbing (Pe'er, 2005). Por otro lado, EB se puede dividir en clases de equivalencia en las cuales estructuras diferentes dentro de la misma clase pueden describir la misma distribución de probabilidad (Chickering, 2002). La principal ventaja de las clases de equivalencia reside en que se evita el movimiento dentro de una misma clase (Chickering, 2002). Sin embargo, incluso la búsqueda de clases de equivalencias no es trivial, sino que es de orden exponencial (Acid, 2003, Gillispie, 2006). Así, dada la complejidad de hallar clases de equivalencia, diseñar algoritmos más exactos para su inferencia es todavía un problema abierto (Daly, 2011). Entre los trabajos más recientes y con mejor desempeño se destacan extensiones del algoritmo greedy (Zhang, 2013), algoritmos evolutivos (Larrañaga, 2013) y aquellos que utilizan optimización basada en colonia de hormigas. En la última categoría se encuentra el algoritmo llamado ACO-E propuesto (Daly, 2009) y el propuesto en (Pinto et al., 2009) llamado MMACO.
En este artículo se propone un nuevo algoritmo para el aprendizaje de clases de equivalencia de redes bayesianas bayesianas extendiendo el trabajo previo en la categoría de colonia de hormigas, combinando el enfoque competitivo propuesto en (Pinto et al., 2009) y la extensión del algoritmo greedy usando operadores para moverse entre clases de equivalencia (Chickering, 2002). El enfoque competitivo de hormigas difiere del modo clásico en el sentido que un conjunto de hormigas construye un solo camino o solución en lugar de que cada hormiga construya su propio camino como en Daly, 2009). De esta forma, se reduce la complejidad computacional y por tanto se obtienen mejores resultados que el algoritmo de búsqueda Greedy clásico (Chickering, 2002) en términos del número de evaluaciones de la función de puntaje Sf y del número de interacciones erróneas en la estructura aproximada. Finalmente, este artículo describe la aplicación del algoritmo propuesto en la inferencia de interacciones de naturaleza estocástica entre moléculas que responden en relación a cambios de la molécula EVI-1 y su aporte al posible descubrimiento de interacciones reales entre ellas. La razón por la cual se escogió este gen, es su asociación a una de las formas más severas de un tipo de patología tumoral denominado Leucemia Mieloide Aguda (LMA) (Wieser. 2007).
Materiales y metodosModelado bayesiano para el aprendizaje de redes bayesianas
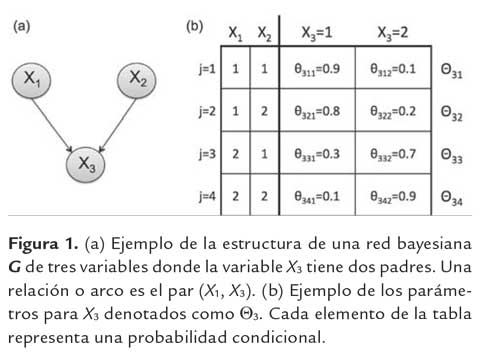
Una red bayesiana es una forma de representar el conocimiento de relaciones entre elementos a traves de un digrafo aciclico y un conjunto de probabilidades condicionales que cuantifican dichas relaciones, de tal forma que codifican una distribucion de probabilidad conjunta de los elementos. Como un ejemplo, una red bayesiana puede representar la regulacion en la expresion de un gen por otros dos genes como se muestra en la figura 1(a). En este caso, cada gen Xi tiene dos posibles estados: estado 1 si el gen esta activo o estado 2 si no lo esta. La regulacion en la red genetica es cuantificada por los parametros de la red bayesiana. En la figura 1(b) el parametro θ311 denota la probabilidad de que X3 este activo cuando X1 y X2 estan activos. Esto es, cuando ambos genes X1 y X2 estan expresados pueden actuar sobre el gen X3 activando su expresion. Las demas relaciones siguen el mismo razonamiento, permitiendo cuantificar las relaciones entre los elementos. Formalmente una Red Bayesiana es una dupla (G,Θ) que representa la distribucion de probabilidad conjunta:
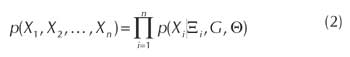
donde G = (X, A) es la estructura de la red representada por un digrafo acíclico tal que sus nodos representan el conjunto de variables aleatorias X = {X1, X2,..., Xn} con Xi ∈ {1,..., ri}y A ⊂ X2 define el conjunto de arcos dirigidos entre los nodos.
Ademas Ξi = {XkΙ (Xk, Xi) ¸ A} describe el conjunto de padres de Xi, y Θ es el conjunto de relaciones entre las variables en X definido como:
![]()
donde θijk representa la probabilidad, p(Xi = vkΙΞi = wj), wj es la jesima combinacion del conjunto Ω = {(v1,...,vΒ,...,vΙΞiΙ)Ι∀Β,XΒ = vΒ, XΒ ¸∈Ξi}y q† = ΙΩΙ. En la figura 1(b), Θ3 es el conjunto de todos los valores dentro de la tabla, Θ3j es el conjunto de valores de la fila j y Θ311 es igual a p(X3 = 1ΙΞ3 = w1. Entonces, el problema a resolver es inferir la dupla (G, Θ) que se ajuste a un conjunto de datos. Estudios se han realizado para obtener el conjunto Θ cuando la estructura G es conocida (Heckerman, 1995; Jensen, 2007). Sin embargo, obtener la estructura es un problema NP-Duro. Formalmente, el modelado bayesiano para aproximar una estructura G se basa en que, dado un conjunto de datos con D = [d1, d2,..., dm] con dh como el estado de las variables en X para un caso h y dada la hipotesis que dichos datos fueron generados a partir de la distribucion de probabilidad conjunta derivada de la red bayesiana con estructura G, la aproximacion bayesiana describe como se actualiza la probabilidad de que esa red genera dichos datos (Cooper, 1992; Heckerman, 1995; Jensen 2007). Asi, la probabilidad de obtener G a partir de D esta dado por:
![]()
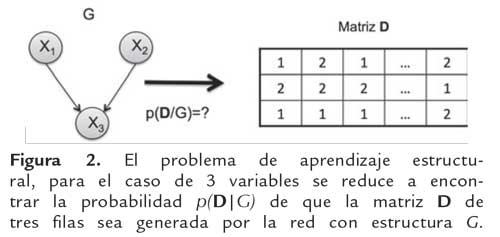
donde p(G) es la probabilidad a priori de la estructura hipotética, p(D) es una constante de normalización y p(DΙG) es la probabilidad marginal de D dada la red con estructura G. Asumiendo que p(G) es uniforme para todas las posibles estructuras, el problema de encontrar la función de puntaje Sf, se reduce a calcular la probabilidad que una red con estructura G pueda generar un conjunto de datos D, p(DΙG).
La figura (2) muestra la matriz D asociada a la estructura de la figura (1(a)). Cada fila i de D está asociada a la variable Xi y cada columna h de D es un caso h en que todas las variables han sido generadas. La pregunta a resolver es, ¿cuál es la probabilidad que los valores en la matriz D sean generados por la red con estructura G? Esta probabilidad define la función de puntaje que puede ser obtenida siguiendo el razonamiento realizado en (Cooper, 1992; Heckerman, 1995; Pe'er, 2005) como es descrito en detalle en el apéndice A. En resumen, a partir de las siguientes suposiciones: (1) El muestreo de las variables de X en D tiene distribución multinomial para cualquier estructura. (2) Cada caso dn es independiente de los demás y para cada variable Xi solo se puede tener un conjunto finito de estados. (3) Existe un valor para todas las variables en todos los casos en D. (4) Los parámetros asociados con cada variable en la estructura son independientes. (5) Los parámetros asociados con cada instancia de los valores de los padres de cada variable son independientes. (6) La función de densidad para los parámetros sigue una distribución de Dirichlet. De esta manera, la ecuación resultante es:
donde s(XiΙΞi) se define como:
La ecuación (5) es llamada ecuacion bayesiana de Dirichlet (BD), donde Γ(·) es la función Gamma, Nijk es el número de veces que la variable Xi tiene el valor vk y los padres Ξi tienen la combinacion de estado wj. El valor de Nijk tiene el mismo significado de Nijk pero con la diferencia que es un valor asumido antes de tener el conjunto de muestras D. Este parámetro Nfijk se ajusta a Nfijk = η.1/riqi asumiendo que dos estructuras equivalentes pueden tener el mismo puntaje y que en η posibles muestras iniciales la probabilidad de que Xi = wk es 1/ri y que Ξi = vj es 1/qi en cada muestra (Daly, 2009).
Clases de equivalencia de redes bayesianas
Formalmente, como es probado en (Chickering, 2002; Jensen, 2007), se dice que dos variables Xα, Xβ ∈ X de una red bayesiana estan direccionalmente separadas si existe una variable intermedia Xγ (diferente de Xα y Xβ) y se cumple una de las siguientes condiciones:
(1) existe una configuración de la forma Xα → Xγ → Xβ o de la forma Xα ← Xγ ← Xβ y se conoce el estado de la variable Xγ, (2) existe una configuración de la forma Xα ← Xγ → Xβ y se conoce el estado de la variable Xγ o (3) existe una configuración de la forma Xα → Xγ ← Xβ y no se conoce el estado de la variable Xγ. Si Xα está d-separada de Xβ dado el conocimiento del estado de la variable Xγ se dice que Xα y Xβ son condicionalmente independientes dado Xγ y se denota (Xα ⊥ XβΙXγ). De tal forma Xα, Xβ y Xγ, satisfacen la siguiente relación,

Dados estos conceptos, dos estructuras G y G' son equivalentes si y sólo si, para toda red bayesiana B = (G, Θ) existe una red Bayesiana B' = (G', Θ') tal que B y B'describen el mismo conjunto de independencias y tienen la misma distribución de probabilidad (Chickering 2002, Jensen 2007). De forma similar, dos estructuras G y G'son equivalentes si y sólo si tienen el mismo esqueleto y las mismas estructuras-v (Chickering, 2002).
Representación de Clases de Equivalencia
Dado el espacio no vacío EB de estructuras de redes bayesianas, la relación entre redes equivalentes en dicho espacio, denotada como ∼, cumple las propiedades de reflexividad, simetría y transitividad, formando todas ellas una clase de equivalencia. Así, para representar de una sola forma, una clase de equivalencia, se define a un digrafo acíclico parcial P con aristas dirigidas y no dirigidas, que describa todos los grafos de dicha clase, así:
![]()
Si la arista Xα → Xβ está presente en todo G ∈ [P], se dice que Xα → Xβ es forzada y si una arista Xα → Xβ no es forzada, se dice que es reversible. Un digrafo acíclico parcial completo Pc es aquel en el que toda arista forzada es una arista dirigida, y toda arista reversible es una arista no dirigida. Dado entonces un espacio de estructuras de redes bayesianas EB, existe un único digrafo acíclico parcial completo por cada clase de equivalencia en dicho espacio.
Movimientos entre clases de equivalencia
El conjunto de todas las clases de equivalencia de redes bayesianas de cierto numero de nodos es denominado EE, donde existen un conjunto de operadores para hacer posible el movimiento entre dichas clases o estados en un solo paso por medio de la transformacion de un digrafo aciclico parcial completo a otro. Este documento utiliza un conjunto de seis operadores con una prueba de validacion para cada uno, con la ventaja de que se puede calcular el puntaje del nuevo grafo generado solo con el puntaje local s(XiΙΞi de acuerdo a la tabla 1 y la ecuacion (6). Los teoremas y pruebas concernientes a la validacion de los operadores se encuentran detallados en (Chickering 2002) y tienen en cuenta que: (1) Ξα es el conjunto de Padres de Xα, es decir, el conjunto de nodos que tienen arcos dirigidos hacia Xα. (2) Ψα es el conjunto de vecindad de Xα, es decir, el conjunto de nodos que tienen arcos no dirigidos hacia Xα. (3) Ψα,β es la interseccion entre Ψα y Ψβ. (4) Ωαβ es el conjunto de padres de Xα que son vecinos de Xβ. Ademas, dentro de la funcion local de puntaje en la ultima columna, se utiliza la notacion Ψ+βα y Ψ.βα, que define el conjunto ΨαÜ{Xβ} y Ψα \{Xβ} correspondientemente. Finalmente, teniendo un operador y dado un digrafo aciclico parcial Ρ, un movimiento / es una instancia de un operador aplicado a un conjunto de nodos de P. M es el conjunto de todos los movimientos / validos a partir de Ρ. Dichos operadores hacen mas eficientes los algoritmos moviendose entre subconjuntos dentro de EB. Cada operador tiene como entrada dos o tres nodos de un grafo aciclico parcial completo y produce un grafo aciclico parcial de acuerdo a la operacion.
Colonia de hormigas en el aprendizaje
El método de optimización basado en colonia de hormigas es un algoritmo heurístico multiagente propuesto por Marco Dorigo (2005). Estos algoritmos son inspirados en el comportamiento colaborativo de las hormigas para encontrar la ruta más corta desde su nicho hasta la comida, y son utilizados con el fin de resolver problemas combinatorios de búsqueda y optimización con alta complejidad (o llamados NP-Duros).
Representación del problema
El primer elemento del problema de aprendizaje es el conjunto de componentes C = {c1, c2,..., cn} que se pueden combinar para obtener una solución. En este problema corresponde al conjunto de movimientos /que pueden ser realizados en un estado representado por el digrafo acíclico parcial P. Un camino (o solución) s es una secuencia de movimientos <li,...,lj> de tal forma que si se aplica cada uno de ellos (en el exacto orden) en un digrafo acíclico parcial P, dará como resultado un digrafo acíclico parcial completo que representa el estado actual. Un conjunto de m hormigas son los agentes que construyen una solución. Así, cada hormiga compite para incluir un nuevo movimiento al camino, hasta que todas construyen una solución S. Existe una feromona τi asociada a cada movimiento li ∈ C en una solución S. El comportamiento de cada τi es representado por un modelo que define su incremento de acuerdo a si el mejor camino S contiene el movimientoli y disminuye exponencialmente de acuerdo a un factor ρ de evaporación. Esta actualización se aplica tanto para la mejor solución de una iteración, como al mejor camino de todas las iteraciones. En el algoritmo todas las hormigas construyen una única solución o camino escogiendo cada una, un sólo movimiento, y aplicando solo aquel que produzca un mejor puntaje Sf hasta que no haya posibilidad de un mejor movimiento para cualquier hormiga.
Modelo de la Feromona
El modelo dinámico de la actualización de la feromona se describe de la siguiente manera:
1. Se inicializa la feromona τi asociada a todos los movimientos posibles desde un digrafo vacío de la siguiente manera:
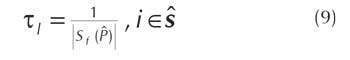
donde Sf esta dada por la ecuacion (5).
2. Se actualiza la feromona asociada a todos los movimientos que se han realizado utilizando una funcion de evaporacion, τi = (1 - ρ) τi.
3. En cada iteracion k, despues de que todas las hormigas han construido el mejor camino s, cada feromona τi es incrementada si el movimiento se encuentra en el digrafo Pk que representa el camino. Asi,

4. Además se actualiza la feromona de aquellos elementos que se relacionan con los movimientos del mejor camino de todas las iteraciones, así:

Información Heurística
La información heurística ηi define el puntaje para un movimiento l. Dado que un movimiento consiste en la transformación de un digrafo acíclico parcial en otro, ese cambio trae consigo un cambio en el puntaje de todo el digrafo. Sin embargo, puede ser simplificado como el cambio en la función local de puntaje s(XiΙΞi) definida en la ecuación (6) para cada operador como se muestra en la tabla 1.
Regla de Probabilidad
Cuando cualquier hormiga compite por escoger el mejor movimiento a partir del grafo parcial P, cada una utiliza una regla de transición pseudoaleatoria (Dorigo, 2005) que permite hacer un balance entre exploración y explotación para obtener el posible siguiente movimiento. Formalmente el siguiente movimiento l para una hormiga está dado por,
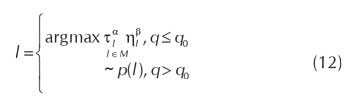
donde 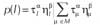 . Los parámetros α y β en el
rango [0,5] (Daly, 2009), son la potencia asignada a la
feromona y a la información heurística respectivamente,
q0 es un número aleatorio con distribución unifor-me en el intervalo [0,1] y es el conjunto de todos los
posibles movimientos para el grafo parcial .
. Los parámetros α y β en el
rango [0,5] (Daly, 2009), son la potencia asignada a la
feromona y a la información heurística respectivamente,
q0 es un número aleatorio con distribución unifor-me en el intervalo [0,1] y es el conjunto de todos los
posibles movimientos para el grafo parcial .
Descripción del Algoritmo
El algoritmo propuesto, mostrado en la tabla 3, está principalmente basado en el procedimiento para el aprendizaje de estructuras de redes bayesianas donde todas las hormigas colaborativamente construyen un camino por cada iteración a partir de una estructura vacía (Pinto et al., 2009). Además, el aprendizaje se hace en el espacio de clases de equivalencia utilizando una representación en la cual cada solución conduce a un digrafo acíclico parcial (Daly, 2009). La notación del algoritmo es presentada en la tabla 2.
Como describe la tabla 3, por cada iteración las hormigas construyen un camino, se escoge el mejor camino de todas las iteraciones y se actualiza el valor de la feromona asociada a cada movimiento. La construcción de una solución por el conjunto de hormigas es descrito en la función ANTS, en la tabla 4. A partir de un digrafo acíclico parcial P vacío, las hormigas construyen el camino de tal forma que cada una haya todos los posibles movimientos a partir del actual P y escoge uno de los movimientos de manera pseudoaleatoria de acuerdo a la regla de probabilidad dada en la ecuación (12). Al final, se aplica dicho movimiento a P obteniendo un nuevo digrafo Pk para la hormiga k.
Cuando todas las hormigas han escogido un posible siguiente movimiento, se escoge la solución sr, donde r∼U[1,...,m]. Si hubo al menos una hormiga que realizó un mejor movimiento, es decir, si Sf(Pr)>Sf(P), las hormigas vuelven a competir por el siguiente movimiento. De otro modo, se retorna a la solución P, se actualiza el mejor camino de todas las iteraciones y se actualiza el valor de la feromona de acuerdo a las ecuaciones (10) y (11).
Aproximación de una red de regulación genética
El algoritmo propuesto puede ser utilizado para mejorar el entendimiento de redes de regulación genética a partir de microarreglos de ADN. Un microarreglo de ADN es un chip que contiene información relativa al nivel de expresión de un conjunto de genes en una célula cuándo ésta última es estimulada. Este valor se obtiene al comparar el nivel de expresión genética de una célula de referencia con la información genética de una célula blanco (Wang, 2011). Por ejemplo, una célula de referencia puede ser una célula sana, y la célula blanco una anormal (Meloni, 2004). Matemáticamente, se puede expresar la relación entre la expresión del gen i en la célula blanco Bi con respecto a su expresión en la célula de referencia Rl como Ti = log2. Cuando Ti > 1 se dice que el gen i está activado en la célula blanco, y si Ti < i se dice que el gen i en la célula blanco está inhibida. De esa forma la discretización mas simple, esto es, de tres estados (inhibido, neutro, activado) es sencilla de hacer (Quackenbush 2002). Generalizando, si el número de células blanco es igual a m, para las cuales se desea obtener la información del nivel de expresión de n genes, el conjunto de datos final es una matriz D de n × m donde dij representa el radio Ti para la célula blanco j, donde i = 1,...,n y j = 1,...,m. A partir de esta matriz es posible obtener una aproximación de una red bayesiana que represente la red de regulación genética deseada, dependiendo de que genes se escojan.
Características del microarreglo utilizado
Como ejemplo, se seleccionó el microarreglo con número de acceso GSE6891 de la base de datos Gene Expression Omnibus (www.ncbi.nlm.nih.gov/geo). Este microarreglo contiene datos de expresión genética de grupos de pacientes con Leucemia Mieloide Aguda (LMA) con el objetivo de estudiar su aplicabilidad en la predicción de subclases asociadas a una anormalidad específica en la LMA (Verhaak, 2009). Dentro de sus resultados se encontró que dentro de una de aquellas subclases, uno de los genes más discriminantes para detectar anomalías de dicha clase fue el gen EVI-1 (o también llamado MECOM). Estas características hacen de este conjunto de datos propicio y viable para ser utilizado en el aprendizaje de la red. Específicamente el conjunto de datos cuenta con información de 461 muestras tomadas de la sangre o de la médula ósea. Para ilustrar el uso del algoritmo de hormigas propuesto en la sección anterior, de este microarreglo se obtuvieron solamente las filas de genes relevantes los cuáles tuvieron cambios de 6 veces o más en su valor cuando EVI-1 cambió su valor. De esta forma se obtiene un conjunto de genes relevantes en diferentes casos con respecto a EVI-1. Este es el conjunto de datos D utilizado por el algoritmo para aproximar la red de interacciones.
Obtención de una Red Semilla
Antes de realizar el proceso de aprendizaje es posible extraer conocimiento previo de interacciones moleculares. Esta información es almacenada en bases de datos. Se escogió la base de datos Biogrid (http://thebiogrid.org/) por ser la base de datos de acceso libre más completa, en la que se encontraban más interacciones, y en la que se mantenía el formato de los nombres de las moléculas como en los microarreglos. Para extraer una red inicial a partir de dicha información, se utilizó el algoritmo basado en el método descrito en (Djebari 2008) puesto que minimiza la redundancia en los datos. El algoritmo es el siguiente: (1) Se obtiene la red inicial de la biomolécula en estudio (EVI-1/MECOM), (2) de cada uno de los elementos de la red anterior se obtiene su propia red (ya que las interacciones de EVI- 1 son muy pocas), (3) se unen las redes filtrando las interacciones repetidas, (4) se filtran las interacciones de acuerdo a los genes listados en el microarreglo y se forma una red unidireccional. (5) Finalmente se aplica el algoritmo en (Djebbari 2008) basado en la técnica depth-first para convertir la red anterior en una grafo acíclico no dirigido. Teniendo entonces el conjunto de datos con los genes relevantes, entonces de la red inicial son utilizados aquellas interacciones cuyos genes se encuentran en el microarreglo.
Resultados y discusión
Evaluación del Algoritmo de Aprendizaje
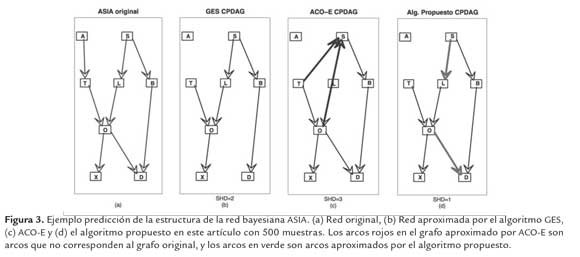
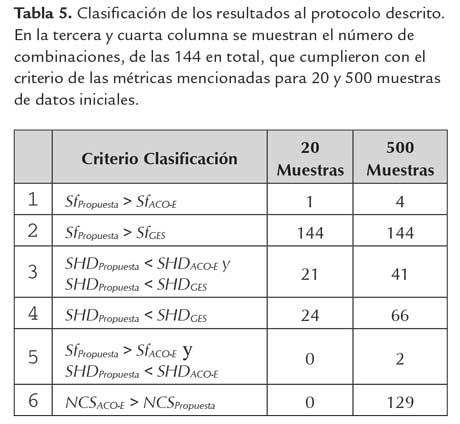
La metodologia de evaluacion del aprendizaje que se utilizo se baso en la descrita en (Daly, 2009), donde: (1) Se selecciona la red ASIA (figura 3(a)) como red teorica o estandar (http://compbio.cs.huji.ac.il/Repository/) para comparar los algoritmos Greedy (Chickering, 2002), ACO-E (Daly 2009) y el nuevo algoritmo propuesto. (2) Se selecciona un conjunto de valores para los parametros del algoritmo propuesto, manteniendo el numero de hormigas m igual a 15. Los conjuntos de valores son ρ =[0.05 0.1 0.3 0.5], q0 =[0.3 0.5 0.7], α =[2 3 4 5] y β =[1 1.5 2]. (3) Para el algoritmo ACO-E se mantuvieron los parametros obtenidos en (Daly 2009), es decir, ρ = 0.3, q0 = 0.7, α = 1 y β = 2.5. (4) Para cada una de las combinaciones de los valores de los parametros se hicieron las simulaciones respectivas para los tres algoritmos utilizando 20 conjuntos de datos con 20, y 500 muestras generadas a partir de la red bayesiana ASIA. El rendimiento y la red o estructura obtenida por cada uno de los algoritmos se evaluaron utilizando las siguientes metricas: (a) la funcion de Puntaje (Sf) para evaluar la cercania de la red construida y los datos segun la ecuacion (5) y (6). Valores altos indican alta probabilidad de que los datos hayan sido generados por el grafo obtenido. (b) Distancia Estructural de Hamming (SHD): Es la diferencia entre los arcos anadidos, omitidos e invertidos (en direccion) entre la estructura generada por el algoritmo y la estructura de la cual se generaron los datos iniciales. Una distancia menor significa entonces, que el digrafo aproximado es mas cercano al teórico. (c) Número de evaluaciones o llamadas estadísticas (NSC) que mide la complejidad computacional del algoritmo al calcular el número de llamadas que hace el algoritmo a la función objetivo. (5) Para la función de puntaje se escogió el tamaño de muestreo equivalente η = 4 utilizado en (Daly, 2009). En la figura 3(b)-(d) se muestra el resultado del protocolo descrito anteriormente para la red ASIA, para una simulación utilizando 500 muestras. La primera red, es la red teórica de la cual se obtienen los datos, las demás son las redes aproximadas por los algoritmos GES, ACO-E y el algoritmo propuesto, respectivamente. El número total de combinaciones para los valores de los parámetros del algoritmo fue 144. Como se muestra en la tabla 5 estas combinaciones se clasificaron de acuerdo a los valores obtenidos en las métricas Sf, SHD y NSC para los tres algoritmos probados. De acuerdo a la segunda fila de la tabla 5, sin importar el número de muestras y la combinación de parámetros, el nuevo algoritmo propuesto tiene una mayor probabilidad de generar los datos que el algoritmo Greedy.
Para el criterio utilizado con respecto a SHD, se presentaron 21 combinaciones de los parámetros para los cuales el algoritmo propuesto tuvo menor valor con respecto a los otros dos algoritmos, es decir es mas eficiente al obtener un grafo mas cercano al teórico. En promedio, se obtuvo un 2,6 % y 4,8 % menor valor que para los algoritmos Greedy y ACO-E respectivamente (figura 4(a-b)). De acuerdo a la suma cuadrática de las diferencias en los valores del SHD de estas 21 combinaciones, la mayor diferencia fue para la combinación ρ = 0,1, q0 = 0,3, α = 5 y β = 1.
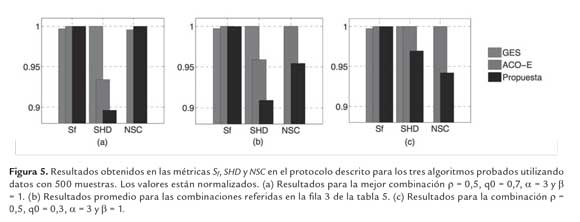
Como es descrito en la figura 4(c), la diferencia de combinaciones erroneas del grafo final del algoritmo propuesto con respecto al algoritmo ACO-E fue de 10,7% menor y con el algoritmo Greedy del 5,3 % menor. Los resultados anteriores muestran que el algoritmo propuesto puede obtener estructuras con menos interacciones erroneas que los algoritmos ACO-E y Greedy para redes pequenas, usando 20 muestras. Para 500 muestras, segun la primera fila de la tabla 5, de las 144 combinaciones de los parametros, en cuatro de ellos el algoritmo propuesto obtuvo mayor valor en Sf con respecto al algoritmo ACO-E. De estas cuatro, aquella combinacion con mayor diferencia en la metrica SHD, ρ = 0,5, q0 = 0,7, α = 5 y β = 1, obtuvo un valor del 10,4 % y 4 % de menos combinaciones erroneas en el grafo final con respecto al algoritmo GES y ACO-E respectivamente, (figura 5(a)). En la tercera fila de la tabla 5, de las 144 combinaciones se obtuvieron 41 para las cuales el valor de SHD del algoritmo propuesto fue menor que los otros dos. Especificamente, en promedio el valor fue de un 5% y 9% menor con respecto al algoritmo ACO-E y Greedy respectivamente como se muestra en la figura 5(b). Segun la quinta fila de la tabla 5, en dos de ellas se obtuvo un mayor valor para la funcion de puntaje. Una de ellas es la mostrada en la figura 5(a), y la otra es la combinacion ρ = 0,5, q0 = 0,3, α = 5 y β = 1, la cual obtuvo un valor del SHD 3% menor para el algoritmo propuesto con respecto a los otros dos algoritmos. La ultima fila de la tabla 5 hace referencia a la medida de complejidad del algoritmo NSC. El numero de llamadas a la funcion de puntaje por el algoritmo propuesto fue menor que en el algoritmo ACO-E en el 90% de las combinaciones utilizando 500 muestras.
En promedio esta diferencia fue del 6,8%, denotando que la complejidad del nuevo algoritmo es menor. Adicionalmente, para las combinaciones referidas en la fila 3 de la tabla 5, mostradas en la figura 5(b), en promedio el algoritmo propuesto obtuvo una disminución del 4,6 % en la métrica SHD con respecto al algoritmo ACO-E. Y por último en promedio para la combinación mostrada en la figura 5(c), referida en la fila cinco de la tabla 5 obtuvo un valor del NSC del 5,8 % menor que el algoritmo ACO-E. Todo lo anterior indica que el algoritmo propuesto, para la mayoría de combinaciones, usando 500 muestras de redes pequeñas tiene un orden de complejidad menor que el algoritmo ACO-E. Además, para las combinaciones referidas en la fila 5 de la tabla 5 y mostradas en la figura 5, tienen la posibilidad de obtener estructuras con menos interacciones erróneas (falsos positivos) que los algoritmos ACO-E y Greedy, y a su vez con mayor o muy semejante probabilidad como indica la función de puntaje.
Aproximación de la red de regulación genética
Dado que es posible que haya información espuria
en varios casos en la tabla de datos, que produzcan posibles falsos positivos, se infirieron un conjunto de
M estructuras y se seleccionaron las interacciones en
común de todas ellas. Para cada estructura Gj obtenida
se obtiene su función de puntaje o probabilidad de
aproximar la información en los datos Sf(Gi) y a partir
de ellas se obtiene un medidor de confianza por el
promedio de las probabilidades, ![]() . El resultado
es entonces un grafo G con las interacciones en común
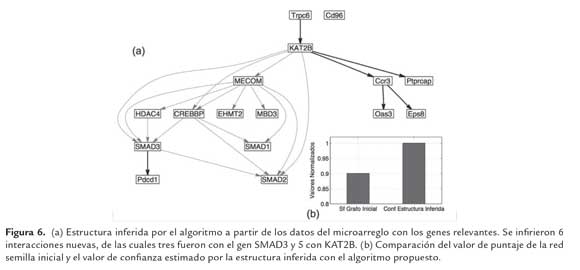
mencionadas, con un valor de confianza conf(G) (Pe'er, 2005). Para los datos del microarreglo con los
genes relevantes filtrados, el resultante modelo G inferido
por el algoritmo propuesto, con un número de 20
arcos entre 16 componentes. La figura 6(b) describe
la comparación del valor de confianza según la ecuación
(14) obtenido para las estructuras inferidas con el
algoritmo propuesto, y la estructura G es presentada
en en la figura 6(a) con las nuevas interacciones en
rojo. Como muestran los valores, el valor de confianza
obtenido por el algoritmo propuesto tiene un puntaje
del 10% mayor para la estructura inferida que para la
estructura inicial. El algoritmo es capaz de inferir interacciones
de naturaleza estadística, y a pesar de que
tiene la posibilidad de obtener estructuras con menos
interacciones erróneas, las interacciones reales sólo
son comprobables a través de métodos experimentales.
Sin embargo, estas nuevas interacciones obtenidas
con el algoritmo son de potencial importancia al elucidar
la exacta interacción que hay entre los elementos.
. El resultado
es entonces un grafo G con las interacciones en común
mencionadas, con un valor de confianza conf(G) (Pe'er, 2005). Para los datos del microarreglo con los
genes relevantes filtrados, el resultante modelo G inferido
por el algoritmo propuesto, con un número de 20
arcos entre 16 componentes. La figura 6(b) describe
la comparación del valor de confianza según la ecuación
(14) obtenido para las estructuras inferidas con el
algoritmo propuesto, y la estructura G es presentada
en en la figura 6(a) con las nuevas interacciones en
rojo. Como muestran los valores, el valor de confianza
obtenido por el algoritmo propuesto tiene un puntaje
del 10% mayor para la estructura inferida que para la
estructura inicial. El algoritmo es capaz de inferir interacciones
de naturaleza estadística, y a pesar de que
tiene la posibilidad de obtener estructuras con menos
interacciones erróneas, las interacciones reales sólo
son comprobables a través de métodos experimentales.
Sin embargo, estas nuevas interacciones obtenidas
con el algoritmo son de potencial importancia al elucidar
la exacta interacción que hay entre los elementos.
Conclusiones
El principal resultado de este trabajo fue el desarrollo de un algoritmo de aprendizaje de clases de equivalencia basado en modelos de búsqueda inspirado en colonia de hormigas. Además se describieron las ventajas del enfoque de puntaje y búsqueda entre clases de equivalencia de redes bayesianas. Principalmente,se evita el movimiento redundante por medio de operadores. Los resultados mostraron que el algoritmo propuesto para redes pequeñas, usando 20 muestras, y en específicas combinaciones de sus parámetros, tiene la posibilidad de obtener estructuras con menos interacciones erróneas (falsos positivos) que los algoritmos ACO-E y Greedy. Además, los resultados mostraron que, para 500 muestras, y redes pequeñas, el algoritmo propuesto tiene la posibilidad de obtener estructuras con menos interacciones erróneas (falsos positivos) que los algoritmos ACO-E y Greedy, y a su vez con mayor o muy semejante probabilidad como indica la función de puntaje y con un orden de complejidad menor que el algoritmo ACO-E. Además, se describió la aplicación de la inferencia de interacciones de naturaleza estocástica entre moléculas que responden en relación a cambios de la molécula EVI-1 y su aporte al descubrimiento de interacciones reales entre ellas. Se describieron los conceptos básicos sobre microarreglos necesarios para la aplicación del algoritmo de aprendizaje de redes bayesianas. Se mostró como es posible emplear datos de microarreglo para la selección de genes relevantes de acuerdo a cambios significativos cuando hay cambios en las condiciones celulares. También se mostró la utilidad de utilizar información a priori de la literatura científica para direccionar mas precisamente el aprendizaje. Por último se mostró los resultados de la aplicación del algoritmo a un microarreglo obtenido por muestras de pacientes con Leucemia Mieloide Aguda (LMA) con el fin de encontrar interacciones con dependencias estadísticas como potenciales interacciones biológicas con alta probabilidad. Este último resultado es de gran importancia puesto que aporta en la generación de conocimiento en investigaciones de como interactúan los genes relacionados con enfermedades como la LMA. Por ejemplo, es posible que al expresarse uno de los genes directamente regule al otro en la dirección aproximada por el algoritmo. También es posible que la regulación entre los genes esté en la dirección inferida, pero puede ser mediada por otros genes que no fueron tenidos en cuenta como genes relevantes en la sección anterior. Por ejemplo los genes SMAD3 y Pdcd1 aproximados por el algoritmo podrían tener genes intermedios. Por último, las interacciones pueden llegar a ser inversas, es decir, en cualquiera de los casos anteriores es posible que la dirección de la regulación sea contraria a la que se aproximó con el algoritmo.
Apéndice A: Deducción de la función de puntaje
Siguiendo el razonamiento realizado en (Cooper, 1992; Heckerman, 1995) para obtener la probabilidad P(DIG) en la ecuacion (5) y (6), se tiene en cuenta que:
1. Se supone que el muestreo de las variables de X en D tiene distribucion multinomial para cualquier
estructura G. Entonces, dado D como el arreglo de
los dih y como los valores de Ξi en dh donde σi = 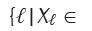 , para todo G en EB, existe un conjunto de
valor positivo Θ tal que,
, para todo G en EB, existe un conjunto de
valor positivo Θ tal que,
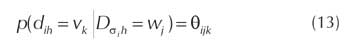
donde ![]() . Dada la existencia de los parametros
Θ={θijk}, se define la ecuacion de puntaje (4) teniendo
en cuenta la incertidumbre que se tiene de
ellos, calculando el promedio de la probabilidad
P(DΙG) sobre todos los posibles valores de los parametros
en G, asi:
. Dada la existencia de los parametros
Θ={θijk}, se define la ecuacion de puntaje (4) teniendo
en cuenta la incertidumbre que se tiene de
ellos, calculando el promedio de la probabilidad
P(DΙG) sobre todos los posibles valores de los parametros
en G, asi:
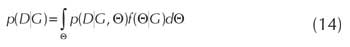
Entonces se requiere definir de (14) la probabilidad p(DΙG,Θ) con respecto a los datos y f(ΘΙG) con respecto a los parámetros.
2. Se asume que cada caso dh es independiente de los demás y para cada variable Xi solo se puede tener un conjunto finito de estados, de tal forma que:
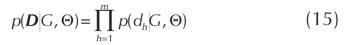
3. Se supone que existe un valor para todas las variables en todos los casos en D, entonces la expresión (15) se puede escribir como,

Sin embargo, dado que G establece los elementos de Ξ se puede descomponer la expresion anterior de acuerdo a las contribuciones locales de cada variable,
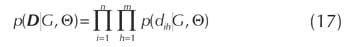
Además, si se define Nijk como el número de veces que dih es igual a un valor vk y Dσih a un elemento wj ∈ Ω para; para todo h en D, la ecuacion anterior puede reagruparse de la siguiente manera,
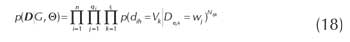
Usando (13) y (18) se tiene que
4. Se asume que los parámetros asociados con cada variable en la estructura son independientes, entonces se tiene que:

A esta propiedad se le llama Independencia Paramétrica Global.

5. Se asume que los parámetros asociados con cada instancia de los valores de los padres de cada variable son independientes. Esto se puede expresar como:
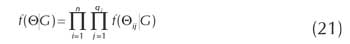
Esta propiedad tiene el nombre de Independencia Paramétrica Local. Así a partir de (19) y (21), la ecuación (14) se puede reescribir de la siguiente forma:
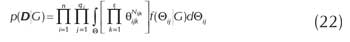
6. Por ultimo, se supone que la funcion de densidad para los parametros en Θij sin conocer D (o a priori), sigue una distribucion de Dirichlet principalmente porque, al actualizarse cuando se hace un muestro multinomial D, siguen siendo Dirichlet (Heckerman 1995). Asi, la funcion de densidad para los parametros es como sigue,
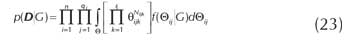
donde Nijk´ es la informacion a priori para las probabilidades
numericas θijk sin tener D. Basado en
las suposiciones anteriores, y dado que la integral
en (22) describe ![]() con respecto a ƒ(ΘijΙG), la
ecuacion resultante para (14) solucionando la integral
segun (Heckerman 1995) es:
con respecto a ƒ(ΘijΙG), la
ecuacion resultante para (14) solucionando la integral
segun (Heckerman 1995) es:

Referencias bibliográficas
1. Acid, S. and de Campos, L. M. 2003. Searching for bayesian network structures in the space of restricted acyclic partially directed graphs. Journal of Artificial Intelligence Research. 18: 445-490.
2. Castro, P. A. D. and Von Zuben, F. J. 2005. An immune-inspired approach to bayesian networks. in HIS '05: Proceedings of the Fifth International Conference on Hybrid Intelligent Systems, (Washington, DC, USA), pp. 23-28, IEEE Computer Society, 2005.
3. Chickering, D. M. 1996. Learning bayesian networks is np-complete. Pp. 121-130.
4. Chickering, D. M. 2002. Learning equivalence classes of bayesiannetwork structures. The Journal of Machine Learning Research. 2: 445 - 498.
5. Cooper, G. F. and Herskovits, E. 1992. A bayesian method for the induction of probabilistic networks from data. Machine Learning. 9(4): 309-347.
6. Daly, R. and Shen, Q. 2009. Learning bayesian network equivalence classes with ant colony optimization. Artificial Intelligence Research. 35: 391-447.
7. Daly, R. Shen; Q. Aitken, S. 2011. Learning Bayesian networks: approaches and issues. The Knowledge Engineering Review. 26(2): 99-157.
8. De Campos, L. M.; Fernández-Luna, J. M.; Gámez, J. A., and Puerta, J. M.. 2002. Ant colony optimization for learning bayesian networks. International Journal of Approximate Reasoning. 31(3): 291 - 311.
9. Djebbari, A.; Quackenbush, J. 2008. Seeded bayesian networks: Constructing genetic networks from microarray data. BMC Systems Biology. 2(1): 57.
10. Dorigo, M.; Blum, C. 2005. Ant colony optimization theory: A survey. Theoretical Computer Science. 344(2-3): 243 - 278.
11. Du, Z.; Wang, Y.; Ji; Z. June 2009. A new structure learning method for constructing gene networks. Bioinformatics and Biomedical Engineering, ICBBE 2009. 3rd International Conference. Pp. 1-4.
12. Gillispie, S. B. 2006. Formulas for counting acyclic digraph markov equivalence classes. Journal of Statistical Planning and Inference. 136(4): 1410 - 1432.
13. Heckerman, D. 1995. A tutorial on learning with bayesian networks. Tech. Rep., Microsoft Research.
14. Jensen, F. V. and Nielsen, T. D. 2007. Bayesian Networks and Decision Graphs. Springer Science + Business Media, LLC.
15. Larrañaga, P.; Karshenas, H.; Bielza, C. and Santana R. 2013. A review on evolutionary algorithms in Bayesian network learning and inference tasks. Information Sciences. 233: 109-125.
16. Meloni, R.; Khalfallah, O., and Faucon Biguet, N. 2004. DNA microarrays and pharmacogenomics. Pharmacological Research. 49(4): 303 - 308.
17. Needham, C.; Manfield, I.; Bulpitt, A.; Gilmartin, P.; and Westhead, D. 2009. From gene expression to gene regulatory networks in arabidopsis thaliana. BMC Systems Biology. 3(1): 85.
18. Pe'er, D. 2005. Bayesian Network Analysis of Signaling Networks: A Primer. Sci. STKE, 281: 14.
19. Pinto, P.; Nagele, A.; Dejori, M.; Runkler, T.; and Sousa, J. 2009.Using a local discovery ant algorithm for bayesian network structure learning. Evolutionary Computation, IEEE Transactions on. 13: 767-779.
20. Quackenbush, J. 2002. Microarray data normalization and transformation. Nature Genetics. 32: 496-501.
21. Verhaak, R.G.W.; Wouters, B.J.; Erpelinck, C.A.J.; Abbas, S.; Beverloo, H.B.; Lugthart, S.; Lwenberg, B.; Delwel, R.; and Valk, P.J.M. 2009. Prediction of molecular subtypes in acute myeloid leukemia based on gene expression profiling. Haematologica. 94(1): 131- 134.
22. Wang, L. and Li, Paul C.H. 2011. Microfluidic DNA microarray analysis: A review. Analytica Chimica Acta. 687(1): 12 - 27.
23. Wieser, R. 2007. The oncogene and developmental regulator evi1: Expression, biochemical properties, and biological functions. Gene. 396(2): 346 - 357.
24. Wong, M. L. and Leung, K. S. Aug. 2004. An efficient data mining method for learning bayesian networks using an evolutionary algorithm-based hybrid approach. Evolutionary Computation, IEEE Transactions. 8: 378-404.
25. Zhang, Y.; Zhang,W.; Xie, Y. 2013. Improved heuristic equivalent search algorithm based on Maximal Information Coefficient for Bayesian Network Structure Learning. Neurocomputing. 117: 186-195.
Referencias
Acid, S. and de Campos, L. M. 2003. Searching for bayesian network structures in the space of restricted acyclic partially directed graphs. Journal of Artificial Intelligence Research. 18: 445-490.
Castro, P. A. D. and Von Zuben, F. J. 2005. An immune-inspired approach to bayesian networks. in HIS '05: Proceedings of the Fifth International Conference on Hybrid Intelligent Systems, (Washington, DC, USA), pp. 23-28, IEEE Computer Society, 2005.
Chickering, D. M. 1996. Learning bayesian networks is np-complete. Pp. 121-130.
Chickering, D. M. 2002. Learning equivalence classes of bayesiannetwork structures. The Journal of Machine Learning Research. 2: 445 - 498.
Cooper, G. F. and Herskovits, E. 1992. A bayesian method for the induction of probabilistic networks from data. Machine Learning. 9(4): 309-347.
Daly, R. and Shen, Q. 2009. Learning bayesian network equivalence classes with ant colony optimization. Artificial Intelligence Research. 35: 391-447.
Daly, R. Shen; Q. Aitken, S. 2011. Learning Bayesian networks: approaches and issues. The Knowledge Engineering Review. 26(2): 99-157.
De Campos, L. M.; Fernández-Luna, J. M.; Gámez, J. A., and Puerta, J. M.. 2002. Ant colony optimization for learning bayesian networks. International Journal of Approximate Reasoning. 31(3): 291 - 311.
Djebbari, A.; Quackenbush, J. 2008. Seeded bayesian networks: Constructing genetic networks from microarray data. BMC Systems Biology. 2(1): 57.
Dorigo, M.; Blum, C. 2005. Ant colony optimization theory: A survey. Theoretical Computer Science. 344(2-3): 243 - 278.
Du, Z.; Wang, Y.; Ji; Z. June 2009. A new structure learning method for constructing gene networks. Bioinformatics and Biomedical Engineering, ICBBE 2009. 3rd International Conference. Pp. 1-4.
Gillispie, S. B. 2006. Formulas for counting acyclic digraph markov equivalence classes. Journal of Statistical Planning and Inference. 136(4): 1410 - 1432.
Heckerman, D. 1995. A tutorial on learning with bayesian networks. Tech. Rep., Microsoft Research.
Jensen, F. V. and Nielsen, T. D. 2007. Bayesian Networks and Decision Graphs. Springer Science + Business Media, LLC.
Larrañaga, P.; Karshenas, H.; Bielza, C. and Santana R. 2013. A review on evolutionary algorithms in Bayesian network learning and inference tasks. Information Sciences. 233: 109-125.
Meloni, R.; Khalfallah, O., and Faucon Biguet, N. 2004. DNA microarrays and pharmacogenomics. Pharmacological Research. 49(4): 303 - 308.
Needham, C.; Manfield, I.; Bulpitt, A.; Gilmartin, P.; and Westhead, D. 2009. From gene expression to gene regulatory networks in arabidopsis thaliana. BMC Systems Biology. 3(1): 85.
Pe'er, D. 2005. Bayesian Network Analysis of Signaling Networks: A Primer. Sci. STKE, 281: 14.
Pinto, P.; Nagele, A.; Dejori, M.; Runkler, T.; and Sousa, J. 2009.Using a local discovery ant algorithm for bayesian network structure learning. Evolutionary Computation, IEEE Transactions on. 13: 767-779.
Quackenbush, J. 2002. Microarray data normalization and transformation. Nature Genetics. 32: 496-501.
Verhaak, R.G.W.; Wouters, B.J.; Erpelinck, C.A.J.; Abbas, S.; Beverloo, H.B.; Lugthart, S.; Lwenberg, B.; Delwel, R.; and Valk, P.J.M. 2009. Prediction of molecular subtypes in acute myeloid leukemia based on gene expression profiling. Haematologica. 94(1): 131- 134.
Wang, L. and Li, Paul C.H. 2011. Microfluidic DNA microarray analysis: A review. Analytica Chimica Acta. 687(1): 12 - 27.
Wieser, R. 2007. The oncogene and developmental regulator evi1: Expression, biochemical properties, and biological functions. Gene. 396(2): 346 - 357.
Wong, M. L. and Leung, K. S. Aug. 2004. An efficient data mining method for learning bayesian networks using an evolutionary algorithm-based hybrid approach. Evolutionary Computation, IEEE Transactions. 8: 378-404.
Zhang, Y.; Zhang,W.; Xie, Y. 2013. Improved heuristic equivalent search algorithm based on Maximal Information Coefficient for Bayesian Network Structure Learning. Neurocomputing. 117: 186-195.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Licencia
Derechos de autor 2014 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).