Biología molecular, una herramienta para la bioprospección del metabolismo secundario de plantas en Colombia Ejemplo práctico en plantas colombianas de interés medicinal
Molecular biology, a tool for bioprospection of plants secondary metabolism in Colombia
Palabras clave:
Bioprospection, secondary metabolism, degenerate primer, microarrays, PLP-dependent decarboxylases, bioprospección, metabolismo secundario, cebadores degenerados, microarreglos, decarboxilasas dependientes de piridoxal (es)|
|
||
|
_____________________REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL. VI No. 2 Diciembre 2004 67-77
Biología molecular, una herramienta
para la bioprospección del metabolismo secundario
de plantas en Colombia
Ejemplo práctico en plantas colombianas de interés medicinal
Molecular biology, a tool for bioprospection of plants secondary metabolism in Colombia
Natalia Palacios Rojas*,**, Daniel Burtin**, Mark Leech*
RESUMEN
Los metabolitos secundarios producidos por las plantas están involucrados en una multitud de interacciones ecológicas, entre ellas las interacciones planta-planta, planta-microorganismos, planta-animales y planta-insectos. Además de protegerlas de algunos patógenos, también ayudan a las plantas a sobrevivir en condiciones medioambientales adversas. Dadas sus características medicinales e industriales, dichos metabolitos también son de gran importancia para el ser humano. Sin embargo, el estudio de estos compuestos en plantas tropicales a nivel bioquímico, molecular y genético, es aún limitado. Dentro de las estrategias de bioprospección de la diversidad de muchos países tropicales como Colombia, la exploración y explotación del metabolismo secundario de plantas constituye un reglón importante. En el presente trabajo se reporta una metodología experimental basada en el aislamiento de ADN genómico de plantas colectadas en campo, y el uso de cebadores degenerados empleados en la amplificación en cadena de la polimerasa (PCR) para identificar genes que codifican enzimas decarboxilasas dependientes de piridoxal. Estas enzimas están involucradas en la vía metabólica para la síntesis de alcaloides. Basados en la homología de secuencias reportadas en las bases de datos, se diseñaron cebadores degenerados para amplificar secuencias conservadas de estas enzimas en 18 familias de plantas diferentes. Se obtuvieron seis secuencias obtenidas de plantas del género Piper sp. Este reporte demuestra el potencial uso de ésta y otras metodologías actuales para aumentar el conocimiento, entendimiento y exploración del metabolismo secundario de las plantas de Colombia.
Palabras clave: bioprospección, metabolismo secundario, cebadores degenerados, microarreglos, decarboxilasas dependientes de piridoxal.
ABSTRACT
Plant secondary metabolites play an important role in plant-plant, plant-microorganisms and plant-insect interactions. They also protect the plants against stress environmental conditions. Plant secondary metabolites are also very important to humans due to their nutritional, pharmaceutical, medical and industrial properties. However, the secondary metabolism of tropical plant species still remains very poorly understood and characterised at the biochemical, molecular and genetic level. Within bioprospection programs to study the biodiversity of Colombian plants, the secondary metabolism is a very important target. Here we present an experimental methodology based on genomic DNA isolation from field collected plants, and the use of degenerate primers to PCR amplify genes that encodes pyridoxal-dependent enzymes which are involved in the alkaloids biosynthesis. Based on sequence homology we designed degenerate primers to
|
||
|
|
||
|
Ph. D. Dirección actual: Max Planck Institute for Plant Physiology. Am Muehlenberg 1. 14476 Golm-Alemania. Correo electrónico: soicalap@yahoo.com ** Ph. D. Molecular biotechnology unit. John Innes Center, Norwich NR4 7UH, UK.
Recibido: abril 14 de 2004. Aceptado: junio 1 de 2004.
|
||
|
|
||
|
67
|
||
|
|
||
|
|
|||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 67-77
|
|||
|
|
|||
|
amplify conserved gene sequences from 18 different plant families. Six putative tydc/tdc decarboxylases sequences were obtained from plants of the Piper genus. This report shows the usefulness of the DNA collection and PCR-based methodology e to increase the understanding and exploration of the secondary metabolism of Colombian plants.
Key words: Bioprospection, secondary metabolism, degenerate primer, microarrays, PLP-dependent decarboxylases.
|
|||
|
|
|||
|
INTRODUCCIÓN
Una de las principales consecuencias de la destrucción medioambiental actual es la extinción de un sinnúmero de especies de plantas. Esto representa una riqueza genética y biológica que se pierde para siempre. Colombia tiene el inventario en flora y fauna por unidad de área más grande del mundo (Instituto Alexander von Humboldt, Colombia). Dada la gran biodiversidad del país, la extinción de especies endémicas representa una amenaza particular. De las plantas de especies de la flora mundial que han sido clasificadas, cerca de 1000 están en peligro de extinción en Colombia, y muy probablemente muchas más especies sin descubrir aún, también están amenazadas. Es importante entonces, el desarrollo de políticas y programas de conservación y bioprospec-ción de nuestra diversidad floral, entre otras (Palacios Rojas, 2000). Uno de los campos de biopros-pección que se implementan a nivel mundial es la explotación del metabolismo secundario de las plantas. Iniciativas como Inbio en Costa Rica, buscan las sustancias activas de las plantas que tengan un uso potencial en la industria farmacéutica, industrial, cos-metológica o agrícola (Reid et al., 1993). Varios proyectos de gran alcance se desarrollan actualmente para estudiar la biodiversidad en países tropicales con el fin de identificar o aislar nuevos compuestos biológicamente activos. Actualmente su usan principalmente dos estrategias para explorar la biodiversidad: una de ellas es financiada por la industria y trata de identificar los compuestos activos de gran cantidad de plantas al mismo tiempo. De estos estudios comerciales a gran escala se puede derivar información para los estudios académicos en bioquímica, enzimología, genética molecular e ingeniería genética. La otra forma de explorar la biodiversidad es obteniendo la información preliminar de algunas plantas, estudiar su enzimología/bioquímica, con el fin de desarrollar estrategias para el mayor aprovechamiento de los productos de la misma, bien sean genes, enzimas o metabolitos (Palacios Rojas, 2000).
|
Los metabolitos secundarios son compuestos que, sin ser esenciales para la supervivencia de células individuales, tienen un papel importante en la vida y supervivencia del organismo en su entorno ecológico (Walton et al., 1999). Las plantas han desarrollado vías complejas para la biosíntesis de metabolitos secundarios. Dichos compuestos están involucrados en una multitud de funciones ecológicas entre ellas las interacciones planta-microorganismos, planta-insectos, planta-planta e interacciones planta-vertebrados. Igualmente, los metabolitos secundarios proveen protección contra condiciones abióti-cas adversas (Kutchan, 1995).
La identificación y el aislamiento de enzimas involucradas en la síntesis de metabolitos secundarios en plantas tropicales puede ser un primer paso en la bioprospección. Los genes que codifican estas enzimas pueden aislarse por medio de las secuencias de ADN altamente conservadas. Después de clonar el gen completo, las enzimas podrían expresarse he-terólogamente en bacterias o levaduras para su caracterización cinética y bioquímica. Así mismo, estas enzimas pueden ser usadas con el fin de incrementar la producción de metabolitos secundarios o generar mayor diversidad química en plantas, microorganismos o in vitro (Leech et al., 1998; Dong et al., 2001). Adicionalmente, el conocimiento de los genes y las secuencias completas, constituye una información valiosa en estudios filogenéticos de las especies tropicales, sobre todo para entender la evolución de estas rutas metabólicas.
En la actualidad se conocen más de 100000 compuestos secundarios (Goossens et al., 2003), los cuales son derivados de complejas rutas metabólicas y reacciones enzimáticas como hidroxilaciones, metila-ciones, acetilaciones y glicosilaciones. Dentro de dicha gama de compuestos están los alcaloides, un grupo grande y diverso de productos naturales encontrados en cerca del 20% de las especies de plantas (Kutchan, 1995). Los alcaloides son generalmente definidos por la presencia de un átomo de nitrógeno en estado
|
||
|
|
|||
|
68
|
|||
|
|
|||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
biología molecular para la bioprospecciÓn del metabolismo secundario de plantas
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
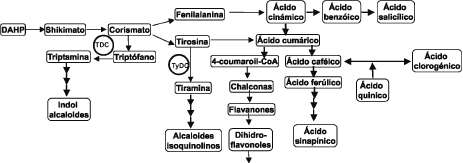
oxidado, dentro del anillo heterocíclico. De los aproximadamente 12000 alcaloides que se conocen de las plantas, muchos presentan actividades farmacológicas y varios son usados como farmacéuticos (Kutchan, 1995), es el caso de la vinblastina y la vincristina, producidos por Catharanthus roseus, y que se usan como anticancerígenos (Walton et al., 1999). Los alcaloides son derivados de intermediarios del metabolismo primario, siendo los aminoácidos los principales precursores. Por ejemplo, los alcaloides isoquinolinos, como morfina y berberina, son sintetizados a partir de tirosi-na; los alcaloides indol, como vinblastina, son derivados del triptófano; los alcaloides tropanos, como cocaína y escopolamina, son derivados de ornitina. Además de servir de precursores para la síntesis de alcaloides, los aminoácidos aromáticos también son los precursores para la síntesis de compuestos fenólicos (lignanos, flavonoides, ácidos hidroxicinámicos) a través de la vía del shikimato (Facchini y De Luca, 1995) (figura 1). Las enzimas decarboxilasas de aminoácidos aromáticos se encuentran en la interfase del metabolismo primario y secundario, jugan
|
compuestos mencionados. En este artículo se presentan resultados obtenidos utilizando plantas medicinales colombianas y la técnica de reacción en cadena de la polimerasa (PCR) con cebadores degenerados. Adi-cionalmente, se muestran algunas de las nuevas tecnologías y perspectivas de la investigación del metabolismo secundario en plantas y su bioprospección.
MATERIALES Y MÉTODOS
Selección de especies de plantas, muestreo y extracción de ADN. Las especies de plantas medicinales usadas en el presente trabajo se seleccionaron con base en reportes taxonómicos, etnobotánicos, fitoquímicos, farmacológicos y distribución geográfica. Se buscó que en la mayoría de las plantas analizadas se hubiera reportado la baja presencia y/o producción de alcaloides indol, isoquinolinos y ben-zilisoquinolinos (tabla 1).
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
do un papel primordial para la regulación y canalización del carbono a estas rutas. Dada la importancia eco
|
Tabla 1. Clasificación taxonómica de las plantas medicinales utilizadas
en el presente estudio. En negrilla se muestras las plantas utilizadas
como controles o referencias
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
lógica de los compuestos fenólicos y los alcaloides (indol e isoquinolinos), y el uso farmacéutico de estos últimos, este trabajo se enfoca en las enzimas Triptofano Decarboxilasa (TDC) y Tirosina Decarboxilasa (TyDC).
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
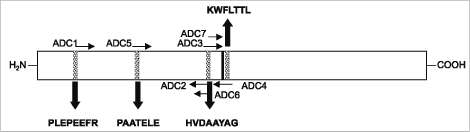
Para que las enzimas TDC y TyDC realicen la reacción de descar-boxilación, requieren del cofactor piri-doxal-5'-fosfato (Lehninger, 1981). Dentro del sitio de unión del piridoxal fosfato se encuentran unos residuos de histidina y prolina que están conservados en varias de las secuencias publicadas en las bases de datos ge-nómicas (Sandermann, 1992) (figura 2). Basados en estas regiones se diseñaron cebadores degenerados para clonar las isoenzimas de las especies tropicales. Para el desarrollo del presente trabajo se utilizó una estrategia basada en la reacción en cadena de la polimerasa (PCR) para la amplificación de enzimas decarboxilasas dependientes de piridoxal fosfatos, involucradas en la síntesis de los
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Fuente: Mabberley, 1997; Cronquist, 1981; Pérez Arbeláez, 1996.
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
69
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 67-77
|
|||
|
|
|||
|
El muestreo de las plantas fue realizado con la colaboración del Instituto Alexander von Humboldt (IVH, Colombia). La clasificación taxonómica se basó en la nomenclatura del código internacional de botánica, usando el sistema de clasificación establecido por Cronquist (1981). En campo se tomaron muestras para la clasificación taxonómica y se colectaron los datos generales de la planta y el medio ambiente en que se encontró.
Se buscaba desarrollar una metodología de colección de material vegetal aplicable en zonas geográficas de difícil acceso, y por tanto, el uso de nitrógeno líquido para conservar el tejido no se contempló por lo impráctico y costoso. Ensayos preliminares demostraron que la conservación del material para herbario no era la más adecuada para la obtención de ADN de calidad suficiente para amplificación por PCR. La metodología utilizada para la colección y preservación del material vegetal fue la siguiente: el muestreo en campo de las plantas para extracción de ADN se hizo utilizando tubos de polipropileno (15 mL) que contenían aproximadamente 5 g de sílica gel. Hojas jóvenes y visiblemente sanas fueron cortadas en pequeñas piezas y almacenadas en el tubo con silica gel, manteniendo una proporción 10:1 entre la sílica y el material vegetal. Se colectaron tres réplicas de cada muestra. Se monitoreó la desecación del material, 24 horas después de colectarlo. En caso de que ésta fuese incompleta, se adicionó más sílica gel al tubo. Las muestras fueron
|
almacenadas a temperatura ambiente hasta que fueron transportadas al laboratorio para la extracción de ADN. Una vez en sílica gel las muestras pueden conservarse por más de 12 meses.
Para la extracción de ADN las muestras (10-50 mg peso seco) fueron maceradas y mezcladas con 750 nl_ de 4X buffer CTAB (100 mM Tris.Cl pH 8,0; 1,4 M NaCl, 20 mM EDTA, 4% CTAB) a 65 °C conteniendo 0,1% p-mercaptoethanol (P-ME). Luego, las muestras fueron incubadas a 65 °C durante 1 h. Un volumen igual de clorofomo: alcohol isoamílico (24:1) fue adicionado y la emulsión se centrifugó a 10000 x g por 5 min a TA en una microcentrífuga de mesa normal. Luego, la fase acuosa fue transferida a un tubo nuevo y se repitió la extracción con clorofor-mo:alcohol isoamílico. Posteriormente, se trataron las muestras con RNAse (2 |ul a 10 mg/mL) y se incubaron a 37 °C por 30 min. El ADN fue precipitado adicionando 0,1 volúmenes de acetato de sodio 3 M y 2 volúmenes de etanol frío al 100%, seguido de una incubación toda la noche a -20 °C. Las muestras se centrifugaron a 8000 x g por 15 min a 4 °C. El pellet fue lavado con etanol al 70%, secado al aire y disuelto en 75 |jL de buffer TE. Para determinar la calidad y cantidad de ADN obtenido se corrieron las muestras en electroforesis de geles de agarosa y se midió la concentración por espectofotometría. El ADN fue almacenado a -20 °C.
|
||
|
|
|||
 |
|||
|
|
|||
|
[Antocianinas]
|
|||
|
|
|||
|
Figura 1. Enzimas decarboxilasas de aminoácidos aromáticos y su papel en la síntesis de alcaloides por la vía del shikimato. Abreviaturas: DAHP: 3-deoxy-D-arabino heptulosonato 7-fosfato; TDC: Triptófano decarboxilasa; TyDC: Tirosina decarboxilasa. Adaptado de Palacios-Rojas, 2000.
|
|||
|
|
|||
|
70
|
|||
|
|
|||
|
|
||||||||||||||||||||||||||||||||||||||||||
|
biología molecular para la bioprospecciÓn del metabolismo secundario de plantas
|
||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||
|
Tabla 2. Secuencias de cebadores degenerados usados
para el screening por PCR de secuencias de enzimas
decarboxilasas de aminoácidos aromáticos. Ver posición
de los cebadores en la figura 2
|
amplificación (94 °C, 45 s; 50 °C, 1 min; 72 °C, 1 min) y un paso final de extensión a 72 °C por 4 min. Los productos de PCR fueron separados por elec-troforesis en geles de agarosa y visualizados con bromuro de etidio bajo lámpara ultravioleta (Sam-brooket al., 1989).
RT-PCR con cebadores degenerados. Los cebadores degenerados fueron diseñados con base en la regiones conservadas de las secuencias de aminoácidos, incluyendo la región conservada de unión del fosfato piridoxal (HVDAAYAG), de diferentes enzimas decarboxilasas (figura 2 y tabla 2). Se usaron diferentes combinaciones de los cebadores ADC1-7 para la amplificación por PCR del cADN obtenido de plantas control: hojas de plantas de perejil (Petroseli-num crispum) de 20 días de edad; hojas de plantas de arveja (Pisum sativum) de un mes de edad; raíces, tallos y hojas de plantas de tabaco (Nicotiana ta-bacum) de un mes de edad; frutos maduros de tomate (Lycopersicum esculentum); hojas de plantas de Catharanthus roseus (medicinal anticarcinogénico) de un mes de edad, y hojas de plantas de amapola de 2 meses de edad. Estas plantas control se utilizaron dada la presencia reportada en bases de datos, de genes que codifican enzimas decarboxilasas dependientes de piridoxal. Se usaron templetas de cADN de hojas de Loniera tatarica y de hojas de trigo como controles negativos (no presencia reportada de genes que codifican enzimas decarboxilasas dependientes de piridoxal). Los cADN fueron sintetizados utilizando el kit de promega Sistema de transcripción reverda II, y siguiendo las instrucciones de la compañía.
|
|||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||
|
* Los nucleótidos en las posiciones degeneradas están representados por código de letras: R, A o G; W, AoT; Y, CoT; K, G o T; S, G o C; H, A, C o T; D, A, G o T; N, A, C, G o T.
|
||||||||||||||||||||||||||||||||||||||||||
|
Con el fin de evaluar la calidad del ADN extraído y su potencial uso para amplificación por PCR, se amplificaron las muestras usando cebadores específicos para las secuencias internas transcritas, espaciadores internos transcritos (ITS) de los genes ribosomales de ARN (Scout y Playford, 1996). La amplificación por PCR fue hecha en un volumen final de 20 uL, incluyendo 2 uL de buffer de reacción 10 X (Perkin Elmer), 1,5 mM de MgCl2, 200 |jJv1 dNTP, 2 |jJv1 de cada cebador (ITS3 cebador 1:5'-GCATCGATGAAGAACGCAGC-3' e ITS4 cebador 2:5'-TCCTCCGCTTATTGATATGC-3'), 1 U de Ampli-Taq DNA polimerasa (Perkin Elmer) y 50 ng ADN. El perfil de amplificación usado fue: de-naturación a 94 °C, 2 min, seguida por 35 ciclos de
|
||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||
 |
||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||
|
Figura 2. Representación esquemática de las secuencias consenso de las enzimas decarboxilasas de aminoácidos aromáticos de plantas registradas en las bases de datos públicas. En negrilla: residuos de aminoácidos conservados. Localización de los cebadores degenerados utilizados (ADC1-ADC7) para la amplificación en cadena de la polimerasa. La línea continua indica los residuos de prolina e histidina que forman parte del sitio de unión del cofactor fosfato de piridoxal.
|
||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||
|
71
|
||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||
|
|
||||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 67-77
|
||||
|
|
||||
|
Una vez que las condiciones de PCR fueron establecidas usando como templeta el cADN de las plantas control, se realizó la amplificación por PCR del ADN aislado de las plantas colombianas.
Hibridación de productos de PCR. Con el fin de
corroborar la especificidad de los productos de amplificación obtenidos por PCR, los fragmentos fueron separados en geles de agarosa al 1% para después ser transferidos por capilaridad a membranas de Nylon H+ (Amersham) e hibridados con una sonda derivada de un fragmento del gen tdc de C. roseus o con un fragmento del gen tydc de perejil.
Los filtros fueron prehibridados durante 2 h en sulfato de dextrano al 10%, SET 4X (NaCl 3M, EDTA pH 8 20 mM, Tris- HCl pH 8 0,6 M, tetrasodio pirofos-fato 11 mM), solución de Denhardt 10 X y SDS al 0,1%. Luego, la sonda radiactiva marcada por la técnica de anillamiento al azar fue adicionada (Kit Amersham). Tanto las condiciones de hibridación como de lavado fueron de baja astringencia, debido a que se utilizó una sonda heteróloga: un fragmento del gen tdc de C. roseus. Los filtros fueron hibridados toda la noche a 55 C. Posteriormente los filtros fueron lavados dos veces con 2 X SSC, 0,5% SDS y una vez con 0,5X SSC, 0,5% SDS, cada lavado fue
 |
hecho por 20 min. La membrana fue expuesta a películas de rayos X a -80 °C.
Clonación y secuenciación. La metodología de clonación y aislamiento de plásmidos se realizó de acuerdo con protocolos básicos de biología molecular (Sambrook et al., 1989). Los fragmentos de PCR que por hibridación con sondas heterólogas daban señal, fueron purificados y clonados en el vector pGEMt de promega, siguiendo las instrucciones de la casa comercial. Los plásmidos recombi-nantes fueron secuenciados usando el kit de ABI PRISM (Perkin Elmer). Los cebadores universales SP6 y T7 fueron usados para este propósito. Las secuencias obtenidas fueron comparadas con bases de datos públicas como NCBI, Swissprot, TAIR yTIGR.
RESULTADOS
Mantenimiento de plantas colectadas en campo y extracción de ADN. El material vegetal fue colectado en campo utilizando sílica gel para secarlo y mantenerlo antes de la extracción de ADN. Previamente se evaluaron varias metodologías de extracción de ADN, siendo la reportada aquí con la que se obtuvo mejor calidad de ADN. Con el fin de asegurar la calidad del ADN para amplificación por PCR, se amplificaron secuencias ITS (Internal Trascribed Spacer) en un 98% de las muestras obtenidas (Datos no mostrados). Tanto el tratamiento con sílica gel como la metodología utilizada para extraer el ADN genómico mostraron ser prácticos y adecuados, dado que se logró obtener ADN de calidad suficiente para análisis moleculares basados en la técnica de PCR.
|
|||
|
|
||||
|
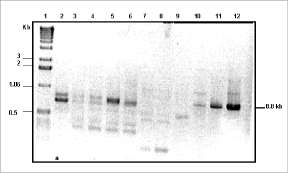
Figura 3. Productos de amplificación obtenidos por RT-PCR usando cebadores degenerados para decarboxilasas de aminoácidos aromáticos (ADC1/ADC6), en plantas de referencia. 1: marcador de peso molecular; 2: arveja (hojas); 3: tabaco (raíz); 4: tabaco (hojas); 5: tomate (hojas); 6: tomate (fruto); 7: control negativo: L. tatarica (hojas); 8: control negativo: L. tatarica (hojas); 9: trigo; 10: perejil (hojas); 11: amapola (hojas); 12: C. roseus. El tamaño esperado del producto de amplificación es 0,8 kb.
|
Detección por RT-PCR de enzimas decarboxilasas dependientes de piridoxal. La homología existente entre las enzimas decarboxilasas de aminoácidos aromáticos dependientes de fosfato de piridoxal fue usada para diseñar cebadores degenerados que se utilizaron en el screening por RT-PCR de plantas medicinales colombianas. Plantas control o referencia de las cuales se conoce tienen las enzimas presentes, fueron usadas
|
|||
|
|
||||
|
72
|
||||
|
|
||||
|
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
biología molecular para la bioprospecciÓn del metabolismo secundario de plantas
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
para estandarizar las condiciones de amplificación. Usando la combinación de los cebadores ADC1 y ADC6, se obtuvieron los mejores productos de amplificación en las plantas de referencia, al usarse 50 °C de temperatura de anillaje y 2 uM de cada cebador (figura 3). Los fragmentos principales fueron clonados y secuenciados, corroborándose la identidad de las enzimas decarboxilasas de aminoácidos aromáticos en todas las muestras usadas a excepción de las hojas de arveja. Sin embargo, al utilizar cADN de hojas de arveja como templeta para el PCR se obtuvieron dos productos de amplificación que pueden considerarse inespecíficos dado que al clonarlos y secuenciarlos no se encontró identidad alguna con enzimas decarboxilasas. Adicionalmen-te, cabe la posibilidad de que el gen que codifica para TDC en arveja no se exprese en el tejido muestreado.
|
DISCUSIÓN
Estudios moleculares del metabolismo secundario de las plantas. En términos moleculares y bioquímicos, la biodiversidad puede considerarse como un bellísimo tesoro el cual puede comprender muchos compuestos nuevos, enzimas y genes "útiles" para los humanos. Es claro que para poder explorar la biodiversidad es necesario, ante todo, el desarrollo y la aplicación de verdaderas políticas de conservación.
El presente trabajo muestra una de las alternativas básicas para explorar la diversidad floral colombiana, en términos del metabolismo secundario. La metodología de colección de material vegetal en campo probó ser de gran utilidad, práctica y económica, comparada con el uso de nitrógeno líquido, secado de muestras en papel o preparación química para muestras de herbario. El uso de sílica gel para secar las muestras en campo es una metodología fácil y práctica cuando se trata de colectar material en zonas de difícil acceso o cuando la colecta requiere el trabajo de varios días consecutivos en campo, sin acceso a nitrógeno líquido o congeladores de -80 °C para garantizar la preservación de los tejidos vegetales. Trabajos preliminares no descritos aquí revelaron gran dificultad para obtener ADN genómico de calidad suficiente para PCR, a partir de material secado por técnicas químicas de conservación y secado de material para herbario.
Del total de 14 familias de plantas utilzadas en el presente estudio, sólo en miembros de la familia Pipe-raceae se obtuvieron productos de PCR, que al clonarlos y secuenciarlos mostraron homología con genes que codifican enzimas decarboxilasas dependientes de piridoxal. Es importante tener en cuenta que, aunque se obtuvieron productos de amplificación al utilizar como templeta ADN genómico de otras plantas, éstos productos fueron inespecíficos (figura 4). Dada las características de baja astringencia del PCR con primers degenerados, es importante asegurarse de la naturaleza del producto amplificado. Para tal propósito, en el presente trabajo, la hibridación de los productos de PCR con zonas conservadas de los genes de interés, se utilizo como procedimiento rápido y menos costoso. Así, sólo aquellos productos que hibridaron fueron clonados y secuenciados.
La familia Piperaceae incluye los géneros Piper, Peperomia y Trianaeopiper. Este último es endémico de Colombia y en general los tres géneros están
|
||||||||||||||||||||||||||||||||||||||||||||||
|
Tabla 3. Identidad de secuencias obtenidas a partir de plantas colombianas del género Piper sp.
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
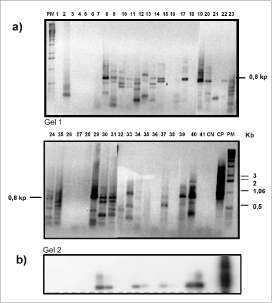
Usando las condiciones de amplificación establecidas con las plantas de referencia, se amplificó el ADN genómico de las diferentes especies de plantas colombianas (figura 4a). Se observaron productos de amplificación del tamaño esperado (800 pares de bases). Sin embargo, sólo las bandas que mostraron señal al hibridarse a baja astringencia con sondas de tdc de C. roseus o de tydc de perejil, fueron clonadas y secuenciadas (figura 4b). Seis de los 7 clones secuenciados presentaron niveles de identidad entre 60-69% con tydc de amapola y de perejil (tabla 3). La identidad con secuencias de tdcfue entre 58 y 66%. Los seis clones putativos relacionados con tydc se obtuvieron de plantas pertenecientes al género Pipersp.
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
73
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 67-77
|
||||
|
|
||||
 |
(AFLP, dato no mostrado), es posible realizar estudios de distribución de la diversidad, los cuales pueden ser útiles en el diseño de programas de conservación. Adicionalmente, el género Piper es usado medicinalmente de diferentes formas; entre los compuestos medicinalmente activos que han sido aislados se encuentran alcaloides, lignanos, terpenos, esteroi-des, charcones y flacones (Parmar et al., 1997). Así, plantas de este género son blancos potenciales para mayores investigaciones bioquímicas, enzimológicas y moleculares de las diferentes vías del metabolismo secundario presentes en estas plantas.
La clonación de fragmentos de genes específicos puede ser usada para estudios fundamentales de expresión génica espacio-temporal, abriéndose así oportunidades de investigación de la biosíntesis de alcaloides bencilsioquinolinos en Piper sp. Los fragmentos clonados se pueden utilizar para obtener las secuencias completas de los genes, utilizando metodologías moleculares como 5'-RACE y 3'-RACE (Rapid am-plification of cDNA ends), y posteriormente sobre expresarlas en el sistema apropiado (bacteria, levadura, baculovirus), con el fin de purificar y caracterizar la proteína in vi-tro, especialmente en cuanto a la especificidad de sustrato. En consecuencia, aquellas enzimas clonadas que puedan utilizar diferentes sustratos podrían ser introducidas en plantas o en cultivos celulares y posteriormente alimentarlas o agregarles los sustratos o análogos de sustratos para que sean convertidos en otras sustancias útiles y deseadas. Esta estrategia puede permitir sintetizar nuevas drogas y compuestos medicinales que por medio de los reactores químicos tradicionales no son sintetizables.
|
|||
|
Figura 4. Screening de genes codificantes de enzimas decarboxilasas de aminoácidos aromáticos en ADN genómico de plantas colombianas. Panel a. Amplificación por PCR usando los cebadores degenerados ADC1/ADC6, separados en gel de agarosa al 1%. Panel b. Hibridación de los productos de PCR (gel 2) con sonda de tdc de C. roseus.
1: Piperaduncum; 2: Siparunasp; 3: Ocotea aurantiadora; 4: Pipersp; 5: Pepe-romia sp; 6: Pipersp; 7: Ladenbergia magnifolia; 8: Pothomorphe umbellata; 9: Cinnamomum cinnamomifolica; 10: Anacardium occidentale; 11: Spondias sp; 12: Toxicodendrum striatum; 13: Annona muricata; 14: Anacardium excelsum; 15: Tecoma stans; 16: Aristolochia sp; 17: Palicourea sp; 18: Boccocia sp, 19: Psycotria sp; 20: Trichanthera gigantea; 21: Croton sp; 22: Annona squamulo-sa; 23: Pothomorphe peltata; 24: Trianaeopiper sp; 25: Zanthoxylum fagara; 26: Zanthoxylum rhoifolia; 27: Zanthoxylum monophylum; 28: Zanthoxylum sp; 29: Siparuna sp, 30: Pipersp, 31: Pipersp; 32: Zanthoxylum sp; 33: Pipersp; 34: Pipersp; 35: Psycotria sp; 36: Psycotria poeppigiana; 37: Pipersp; 38: Tria-nopipersp; 39: Pipersp; 40: Pipersp; 41: Crescentia cujete; CN: agua; CP: Clon de tdcde C. roseus; PM: marcador de peso molecular. Los productos de PCR de plantas en rojo fueron clonados y secuenciados.
|
||||
|
|
||||
|
ampliamente distribuidos por todo el país (Mabberley, 1997; Palacios Rojas, 2000). El aislamiento de ADN de plantas del género Pipersp. y dada la calidad del mismo para uso en técnicas por PCR, incluyendo polimorfismo en la longitud de los fragmentos amplificados
|
La viabilidad de intercambiar enzimas del metabolismo secundario entre especies separadas por largas distancias evolutivas ha sido ejemplarmente demostrada en varios estudios. Por ejemplo, Dong et al. (2001), trabajando en la síntesis de flavonoides en plantas demostraron que los productos de los genes C2 (chalcona sintasa), CHI1 (chalcona isomerasa) y
|
|||
|
|
||||
|
74
|
||||
|
|
||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
biología molecular para la bioprospecciÓn del metabolismo secundario de plantas
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
A1 (dihidroflavonol 4-reductasa) de maíz complementan las mutaciones tt4, tt5 y tt3 en Arabidopsis, restaurando la habilidad de estos mutantes para acumular pigmentos en cubierta de semillas y en plántulas. Por tanto, también podría ser posible la sobreexpresión de dichas enzimas en otras plantas, lo cual puede generar diferencias cuantitativas y cualitativas importantes en la producción de nuevos metabolitos a través de ingeniería genética (Dong et al., 2001;Leechetal., 1998).
Es evidente la necesidad de desarrollar un método eficiente para probar en masa las actividades catalíticas de las enzimas cuyas secuencias están siendo descubiertas en los proyectos de secuencia-ción. En el estudio del metabolismo secundario, sólo la demostración de la actividad enzimática puede identificar sin ambigüedades la función de la proteína (Pichersky y Gang, 2000).
Screening de genes que codifican enzimas del metabolismo secundario. Los proyectos de se-cuenciación han contribuido al descubrimiento de varias familias de genes, definidas por tener dominios comunes en las proteínas que codifican (los cuales pueden ser el sitio activo y/o dominios de sustratos y cofactores). Homólogos de varias enzimas involucradas en la síntesis de alcaloides han sido detectados en el genoma de Arabidopsis, a pesar de la aparente carencia de alcaloides complejos en esta planta. La mayoría de estos homólogos son genes putativos que codifican enzimas decorativas como hidroxilasas, metiltransferasas y acetiltransferasas (tabla 4) (Facchini et al., 2004).
|
Toda esta información de secuencias de otras plantas que está disponible puede usarse para el estudio de la diversidad y el metabolismo secundario de especies medicinales. El presente trabajo es un ejemplo de ello. Las secuencias similares a tdc/tydc fueron amplificadas de ADN genó-mico de plantas del género Pipersp., usando cebadores degenerados basados en las secuencias disponibles en las bases de datos. Aunque el uso de ADN presenta algunas limitaciones por la posible presencia de intrones en los genes motivo de estudio, lo más importante en la estrategia basada en PCR, reportada aquí, es el diseño de los cebadores.
El uso de cADN como templeta para la amplificación por PCR puede ser más atractivo por ser un templado menos complejo que el ADN genómi-co y permitir una mejor especificidad de la amplificación. Sin embargo, es importante tener en cuenta la necesidad de mezclar cADN de diferentes tejidos, órganos, estados de desarrollo y condiciones medioambientales, para asegurarse de que el ARN del gen esté presente en la muestra. Esta limitación se debe a la regulación específica y a la baja expresión de muchos de los genes involucrados en el metabolismo secundario (burlat et al., 2004; St-Pierre et al., 1999). Adicionalmente, el ARN es menos estable en las muestras disecadas y la metodología para aislar ARN también es un punto crítico que tiene que ser optimizado para cada especie.
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Tabla 4. Familias de genes de plantas con al menos un miembro involucrado en el metabolismo secundario
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
* No. de Arabidopsis. Modificado de Facchini et al., 2004.
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
75
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 67-77
|
|||
|
|
|||
|
Discusión sobre revisión bibliográfica
de metodologías moleculares para el estudio
del metabolismo secundario
Aplicación de nuevas metodologías y técnicas para la exploración del metabolismo secundario en plantas tropicales. Otras estrategias diferentes a la metodología basada en amplificación por PCR presentada en este estudio pueden usarse para explorar la biodiversidad y el metabolismo secundario. Para esto es importante tener en cuenta no sólo el conocimiento bioquímico, enzimológico, etnobotánico y/o fi-toquímico que se tenga de las plantas a estudiar, sino también el costo y la accesibilidad a las tecnologías requeridas para la preparación y análisis de las muestras. Así, en laboratorios y/o países donde la tecnología es limitante, los programas de inventarios, conservación de germoplasma, generación de bancos de ADN genómico, librerías de cADN, análisis fito-químicos de especies de plantas diferentes a las especies cultivadas son de gran importancia como base del conocimiento para el desarrollo de programas co-laborativos con industrias o institutos de investigación que tengan acceso a tecnologías analíticas más sofisticadas.
La técnica de polimorfismo en la longitud de los fragmentos amplificados de cADN (AFLP) constituye una alternativa atractiva para identificar genes involucrados en el metabolismo secundario de plantas. Con este método se obtienen perfiles de expresión, los cuales no requieren de información previa sobre la secuencia. Utilizando esta técnica y usando cADN obtenido de células de tabaco inducidas con metilo de jasmonato (Goossens et al., 2003) se obtuvo un amplio repertorio de genes conocidos y de genes nuevos los cuales están involucrados estructural o regulato-riamente en el metabolismo secundario en tabaco.
El uso de microarreglos heterólogos. Con el geno-ma de Arabidopsis completamente secuenciado, y con la creciente disponibilidad de las secuencias genómi-cas de otras especies de plantas, el reto es no sólo identificar el método por el cual mapas, secuencias y, eventualmente, información de genómica funcional de una especie pueda usarse y compararse, sino encontrar la forma de explotar dicha información a través de todas las especies de plantas, tanto en términos del metabolismo primario como del secundario. En los últimos años, el interés por cumplir dicho reto ha incrementado y desarrollado proyectos multidisciplinarios (biólogos, bioquímicos, bioinformáticos, fitoquímicos,
|
ecólogos, etc.) los cuales se ilustran claramente en literatura reciente. Por ejemplo, los microarreglos basados en EST (Expressed Sequenced Target), los cuales son herramientas muy poderosas para el descubrimiento de genes y estudios de transducción de señales en especies altamente caracterizadas como Arabidopsis, tomate, tabaco y arroz, han sido utilizados exitosamente en diferentes especies, incluyendo algunas no caracterizadas. Horvath et al. (2003) hi-bridaron el microarreglo 11 K de Arabidopsis con cADN de hojas maduras y plántulas de diferentes especies (Avena silvestre- Avena fatua, poplar- Populus deltoidsies, Euphorbia esula, Arabidopsis thaliana), detectando expresión del 23-47% de los genes y demostrando así que un gran número de genes de especies relativamente distantes pueden evaluarse con los microarreglos de Arabidopsis, especialmente genes involucrados en división celular, respuesta a estrés y desarrollo. La posibilidad de utilizar microarreglos heterólogos abre la oportunidad de estudiar más a fondo un sinnúmero de especies tropicales y colombianas.
El uso de un set de genes ortólogos conservados.
Fulton et al. (2002), por su parte, han identificado un set de genes conservados a través de la evolución en secuencia y número de copia. Este set de más de 1000 genes conservados, llamados set de marcadores ortólogos conservados (conserved ortholog set- COS) fueron identificados computacionalmente comparando la secuencia genómica de Arabidopsis con la base de datos de EST de tomate (13000 EST, mitad del contenido génico). Tomate y Arabidopsis pertenecen a diferentes familias (Solanaceae y Brassicaceae), las cuales divergieron muy temprano en la evolución de las plantas con flor, hace 100 a 150 mil años. Así, estos marcadores COS podrían usarse para otros estudios de mapeo comparativo y filogenético, y contribuir a elucidar la naturaleza de genes conservados a través de la evolución de las plantas tropicales.
CONCLUSIÓN
La diversidad floral colombiana guarda una gran oportunidad de exploración a nivel del metabolismo secundario, y con el avance científico-tecnológico mundial será cada vez más importante el estudio extensivo y coherente para explorar esta riqueza. Esta tarea de bioprospección y conservación debe ser hoy una prioridad, ya que la destrucción de las selvas y bosques tropicales de Colombia continúa acelerándose irremediablemente.
|
||
|
|
|||
|
76
|
|||
|
|
|||
|
|
|||
|
biología molecular para la bioprospecciÓn del metabolismo secundario de plantas
|
|||
|
|
|||
|
AGRADECIMIENTOS
Los autores agradecen a Andrés Giraldo y al doctor Cristian Samper, del Instituto Alexander von Hum-boldt (Colombia); a Camilo López y al doctor Joe Toh-me del Centro Internacional de Agricultura Tropical (CIAT, Colombia), y al doctor Paul Christou de la Unidad de Biotecnología del John Innes Centre (Inglaterra) por su colaboración para la realización de este trabajo. Agradecemos los valiosos comentarios sobre este manuscrito de los doctores Axel Tiessen y Wolf-gang Lein. Natalia Palacios fue apoyada económicamente por una beca-crédito doctoral del Instituto Colombiano de Ciencia y Tecnología "Francisco José de Caldas" - Colciencias. El centro de investigación John Innes en Norwich, Inglaterra, apoyó económicamente parte de la realización del presente trabajo.
BIBLIOGRAFÍA
Bohlmann, J.; Meyer, G.; Croteau, R. 1998. Plant terpenoid synthases: molecular biology and phylogenetic analy-sis. Proc. Natl. Acad. Sci. 95: 4126-4133.
Burlat, V.; Oudin, A.; Courtois, M.; Rideau, M.; St-Pierre, B. 2004. Co-expression of three MEP-pathway genes and geraniol-10- hydoxylase in internal phloem parenchyma of Catharanthus roseus implicates multicellular trans-location of intermediates during the biosynthesis of monoterpenes indol alkaloids and isoprenoid-derived primary metabolites. Plant Journal. 38: 131-141.
Cronquist A., 1981. An integrated system of classification of flowering plants. New York: Columbia University Press.
Dong,X.; Braun, E.; Grotewold, E. 2001. Functional conserva-tion of plant secondary metabolic enzymes revealed by complementation of Arabidopsis flavonoid mutants with maize genes. Plant physiology. 127: 46-57.
Facchini, P.; De Luca, V. 1995. Expression in Escherichia coli and partial characterization of the two tyrosine/DOPA decarboxylases from Opium poppy. Phytochemistry. 38: 1119-1126.
Facchini, P.; Bird, D.; St-Pierre, B. 2004. Can Arabidopsis make complex alkaloids? Trends in plant science 9: 116-122.
Fulton, T.; Van der Hoeven, R.; Eannetta, N.; Tanksley, S. 2002. Identification, analysis, and utilization of con-served ortholog set markers for compartive genomics in higher plants. The Plant Cell. 14: 1457-1467.
Goossenes, A.; Häkkinen, S.; Laakso, I.; Seppanen, T.; Biodi, S.; De Sutter, V.; Lammertyn, F.; Nuutila, A.; Söderlund, H.; Zabeau, M.; Oksman-Caldentay, K. 2003. A functional genomics approach toward the understanding of
|
secondary metabolism in plant cells. Proc. Natl. Acad. Sci. 10:8595-8600.
Gupta, M. 1995. 270 plantas medicinales iberoamericanas. Convenio Andrés Bello. 617 p.
Horvarth, D.; Schaffer, R.; West, M.; Wilsman, E. 2003. Arabidopsis microarrays identify conserved and differ-entially expressed genes involved in shoot growth and development from distantly related plant species. The Plant Journal. 34: 125-134.
Kutchan, T. 1995. Alkaloid biosynthesis: the basis for metabolic engineering of medicinal plants. The Plant Cell. 7: 1059-1070.
Leech, M.; May, K.; Hallard, D.; Verpoorte, R.; De Luca, V.; Christou, P. 1998. Expression of two consecutive genes of secondary metabolic pathway in transgenic tobacco: molecular diversity influences levels of expression and productaccumulation. Plant Mol. Biol. 38: 765-774.
Lehninger, A. 1981. Biochemistry. New York: Worth publishers.
Mabberley, D. J. 1997. The plant-book. A portable dictionary of the vascular plants. 2and ed., Cambridge: Cambridge University Press.
Palacios Rojas, N. 2000. Metabolismo secundario en plantas: screening molecular de la biodiversidad en flora y estudios de rutas biosintéticas en Lonicera tatarica y Catharanthus roseus. Tesis doctoral, University of East Anglia-John Innes Center, Norwich, UK.
Parmar, V. J.; Bisht, K.; Jain, R.; Taneja, P.; Jha, A.; Tyagi, O.; Prassad, A.; Wengel, J.; Olsen, C.; Boll, P. 1997. Phytochemistry of the genus Piper. Phytochemistry46:597-673.
Pérez Arbeláez, E. 1996. Plantas útiles de Colombia. 5 edición. Bogotá: Fondo FEN.
Pichersky, E.; Gand, D. 2000. Genetics and biochemistry of secondary metabolites in plants: an evolutionary per-spective. Trends in plant science. 5: 439-445.
Reid, W.; Laird, S.; Meyer, C.; Gámez, R.; Sittenfeld, A.; Janzen, D.; Gollin, D.; Juma, C. 1993. La prospección de la biodiversidad: el uso de los recursos genéticos para el desarrollo sostenible. Costa Rica: World resources institute.
Sambrook, J.; Fritsch, E.; Manniatis, T. 1989. Molecular clon-ing: a laboratory manual. New York: Cold spring Harbor.
Sandermann, H. 1992. Plant metabolism of xenobiotics. TIBS 17: 82-84.
Scott K.; Playford J. 1996. DNA extraction technique for PCR in rain forest plant species. Biotechniques Euro Edition 20-21.
St-Pierre, B.; Vásquez-Flota, F.; Peluca, V. 1999. Multicellular compartmentation of Catharanthus roseus alkaloid biosynthesis predicts intercellular translocation of a pathway intermediate. Plant Cell 11: 887-900.
Walton, N.; Rhodes, M.; Michael, A.; Parr, A. 1999. Charac-terisation and control of plant secondary metabolism. Walton y Brown, eds. In: Chemicals from plants. Lon-don: Imperial College Press, pp. 27-90.
|
||
|
|
|||
|
77
|
|||
|
|
|||
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2004 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).


















