Primer reporte de un begomovirus presente en maracuyá amarillo [Passiflora edulis f. flavicarpa (Degener)] en Valle del Cauca, Colombia
First report of a begomovirus presents in yellow passionfruit [Passiflora edulis f. flavicarpa (Degene)] in Valle del Cauca, Colombia
DOI:
https://doi.org/10.15446/rev.colomb.biote.v18n2.52904Palabras clave:
Geminivirus, Passiflora edulis f. flavicarpa, PSLDV. (es)Geminivirus, Passiflora edulis f. flavicarpa, PSLDV (en)
El maracuyá amarillo es un cultivo de importancia económica para el Valle del Cauca, pero en los últimos años ha presentado una reducción de hasta un 80% en la producción debido a problemas virales. El objetivo de este estudio fue detectar y caracterizar molecularmente un Begomovirus que está afectando cultivos de maracuyá amarillo localizados en dos zonas del Valle del Cauca, La Unión y Palmira. Se colectaron hojas de maracuyá con síntomas típicos de enfermedad viral, se purificó su DNA genómico y se identificó la presencia de Begomovirus bipartitas mediante PCR empleando cebadores degenerados. Cuatro genomas virales parciales obtenidos de muestras vegetales colectadas en La Unión y Palmira fueron clonados, secuenciados y analizados. El análisis de secuencia del Begomovirus que está afectando maracuyá amarillo en Colombia con otros reportados en la base de datos, indica que éste es un Begomovirus bipartita nuevo que tiene una relación cercana con un Begomovirus aislado de frijol y no tiene ninguna relación con Passionfruit severe leaf distortion virus, un Begomovirus reportado previamente afectando maracuyá amarillo en Brasil. Para nuestro conocimiento, este es el primer reporte de un nuevo Begomovirus bipartita que afecta maracuyá amarillo en Colombia y es diferente de otros Begomovirus previamente reportados a nivel mundial afectando este cultivo.
The yellow passion fruit is a crop of economic importance to the Valle del Cauca, but in recent years has been a reduction of up to 80% in production due to viral problems. The aim of this study was to detect and characterize partially begomovirus that could be affecting crops of yellow passionfruit located in two growing areas of the Valle del Cauca (La Union and Palmira). Passionfruit leaves with typical symptoms of viral disease were collected, purified genomic DNA and its bipartite begomovirus were detected by PCR using primers specific for component A and B, respectively. Five DNA fragments corresponding to geminiviral A and B genome were cloned, sequenced and analyzed with bioinformatics tools. The results show for the first time in Colombia the presence of a begomovirus in yellow passionfruit. The analysis of the nucleotide sequences indicates that this begomovirus is more related to bean BDMV, a geminivirus affecting beans in Colombia, not PSLDV, which was reported affecting yellow passionfruit in Brazil. To date, this result would be the first report of a begomovirus (belonging to the family Geminiviridae) affecting yellow passion fruit in Colombia, and that might be different from other begomovirus previously reported worldwide affecting this crop.
Key words: Geminivirus; Passiflora edulis f. flavicarpa; PSLDV.
DOI: https://doi.org/10.15446/rev.colomb.biote.v18n2.52904
ARTÍCULO DE INVESTIGACIÓN
Primer reporte de un begomovirus presente en maracuyá amarillo [Passiflora edulis f. flavicarpa (Degener)] en Valle del Cauca, Colombia
First report of a begomovirus presents in yellow passionfruit [Passiflora edulis f. flavicarpa (Degene)] in Valle del Cauca, Colombia
Juan Carlos Vaca-Vaca*, Emerson Clovis Carrasco-Lozano**, Mónica Rodríguez-Rodríguez***, Jhon Fredy Betancur-Perez****, Karina López-López*****
* Ph.D, Biotecnología de Plantas; MSc. Microbiología con Énfasis en Biotecnología. Departamento de Ciencias Agrícolas, Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia. AA 237. Palmira, Valle del Cauca, Colombia. Autor para correspondencia, Email:
jcvacava@unal.edu.co
** MSc. Ciencias Biológicas. Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia. AA 237. Palmira, Valle del Cauca, Colombia. Dirección Actual: Facultad de Agronomía, Universidad Nacional del Centro del Perú, Perú.
*** Ingeniera Agrónoma. Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia sede Palmira, Colombia
**** Doctor en Ciencias Agrarias. Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia. AA 237. Palmira, Valle del Cauca, Colombia. Dirección Actual: Facultad de Ciencias de la Salud, Universidad de Manizales, Manizales, Caldas.
***** Ph.D, Biotecnología de Plantas. Departamento de Ciencias Biológicas, Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia. AA 237. Palmira, Valle del Cauca, Colombia.
Recibido: marzo 10 de 2016 Aprobado: octubre 24 de 2016
Resumen
El maracuyá amarillo es un cultivo de importancia económica para el Valle del Cauca, pero en los últimos años ha presentado una reducción de hasta un 80% en la producción debido a problemas virales. El objetivo del presente trabajo fue detectar y caracterizar parcialmente begomovirus que podrían estar afectando los cultivos de maracuyá amarillo localizados en dos zonas productoras del Valle del Cauca (La Unión y Palmira). Se colectaron hojas de maracuyá con síntomas típicos de enfermedad viral, se purificó su DNA genómico y se detectó la presencia de begomovirus bipartitas mediante PCR empleando cebadores específicos para el componente A y B, respectivamente. Cinco fragmentos de DNA correspondientes al genoma geminiviral A y B, fueron clonados, secuenciados y analizados con herramientas bioinformáticas. Los resultados evidencian por primera vez en Colombia de la presencia de un begomovirus en maracuyá amarillo. El análisis de la secuencias de nucleótidos indica que este begomovirus está más relacionado con el virus del mosaico enano del frijol, un geminivirus que afecta frijol en Colombia, y no con el virus de la distorsión severa de la hoja de maracuyá, el cual se reportó afectando maracuyá amarillo en Brasil. A la fecha, este resultado constituiría el primer reporte de un begomovirus (perteneciente a la familia Geminiviridae) que afecta maracuyá amarillo en Colombia y que podría ser diferente de otros begomovirus previamente reportados a nivel mundial afectando este cultivo.
Palabras clave: Geminivirus, Passiflora edulis f. flavicarpa, PSLDV.
Abstract
The yellow passion fruit is a crop of economic importance to the Valle del Cauca, but in recent years has been a reduction of up to 80% in production due to viral problems. The aim of this study was to detect and characterize partially begomovirus that could be affecting crops of yellow passionfruit located in two growing areas of the Valle del Cauca (La Union and Palmira). Passionfruit leaves with typical symptoms of viral disease were collected, purified genomic DNA and its bipartite begomovirus were detected by PCR using primers specific for component A and B, respectively. Five DNA fragments corresponding to geminiviral A and B genome were cloned, sequenced and analyzed with bioinformatics tools. The results show for the first time in Colombia the presence of a begomovirus in yellow passionfruit. The analysis of the nucleotide sequences indicates that this begomovirus is more related to bean BDMV, a geminivirus affecting beans in Colombia, not PSLDV, which was reported affecting yellow passionfruit in Brazil. To date, this result would be the first report of a begomovirus (belonging to the family Geminiviridae) affecting yellow passion fruit in Colombia, and that might be different from other begomovirus previously reported worldwide affecting this crop.
Key words: Geminivirus, Passiflora edulis f. flavicarpa, PSLDV.
Introducción
La familia Passifloraceae presenta 18 géneros y alrededor de 660 especies (Holm-Nielsen et al., 1988). Los principales productores a nivel mundial son Brasil, Ecuador, Colombia y Perú con aproximadamente 745 000 t/año (Passionfruit, 2015). En Colombia, el género Passiflora presenta 170 especies (Ocampo et al., 2007) de los cuales, el maracuyá amarillo [Passiflora edulis f. flavicarpa (Degener)], es la especie más importante, debido a que sus frutos son comercializados como fruta fresca y procesada en mercados nacionales e internacionales (Ulmer & MacDougal, 2004). El Sistema de Estadísticas Agropecuarias–SEA (AGRONET, 2015), reportó una producción del cultivo de maracuyá de 95,152.2 toneladas y un área cultivada de 5,788.5 hectáreas para el año 2013, donde el departamento de Huila contribuyó con el 21.9% de la producción, seguido por el departamento de Meta con el 19.4% y el Valle del Cauca con 17.3%. El cultivo de maracuyá amarillo es afectado por diferentes enfermedades que disminuyen su producción y calidad, siendo las enfermedades virales una de las principales limitantes que están causando grandes pérdidas a los productores. Se estima que en el Valle del Cauca está ocasionando la disminución del rendimiento hasta en un 50% y cuando el cultivo alcanza una incidencia del virus entre el 30 y el 50% se afecta mucho la calidad del fruto causando pérdidas económicas que llegan a un 70% (Pérez, 2009)1. En los últimos años, en el Valle del Cauca se han evidenciado altas poblaciones de mosca blanca Bemisia tabaci (Gennadius) biotipo B asociadas a cultivos agrícolas como el tomate, ají, pimentón y maracuyá (Rodríguez et al., 2012). B. tabaci biotipo B es el vector biológico de Begomovirus, un género viral de la familia Geminiviridae, que tiene un genoma circular de DNA cadena sencilla y que constituye el grupo más importante de fitopatógenos que están causando pérdidas significativas en diferentes cultivos localizados tanto en ecosistemas tropicales como subtropicales a nivel mundial (Rojas et al., 2005; Seal et al., 2006). Los Begomovirus afectan plantas monocotiledóneas y dicotiledóneas, representan el género viral con mayor número de especies reportadas a la fecha, con 288 especies (ICTV, 2015), y su genoma puede ser monopartita o bipartita. El genoma de los begomovirus bipartitas está compuesto por dos moléculas de DNA con un tamaño aproximado de 2.6kb donde cada uno de los componentes genómicos se denomina DNA-A y DNA-B, respectivamente. El DNA-A contiene los marcos de lectura abiertos que codifican para proteínas necesarias para la replicación, la transactivación y la encapsidación viral, mientras que el DNA-B contiene los marcos de lectura abiertos que codifican para proteínas necesarias para el movimiento del virus a corta y larga distancia dentro de la planta (Rojas et al., 2005).
En Colombia, se han identificado dos virus afectando el cultivo de maracuyá amarillo: un potyvirus, cuya identidad fue superior al 97% con aislamientos del virus del mosaico de la soya (SMV) y un tymovirus, idéntico al virus del mosaico amarillo del maracuyá descrito en Brasil (Morales et al., 2001). A la fecha, Brasil es el único país que ha reportado begomovirus afectando el cultivo de maracuyá amarillo. Éste se identificó y caracterizó a nivel molecular y se denominó virus de la distorsión severa de la hoja de maracuyá (PSLDV, por sus siglas en Inglés), basado en los síntomas que ocasiona en maracuyá: mosaico amarillo en hoja, reducción severa de la lámina foliar y del crecimiento vegetal, reducción en el número y tamaño de frutos, y malformación de frutos (Novaes et al., 2003; Ferreira et al., 2010).
Debido a que existía un reporte previo de un geminivirus (PSLDV) afectando el cultivo de maracuyá en Brasil y el hecho de que las poblaciones de mosca blanca en el Valle del Cauca así como en otros departamentos de Colombia están en aumento, surgió la inquietud de examinar los cultivos de maracuyá en esta región del país en busca de la presencia o no de geminivirus. Por tanto, el objetivo del presente trabajo fue detectar y caracterizar parcialmente begomovirus que podrían estar afectando los cultivos de maracuyá amarillo localizados en dos zonas productoras del Valle del Cauca (La Unión y Palmira). Los resultados obtenidos en esta investigación evidencian por vez primera en Colombia de la presencia de un begomovirus en maracuyá, el cuál podría ser diferente del geminivirus (PSLDV) previamente reportado afectando éste cultivo en Brasil.
Materiales y métodos
Colecta de material vegetal. La recolección de material vegetal se realizó en dos cultivos comerciales de maracuyá amarillo Passiflora edulis f. flavicarpa (Degener) ubicados en los municipios de La Unión y Palmira, en el Departamento del Valle del Cauca entre los años 2010-2014. El cultivo se encontraba en etapa de floración con una edad aproximada de 5.5 meses de establecido. El muestreo en campo se llevó a cabo mediante la recolección de hojas jóvenes de maracuyá que presentaban síntomas típicos de la enfermedad geminiviral tales como: enanismo, mosaicos amarillo brillante, moteados cloróticos, clorosis foliar marginal, enrollamiento foliar, deformaciones foliares y arrugamientos de las hojas. Las muestras colectadas en campo fueron transportadas al Laboratorio de Fitopatología Molecular de la Universidad Nacional de Colombia –Sede Palmira, en donde fueron sometidas a análisis moleculares.
Extracción de DNA total. Para llevar a cabo los diferentes análisis moleculares se realizó una extracción de DNA genómico de las hojas de maracuyá colectadas empleando el DNeasy Plant Kit (QIAGEN®), siguiendo las instrucciones del fabricante.
Reacción en cadena de la polimerasa (PCR). Para implementar esta estrategia molecular de análisis se siguió la metodología propuesta por Rojas et al. (1993); empleando los primers específicos de begomovirus bipartitas, PAL1v1978/ PARc496 y PBL1v2040/ PCRc1, que amplifican un fragmento aproximado de 1200 pb correspondiente al DNA del componente genómico A, y un fragmento aproximado de 600pb correspondiente al DNA del componente genómico B geminiviral, respectivamente. El fragmento de 1200pb contiene la región intergénica y parte de los marcos de lectura abiertos relacionados con replicación (proteína asociada a replicación, Rep) y encapsidación viral (proteína de la cápside, Cp); y el fragmento de 600pb contiene una sección de la región intergénica y del gen BC1 involucrado en el movimiento célula a célula del virus a través de los plasmodesmos. La reacción de PCR se llevó a cabo en un volumen de 25 µl con los siguientes componentes: 500ng DNA genómico total, 1X buffer, 0.2 mM de una mezcla de dNTPs, 1.0 µM de cada primer, y 1.25U Taq polimerasa (Fermentas). Cada reacción de PCR se colocó en un termociclador C1000 (Biorad®) con las siguientes condiciones de amplificación: 95°C/180s, 35 ciclos de 95°C/30s, de 52°C/30s y de 72°C/60s; y una extensión de 5-30 minutos a 72°C. Los controles utilizados en las reacciones de PCR fueron: control positivo, DNA plasmídico que porta el virus del mosaico amarillo de la papa (PYMV), un begomovirus bipartita que afecta tomate en Colombia (Vaca-Vaca et al., 2012); y control negativo, agua. Los fragmentos de DNA amplificados fueron sometidos a electroforesis en un gel de agarosa al 0,8% y se visualizaron y fotografiaron utilizando un equipo de fotodocumentación (Molecular Imager Gel DocXR+ Systems BIORAD ®).
Clonación y secuenciación. Los productos de DNA amplificados por PCR fueron ligados en el vector pGem T-Easy (Promega, Madison, WI, USA) siguiendo las instrucciones del fabricante, transformadas en células de E. coli por choque térmico (Sambrook y Russell, 2001) y se verificó la presencia del inserto mediante un análisis de restricción con la enzima EcoRI. El DNA de las clonas que portaban inserto fue enviado a secuenciar en MacroGen®-Korea. Para la secuenciación de los fragmentos clonados se utilizaron los iniciadores universales M13 forward y M13 reverse, empleando el método fluorescente del kit de secuenciación enzimática The Big Dye Terminador DNA (Perkin – Elmer Inc., Branchburg, NJ) y un secuenciador ABI Prisma 377 (Applied Biosystem Inc. Foster City, CA).
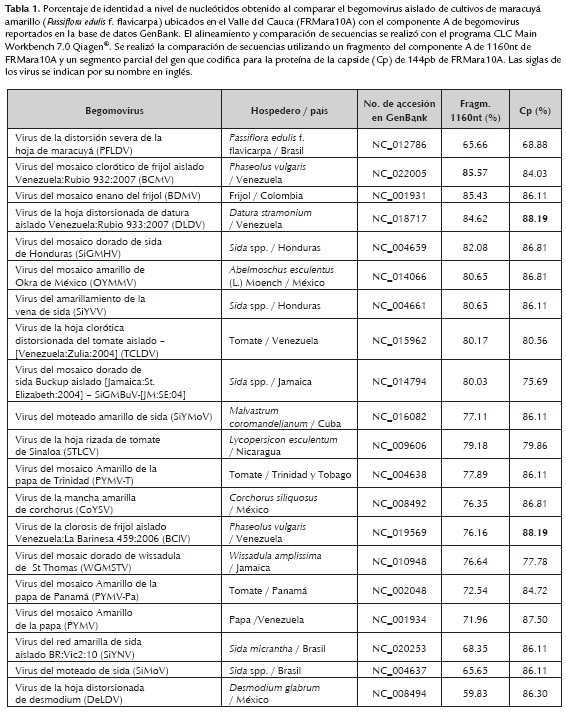
Análisis bioinformático. Las lecturas de secuenciación de los fragmentos virales clonados fueron ensamblados empleando el software CLC Main Workbench 7.0 Qiagen®. Para conocer la identidad de los fragmentos secuenciados se realizó una comparación utilizando el programa Blastn contra la base de datos "refseq_genomic" de Genbank (Altschul et al., 1997). De acuerdo a este análisis, se bajaron del GenBank 19 secuencias parciales de begomovirus que mostraron la mayor identidad con el begomovirus aislado de maracuyá amarillo, con el objetivo de calcular el % de identidad a nivel de nucleótidos empleando el software CLC Main Workbench 7.0 Qiagen® (tabla 1). Con estas secuencias se realizó un alineamiento múltiple, donde se analizó un fragmento de 144nt correspondiente a la región que codifica para la proteína de la cápside (Cp) empleando el programa MUSCLE (Multiple Sequence Comparison by Log-Expectation) (Edgar, 2004). En este análisis se incluyeron adicionalmente las siguientes secuencias de begomovirus: virus de la distorsión severa de la hoja de maracuyá (PSLDV, GenBank: NC_012786), virus del mosaico dorado del tomate (TGMV, GenBank: NC_001507), virus del moteado clorótico del tomate (TCMV, GenBank: NC_003664.1), virus del mosaico dorado del frijo (BGMV, GenBank: NC_004042); y dos begomovirus del viejo mundo: virus de la hoja curva de ají (ChLCV, GenBank: NC_004628) y el virus del rizado amarillo del tomate (TYLCV, GenBank: NC_004005). Para establecer y verificar las relaciones de parentesco, se utilizó el software MEGA 6, aplicando el método de máxima probabilidad, basado en el modelo Tamura-Nei (Tamura et al., 2013), con 1000 réplicas del valor de arranque.
Resultados y discusión
Detección de begomovirus en maracuyá amarilla colectada en La Unión y Palmira, Valle del Cauca
En la figura 1 se observan los síntomas virales observados en las plantas de maracuyá colectadas: deformación de hojas y frutos, presencia de rugosidades, mosaicos amarillos, reducción en el tamaño del fruto, abultamiento y enrollamiento foliar. Mediante PCR se detectó la presencia de los dos componentes genómicos A y B típicos de un begomovirus bipartita en las hojas de maracuyá amarillo colectadas en los municipios de La Unión y Palmira, Valle del Cauca. En la figura 2A, se observa la amplificación en todas las muestras colectadas en Palmira de fragmentos de 1.2 y 0.6kb, correspondientes a los componente A y B begomoviral, respectivamente. Donde, las muestras 1, 2 y 4 evidencian la presencia de ambos componentes, A y B, indicativo de la presencia de un begomovirus bipartita; mientras la muestra 3 solo presenta el componente A y la muestra 5, el componente B. Para las muestras colectadas en La Unión, dos muestras fueron positivas para el componente A y una muestra para el componente B (figura 2B). Este resultado indica que la muestra 2 presenta ambos componentes, A y B; mientras la muestra 1, únicamente se detecta la presencia del componente A. Este resultado se puede explicar teniendo en cuenta que los genomas de los begomovirus bipartitas se encapsidan de manera independiente, lo cual trae consecuencias en cuanto a su transmisión por su vector biológico B. tabaci. Es decir, se puede verificar cualquiera de los dos siguientes casos dependiendo de la distribución azarosa de cualquier de los dos componentes en la planta: uno, ambos componentes genómicos podrían ser transportados por B. tabaci en un mismo evento de alimentación de una planta infectada con este geminivirus a otra; y un segundo caso, es que al alimentarse el vector biológico de la planta infectada tan solo pueda adquirir uno de los dos componentes dependiendo de la disponibilidad de los mismos. Esta observación explicaría las razones por las cuales hay muestras de maracuyá que presentan ambos componentes A y B, o muestras que tan solo uno de los dos componentes es detectado por PCR. Es necesario que ambos componentes estén al mismo tiempo presentes para que el ciclo infectivo del virus se verifique, por lo que las muestras que dieron positivo para ambos componentes es muy probable que el ciclo infectivo del virus se esté verificando en todas sus fases; mientras que en aquellas donde solo se detectó la presencia de un único componente, por ejemplo el A, tan solo se esté verificando parte del ciclo infectivo sin que involucre movimientos a larga distancia ya que para ello se requiere la presencia del componente B. Es decir, el componente A por si solo contiene determinantes de patogenicidad que pueden generar la aparición de sintomatología local no sistémica. La presencia de estos dos componentes virales, A y B, en las muestras de maracuyá colectadas en campo confirma la detección por primera vez de un begomovirus típico del nuevo mundo perteneciente a la familia Geminiviridae afectando cultivos de maracuyá amarillo en Colombia. Este resultado es relevante, ya que los begomovirus son considerados virus emergentes y pueden ocasionar daños severos a los cultivos hasta convertir estos en inviables.
Caracterización parcial de un begomovirus aislado de maracuyá amarillo en La Unión y Palmira, Valle del Cauca
El fragmento de 1.2kb del componente A de la muestra 2 de La Unión y muestra 1 de Palmira, fue clonado y secuenciado. Se obtuvieron dos clonas para el fragmento de La Unión y una clona para el fragmento de Palmira. Los análisis de la secuencia de nucleótidos de los componentes A clonados presentaron un tamaño de 1160 nucleótidos, donde los tres insertos virales clonados mostraron ser idénticos. Este resultado indica que el begomovirus aislado de cultivos de maracuyá amarillo ubicados en Valle del Cauca (La Unión y Palmira) es una misma entidad viral. Un resultado similar fue encontrado para el virus del mosaico amarillo de la papa (PYMV), un begomovirus que predomina afectando el cultivo de tomate en Valle del Cauca, donde todos los fragmentos clonados de muestras colectadas en diferentes lotes fueron idénticos (Vaca-Vaca et al., 2012). El fragmento de 1160nt secuenciado incluye un marco de lectura parcial de 700nt que codifica para una proteína asociada a replicación Rep (AL1); la región intergénica de 316nt que a su vez incluye una estructura de tallo y asa que contiene un elemento de secuencia nonanucleotídico conservado (TAATATTAT) típico de Begomovirus así como un marco de lectura parcial de 144nt que codifica para la proteína de la cápside (Cp o AR1). La secuencia parcial de 1160nt del componente A del begomovirus aislado de la muestra de maracuyá de La Unión fue depositada en la base de datos Genbank con el número de accesión KX827633. Al comparar la secuencia de 1160 nucleótidos del componente A contra PSLDV-A (GenBank: FJ972767.1), un begomovirus que afecta maracuyá en Brasil (Ferreira et al., 2010), se obtuvo un porcentaje de identidad de 73%, lo que estaría indicando que el begomovirus identificado en este trabajo es una entidad viral diferente de la detectada en Brasil.
Con el fin de conocer si éste begomovirus estaba relacionado con algún otro miembro de la familia Geminiviridae se llevó a cabo una comparación de la secuencia de 1160 nucleótidos del componente A con secuencias de begomovirus reportados en la base de datos GenBank (base de datos utilizada: Refseq_genomic database). Los resultados de este análisis permitieron evidenciar que este begomovirus presentaba los porcentajes más altos de identidad a nivel de nucleótidos con begomovirus previamente aislados de frijol, malezas y tomate: 85% con el virus del mosaico clorótico de frijol aislado Venezuela:Rubio 932:2007 (BCMV-A, Genbank, NC_022005.1), que afecta frijol en Venezuela; 84% con el virus de la hoja distorsionada de datura aislado Venezuela:Rubio 933:2007 (DLDV-A, Genbank, NC_018717.1), reportado en la maleza Datura stramonium L. en Táchira, Venezuela; 85% con el virus del mosaico enano del frijol (BDMV-A, Genbank, NC_001931.1), que afecta frijol en Palmira, Valle del Cauca, Colombia; 81% con el virus del mosaico dorado de sida Buckup aislado Jamaica:St. Elizabeth:2004 (SiGMBuV-A [JM:SE:04], Genbank, NC_014794.1), reportado en la maleza Sida spp. en Jamaica; 81% con virus de la hoja clorótica distorsionada del tomate aislado Venezuela:Zulia:2004 (TCLDV-A, Genbank, NC_015962.1); y 81% con el virus de la hoja curva moteada del tomate de Zulia (Genbank, NC_015122.1), que afecta tomate en Venezuela.
Para el componente B geminiviral se obtuvieron dos clonas, una de la muestra colectada de maracuyá amarrillo en La Unión y una de Palmira. Los insertos presentaron un tamaño de 520 nucleótidos y fueron idénticos. El análisis bioinformático de esta secuencia en la base de datos GenBank (base de datos utilizada: Refseq_genomic database) empleando el programa blastn mostró porcentajes de identidad bajos con begomovirus que afectan frijol: 74% con BCMV-B (Genbank, NC_022003.1) que afecta frijol en Venezuela; 69% con BDMV-B (Genbank, NC_001930.1) que afecta frijol en Palmira (Colombia). Mientras que con PSLDV-B (GenBank, NC_012787.1) esté fragmento del componente B mostró una identidad del 83% pero con una cobertura del fragmento de sólo 34%.
En la tabla 1 se muestran los resultados obtenidos al calcular el % de identidad a nivel de nucleótidos del fragmento de 1160nt del begomovirus aislado de maracuyá amarillo con begomovirus reportados en la base de datos GenBank. PSLDV-A aislado de maracuyá en Brasil mostró una identidad de únicamente 65.66% al comparar el fragmento completo de 1160 nt; y cuando se realizó la comparación con el segmento que codifica para la proteína de la cápside la identidad fue de únicamente 68.88% (tabla 1). El mayor % de identidad encontrada al analizar el fragmento de 1160nt fue de 85.57% con BCMV-A, un begomovirus aislado de Phaseolus vulgaris en Venezuela. Cuando se realizó la comparación con el segmento que codifica para la proteína de la cápside, la mayor identidad fue 88.19%con dos begomovirus: DLDV-A aislado de Datura stramonium y BClV-A, aislado de Phaseolus vulgaris, ambos begomovirus reportados en Venezuela. Recientemente, Brown et al. (2015), establecieron como criterio taxonómico para la demarcación de una nueva especie begomoviral un valor por debajo de 91% de identidad a nivel de nucleótidos del genoma completo del componente genómico A. En el presente trabajo de investigación se cuenta de una secuencia parcial de 1160 nt del genoma A (40% del genoma completo que tiene aproximadamente 2600nt), que permite únicamente concluir que la entidad begomoviral aislada de maracuyá amarillo en Valle de Cauca es diferente al aislado viral reportado de maracuyá en Brasil, así como de otros begomovirus reportados previamente a nivel mundial cuyas secuencias están depositadas en la base de datos GenBank.
Vale la pena resaltar la relación de identidad del aislado begomoviral colombiano con otros geminivirus aislados de malezas tales como Datura y Sida spp, ya que las evidencias actuales indican que son las arvenses aquellas que fungen como hospederos alternativos por excelencia para la mayoría de los virus vegetales y los geminivirus no son la excepción a esta regla. Desde este punto de vista, las malezas se convierten en un sitio de paso o en un puente a partir del cual la mosca blanca B. tabaci puede tomar y transportar estas nuevas variantes de geminivirus hacia nuevos cultivares en los cuáles éstos se adaptan evolutivamente y emerger rápidamente como una amenaza fitopatológica para los mismos. Los resultados de Caraballi et al. (2004) confirman este hecho, al demostrar como la mosca blanca podría trasmitir el geminivirus ACMV, el virus africano del mosaico de la yuca, a la yuca en Colombia sí previamente era capaz de adaptarse a hospederos alternativos vegetales emparentados con este cultivo. Este pudo ser el camino que siguió el aislado begomoviral colombiano: es decir, inicialmente estuvo en una maleza y desde allí fue transportado por su vector biológico a los cultivos de maracuyá, posiblemente en varias ocasiones durante el devenir de su transcurso evolutivo, de tal manera que con el paso del tiempo llegó a convertirse en una entidad viral propia genéticamente muy diferente de otros geminivirus que afectan este mismo cultivo en otras latitudes del planeta. En cuanto a los valores de identidad que el aislado colombiano presentó con geminivirus reportados afectado el cultivo de frijol, bien puede hipotetizarse que un fenómeno similar al planteado en malezas pudo verificarse: es decir, el aislado colombiano que afecta al maracuyá pudo haber evolucionado de un geminivirus que afectaba el cultivo del frijol o ambos evolucionaron de un antecesor común, sugiriendo que durante el devenir evolutivo el aislado colombiano que afecta el maracuyá bien sea se adaptó a infectar maracuyá o perdió su capacidad para infectar frijol.
Los experimentos de pesudorecombinación obtenidos por Ferreira et al., (2010) demuestran que para el caso de PSLDV, este evolucionó muy probablemente a partir de uno o varios antecesores begomovirales que afectaban el tomate en épocas pretéritas. Si este es el caso del aislado geminiviral que está afectando el cultivo de maracuyá en Colombia, es materia aún por comprobar. Sin embargo es bien sabido que la región del Valle del Cauca ha sido un departamento líder en la producción de frijol en Colombia; miles de hectáreas de Valle del Cauca estuvieron cultivadas con frijol y hoy este cultivo se halla en declive debido en gran medida a la susceptibilidad de este cultivo a los virus, particularmente geminivirus (Cuellar & Morales, 2006; Cuellar et al., 2011). Es probable que ante la sensible disminución de las áreas sembradas de frijol, los geminivirus que lo afectaban se vieran presionados a buscar un hospedero alterno como el maracuyá. Allí los geminivirus se coadaptaron a tal punto que los aislados colombianos divergieron tanto que no se encuentran filogenéticamente relacionados con otros begomovirus que afectan este cultivo en otras latitudes del planeta. Este tipo de eventos ya han sido comprobados entre en geminivirus tales como el virus del mosaico del abutilón (AbMV), el virus del moteado del tomate (ToMoV) y el virus del mosaico enano del frijol (BDMV). Estos tres geminivirus están filogenéticamente relacionados, hecho que ha llevado a pensar que este complejo geminiviral evolucionó de un antecesor viral semejante al AbMV, a partir del cual divergieron el ToMoV y el BDMV los cuales con el tiempo se adaptaron al tomate y al frijol respectivamente (Gilbertson et al., 1993).
Para conocer las relaciones filogenéticas de aislado begomoviral obtenido en este estudio con otros reportados a nivel mundial, se llevó a cabo un análisis de secuencia de nucleótidos empleando un fragmento que porta la región 5' del gen que codifica para la proteína de la cápside (Cp) que tiene un tamaño de 144nt. En la figura 3 se aprecia el árbol filogenético obtenido a partir de la comparación de Cp entre geminivirus reportados a la fecha y el aislado colombiano que afecta el maracuyá (FRMara10A) corroborando que el aislado colombiano y PSLDV-A forman dos grupos diferentes a pesar de afectar un mismo hospedero y ser ambos geminivirus bipartitas típicos del hemisferio occidental. El aislado colombiano FRMara10A se agrupa con los geminivirus DLDV-A aislado de Datura stramonium en Venezuela y BClV-A aislado de Phaseolus vulgaris en Venezuela. Este resultado permite hipotetizar que el geminivirus que está afectando el cultivo de maracuyá en Colombia está emparentado con begomovirus reportados en Venezuela que afectan cultivos de frijol así como con la maleza Datura stramonium y podría estar indicando que esta maleza podría ser huésped natural de este virus en campo. Mientras que PSLDV se relaciona más con TGMV, un begomovirus que afecta grandemente el cultivo de tomate en Brasil.
La ausencia de reportes previos de geminivirus afectando maracuyá en Colombia puede estar correlacionada con el hecho de que los biotipos predominantes de mosca blanca estaban pobremente adaptados a alimentarse del maracuyá. Pudieron entonces surgir algunos individuos dentro de la población de mosca blanca que sí lograron colonizar maracuyá, cumpliéndose el principio de la virología clásica que afirma que para que una infección geminiviral se desarrolle debe primero ocurrir un fenómeno de coadaptación entre el virus, el vector biológico y el hospedero. Evidencias de este principio las obtuvo el grupo de Harkins et al. (2009), quienes demostraron que el virus del estriado del maíz (MSV) apareció por sucesivos eventos de recombinación que sucedieron en malezas relacionadas filogenéticamente con el maíz en África. Y que una vez el maíz fue introducido a este continente hacia los años 1800, variantes de estos MSV lograron colonizar desde las malezas al maíz emergiendo allí como una grave patología que afecta fuertemente la productividad de cultivo hoy en día.
Conclusiones
Se detectó por primera vez la presencia de un begomovirus en cultivos de maracuyá amarillo ubicados en los municipios de la Unión y Palmira, Valle del Cauca, Colombia.
Los análisis de secuencia de nucleótidos y el análisis filogenético mostraron que el begomovirus aislado de maracuyá en Colombia es un geminivirus diferente a PSLDV, un begomovirus previamente reportado afectando éste cultivo en Brasil.
Los análisis de secuencias mostraron que el begomovirus aislado de maracuyá presenta un porcentaje de identidad menor de 91% con otros begomovirus reportados previamente a nivel mundial en el GenBank, indicando que podría ser una entidad viral nueva.
Finalmente, es importante continuar estudiando los begomovirus que afectan el cultivo de maracuyá en otras regiones productoras en Colombia, obtener el genoma completo de este virus para realizar su identificación plena, conocer su organización genómica y su diversidad genética, así como utilizarlo para establecer un programa de mejoramiento genético orientado a la identificación de materiales con resistencia a este geminivirus, acciones que permitan desarrollar estrategias efectivas de manejo integrado para disminuir la incidencia de esta enfermedad viral en cultivos de maracuyá.
Agradecimientos
Los autores expresan sus agradecimientos al Ingeniero Santiago Pérez quien colaboró con sus cultivos para la toma de las muestras de maracuyá y a la Dirección de Investigación de la Universidad Nacional de Colombia sede Palmira (DIPAL) quien financió el proyecto (CÓDIGO HERMES 13711 y 26138). E. Carrasco-Lozano agradece al Programa Nacional de Becas (PRONABEC) del gobierno Peruano, por la beca otorgada para sus estudios de Maestría.
Notas
1 Santiago Pérez, Ingeniero Agrónomo Frutas Tropicales Valle del Cauca-Colombia.
Referencias bibliográficas
AGRONET. (2015). Sistema de Estadísticas Agropecuarias-SEA). Agronet: Bogota, Colombia Recuperado de: http://www.agronet.gov.co/agronetweb1/Estadisticas.aspx.
Altschul, S. F., Madden T. L., Schaffer, A. A., Zhang, J., Zhang, Z., Miller, W., & Lipman, D. J. (1997). Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Research, 25(17), 3389-3402.
Caraballi, A., Bellotti, A. C., Montoya, J., & Cuellar, M. E. (2004). Adaptation of Bemisia tabaci biotype B (Gennadious) to cassava Manihot esculenta (Crantz). Crop Protection,24 (7), 643-649.
Cuéllar, M. E., & Morales, F. J. (2006). La mosca blanca Bemisia tabaci (Gennadius) como plaga y vectora de virus en fríjol común (Phaseolus vulgaris L.). Revista Colombiana de Entomología, 32(1), 1-9.
Cuellar, M. E., Morales, F. J., & Lerma, J. M. (2011). Resistencia genética al Virus del Arrugamiento Foliar del Fríjol transmitido por Bemisia tabaci (Hemiptera: Aleyrodidae. Revista Colombiana de Entomología, 37(1), 8-15.
Edgar, R.C. (2004). MUSCLE: multiple sequence alignment with high accuracy and high throughput". Nucleic Acids Research, 32(5), 1792-1797.
Ferreira, S. S., Barros, D. R., Almeida, M. R., & Zerbini, F. M. (2010). Characterization of Passionfruit severe leaf distortion virus, a novel begomovirus infecting passion fruit in Brazil, reveals a close relationship with tomato-infecting begomoviruses. Plant Pathology, 59(2), 221-230.
Gilbertson, R. J., Hidayat, S. H., Paplomatas, E. J., Rojas, M. R., Hou, Y. M., & Maxwell, D. P. (1993). Pseudorecombination between infectious cloned DNA components of tomato mottle and bean dwarf mosaic geminiviruses. Journal of General Virology, 74(1), 23-31.
Harkins, G., Martin, D., Duffy, A., & Monjane, A. (2009). Dating the origins of the maize-adapted strain of maize streak virus (MSV-A). Journal of General Virology, 90(12), 3066-3074.
Harrison, B. D, Swanson, M. M., & Fargette, D. (2002). Begomovirus coat protein: serology, variation and functions. Physiological and molecular plant pathology,60(5), 257-271.
Holm-Nielsen, L. B., Jorgensen P. M., & Lawesson. J. E. (1988). Passifloraceae. pp. 1-130. En: Harling, G. y L. Anderson (eds.). Flora del Ecuador 31. University of Göteborg, Copenage.
ICTV (2015). International Committee on Taxonomy of Viruses. Recuperado de: http://www.ictvonline.org/virusTaxonomy.asp.
Morales, F., Lozano, I., Muñoz, C., Castaño, M., Arroyave, J., Varon, F., Chavéz, B., & Castillo, G. (2001). Caracterización molecular de los virus que afectan el maracuyá (Passiflora edulis Sims) y otras pasifloras en Colombia. Fitopatología Colombiana,25, 100-102.
Novaes, Q. S., Freitas-Astua, J., Yuki, V. A., Kitajima, E. W., Camargo, L. E. A., & Rezende, J. A. M. (2003). Partial characterization of a bipartite begomovirus infecting yellow passion flower in Brazil. Plant Pathology,52(5), 648-654.
Ocampo, P. J., Coppens d'Eeckenbrugge, G., Restrepo, M., Jarvis, A., Salazar, M., & Caetano, C. (2007). Diversity of Colombian Passifloraceae: biogeography and an updated list for conservation. Biota Colombiana,8(1), 1-45.
Passionfruit. (2015). Supply and demand. Recuperado de: http://www.passionfruitjuice.com/supply.php?MENU=5.
Rodríguez, I. V., Bueno, J. M., Cardona, C., & Morales, H. (2012). Biotipo B de Bemisia tabaci (Hemiptera: Aleyrodidae): plaga de pimentón en el Valle del Cauca, Colombia. Revista Colombiana de Entomología, 38(1), 14-22.
Rojas, M. R., Charles, H., William, L. J., & Gilbertson, R. L. (2005). Exploiting chinks in the plant's armor: evolution and emergence of geminiviruses. Annual Review of Phytopathology, 43, 361-394.
Rojas, M. R., Gilbertson, R. L., Russell, D. R., & Maxwell, D. P. (1993). Use of degenerate primers in the polymerase chain reaction to detect whitefly-transmitted geminiviruses. Plant Disease, 77, 340-347.
Sambrock, J., & Russell, W. D. (2001). Molecular cloning: a laboratory manual. 3rd ed. Cold Spring Harbor Laboratory Press. New York. Vol. I-III.
Seal, S. E., VandenBosch, F., & Jeger, M. J. (2006). Factors influencing begomovirus evolution and their increasing global significance: implications for sustainable control. Critical Reviews in Plant Sciences,25 (1), 23-46.
Tamura, K., Peterson, D., Peterson, N., Stecher, G., Nei, M., & Kumar, S. (2011). MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Molecular Biology and Evolution, 28(10), 2731-2739.
Ulmer, T., & MacDougal, J. M. (2004). Passiflora: Passion-flowers of the world. Timber Press, Oregon, OR. Inc. 430 pp.
Vaca-Vaca, J. V., Betancur-Pérez, J. F., & López-López, K. (2012). Distribución y diversidad genética de Begomovirus que infectan tomate (Solanum lycopersicum L.) en Colombia. Revista Colombiana de Biotecnología,14(1), 60-76.
Referencias
AGRONET, 2015. Consultado el 3 de Agosto de 2015. Sistema de Estadísticas Agropecuarias–SEA): http://www.agronet.gov.co/agronetweb1/ Estadisticas.aspx.
Altschul, S. F., Madden, T.L., Schaffer, A. A., Zhang, J., Zhang, Z., Miller, W. and Lipman, D. J. 1997. Gapped BLAST and PSI‐BLAST: a new generation of protein database search programs. Nucleic Acids Research 25: 3389–3402.
Caraballi, A., Bellotti, A. C., Montoya, J. and Cuellar, M. E. 2004. Adaptation of Bemisia tabaci biotype B (Gennadious) to cassava Manihot esculenta (Crantz). Crop Protection 24: 643-649.
Cuéllar, M. E. y Morales, F. J. 2006. La mosca blanca Bemisia tabaci (Gennadius) como plaga y vectora de virus en fríjol común (Phaseolus vulgaris L.). Rev. Colomb. Entomol. 32: 1-9.
Cuellar, M. E., Morales, F. J. y Lerma, J. M. 2011. Resistencia genética al Virus del Arrugamiento Foliar del Fríjol transmitido por Bemisia tabaci (Hemiptera: Aleyrodidae). Rev. Colomb. Entomol. 37: 8-15.
Edgar, R.C. 2004. MUSCLE: multiple sequence alignment with high accuracy and high throughput". Nucleic Acids Research. 32: 1792–97.
Ferreira, S. S., Barros, D. R., Almeida, M. R. and Zerbini, F. M. 2010. Characterization of passiónfruit severe leaf distortion virus, a novel begomovirus infecting pasión fruit in Brazil, reveals a close relationship with tomato-infecting begomoviruses. Plant Pathology 59: 221-230.
Gilbertson, R. J., Hidayat, S. H., Paplomatas, E. J., Rojas, M. R., Hou, Y. M. and Maxwell, D.P. 1993. Pseudorecombination between infectious cloned DNA components of tomato mottle and bean dwarf mosaic geminiviruses. Journal of General Virology 74: 23-31.
Harkins, G., Martin, D., Duffy, A. and Monjane, A. 2009. Dating the origins of the maize-adapted strain of maize streak virus (MSV-A). Journal of General Virology 90: 3066-3074.
Harrison, B. D, Swanson, M. M., Fargette, D. 2002. Begomovirus coat protein: serology, variation and fuctions. Physiological and molecular plant pathology 60: 257-271.
Holm-Nielsen, L. B., Jorgensen P. M., Lawesson. J. E. 1988. Passifloraceae. pp. 1-130. En: Harling, G. y L. Anderson (eds.). Flora del Ecuador 31. University of Göteborg, Copenage.
ICTV 2015. Consultado el 3 de Agosto de 2015 en: International Committee on Taxonomy of Viruses: http://www.ictvonline.org/virusTaxonomy.asp.
Morales, F., Lozano, I., Muñoz, C., Castaño, M., Arroyave, J., Varon, F., Chavéz, B. y Castillo, G. 2001. Caracterización molecular de los virus que afectan el maracuyá (Passiflora edulis Sims) y otras pasifloras en Colombia. Fitopatología colombiana 25: 100-102.
Novaes, Q. S., Freitas-Astua, J., Yuki, V. A., Kitajima, E. W., Camargo, L. E. A., Rezende, J. A. M. 2003. Partial characterization of a bipartite begomovirus infecting yellow passion flower in Brazil. Plant Pathology 52: 648-654.
Ocampo, P. J., Coppens d’Eeckenbrugge, G., Restrepo, M., Jarvis, A., Salazar, M. y Caetano, C. 2007. Diversity of Colombian Passifloraceae: biogeography and an updated list for conservation. Biota Colomb. 8(1), 1-45.
Passionfruit 2015. Consultado el 3 de Agosto 2015 en: Supply and demand. http://www.passionfruitjuice.com/supply.php?MENU=5.
Rodríguez, I. V., Bueno, J. M., Cardona, C. y Morales, H. 2012. Biotipo B de Bemisia tabaci (Hemiptera: Aleyrodidae): plaga de pimentón en el Valle del Cauca, Colombia. Rev. Colomb. Entomol. 38: 14-22. 0120-0488.
Rojas, M. R., Charles, H., William, L. J. and Gilbertson, R. L. 2005. Exploiting chinks in the plant’s armor: evolution and emergence of geminiviruses. Annual Review of Phytopathology 43:361.
Rojas, M. R., Gilbertson, R. L., Russell, D. R. and Maxwell, D. P. 1993. Use of degenerate primers in the polymerase chain reaction to detect whitefly-transmitted geminiviruses. Plant Disease 77: 340-347.
Sambrock, J. and Russell, W. D. 2001. Molecular cloning: a laboratory manual. 3rd ed. Cold Spring Harbor Laboratory Press. New York. Vol. I-III.
Seal, S. E., VandenBosch, F. and Jeger, M. J. 2006. Factors influencing begomovirus evolution and their increasing global significance: implications for sustainable control. Critical Reviews in Plant Sciences 25:23.
Tamura, K., Peterson, D., Peterson, N., Stecher G., Nei, M. and S. Kumar. 2011. MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Molecular Biology and Evolution 28: 2731-2739.
Ulmer, T. y MacDougal, J. M. 2004. Passiflora: Passion-flowers of the world. Timber Press, Oregon, OR. Inc. 430 pp.
Vaca-Vaca, J. V., Betancur-Pérez, J. F., & López-López, K. (2012). Distribución y diversidad genética de Begomovirus que infectan tomate (Solanum lycopersicum L.) en Colombia. Rev. Colomb. Biotecnol, 14, 60-76.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
CrossRef Cited-by
1. Francy Jhoana Ceballos-Burgos, Jorge Iván Pérez-López, Andres Felipe Nieto-Cardenas, Diana Marcela Rivera-Toro, Jhon Albeiro Ocampo, Karina Lopez-Lopez, Juan Carlos Vaca-Vaca. (2024). Virus prevalence and mixed infections in yellow passion fruit (Passiflora edulis f. flavicarpa) crops in Valle del Cauca, Colombia. Revista U.D.C.A Actualidad & Divulgación Científica, 27(1) https://doi.org/10.31910/rudca.v27.n1.2024.2364.
Dimensions
PlumX
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2016 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).