Caracterización molecular de un banco de germoplasma del género Theobroma mediante la técnica RAPD*
Molecular characterisation of a germplasm bank for Theobroma genus using the RAPD technique
Palabras clave:
Theobroma grandiflorum, Theobroma bicolor, RAPD, caracterización molecular, Theobroma bicolour, molecular characterisation (es)|
|
||
|
____________________REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL VI No. 2 Diciembre 2004 15-24
Caracterización molecular de un banco de germoplasma del género Theobroma mediante la técnica RAPD*
Molecular characterisation of a germplasm bank for Theobroma genus using the RAPD technique
|
||
|
|
||
|
** ***
|
||
|
|
||
|
Yovany Moreno** , Luz Marina Melgarejo*** , María Soledad Hernández**** , Lorena Quintero*****, Guillermo Vargas***
RESUMEN
Mediante la utilización de marcadores RAPD (Random Amplified Polymorphic DNA), se analizaron 145 materiales (128 de T. grandiflorum y 17 de T. bicolor) pertenecientes al banco de germoplasma ex situ del género Theobroma del Instituto Sinchi, localizado en San José del Guaviare. A partir de un número inicial de 20, se seleccionaron los 5 cebadores capaces de generar mayor número de polimorfismos para generar 114 bandas que lograron distinguir entre más del 99% de los materiales analizados: 57 bandas para T. grandiflorum (84,2% polimórficas), 45 bandas para T. bicolor (26,7% polimórficas) y 12 bandas compartidas entre las dos especies (58,3% polimórficas). A partir de la matriz de semejanza generada utilizando el índice de Dice, representada en un dendrograma UPGMA, y el análisis de componentes principales, se determinó un alto grado de semejanza intraespecífica en los materiales analizados, particularmente en T. bicolor. Luego de comparar este análisis con el morfoagronómico previamente realizado en algunos materiales de T. grandiflorum, se encontró que los grupos generados por dicha evaluación morfológica y agronómica son heterogéneos a nivel molecular. La información obtenida se utilizará como herramienta para la toma de decisiones en cuanto a las estrategias de mantenimiento, enriquecimiento y uso del banco.
Palabras clave: Theobroma grandiflorum, Theobroma bicolor, RAPD, caracterización molecular.
ABSTRACT
RAPD markers (Random Amplified Polymorphic DNA) were used for analysing 145 individuals (128 T. grandiflorum and 17 T. bicolor) from the ex situ Theobroma genus germplasm bank at Instituto Sinchi, located at San José del Guaviare. 5 primers able to generated polymorphism were selected from an initial set of 20, generating 114 bands that enable to us to distinguish between more than 99% of individuals analysed: 57 bands for T. grandiflorum (84.2% polymorphic), 45 bands for T. bicolor (26.7% polymorphic) and 12 bands
|
||
|
|
||
|
* Este artículo corresponde a la presentación en pósterque obtuvo el tercer lugar en la sala de biotecnología agrícola
del segundo Congreso Colombiano de Biotecnología. ** Químico, estudiante del programa de maestría en ciencias-bioquímica de la Universidad Nacional de Colombia.
Instituto Amazónico de Investigaciones Científicas (Sinchi), Calle 20 No. 5-44, AA.03471, Bogotá, D. C. Colombia.
Correo electrónico: p_infestans@yahoo.com
*** Bióloga, Dr. Sc. química. Departamento de Biología, Facultad de Ciencias, Universidad Nacional de Colombia-Sede Bogotá. Correo electrónico: lmmelgarejom@unal.edu.co **** Bióloga, Dr. Sc. agropecuarias con énfasis en fisiología vegetal. Instituto Amazónico de Investigaciones Científicas
(Sinchi), Calle 20 No. 5-44. AA.03471, Bogotá, D.C. Colombia. Correo electrónico: shernandez@sinchi.org.co ***** Bióloga, estudiante del programa de maestría en ciencias agropecuarias con énfasis en fitomejoramiento de la
Universidad Nacional de Colombia. Instituto Amazónico de Investigaciones Científicas (Sinchi), Calle 20 No. 5-44.
AA.03471 Bogotá, D. C. Colombia. Correo electrónico: lquintero@sinchi.org.co ****** Ingeniero agrónomo, M. Sc. fitotecnia con énfasis en fisiología de cultivo. Instituto Amazónico de Investigaciones
Científicas (Sinchi), Estación Experimental "El Trueno", San José del Guaviare, Departamento del Guaviare,
Colombia. Correo electrónico: gvargas@sinchi.org.co
Recibido: octubre 26 de 2004. Aceptado: octubre 29 de 2004.
|
||
|
|
||
|
15
|
||
|
|
||
|
|
|||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 15-24
|
|||
|
|
|||
|
shared between the two species (58.3% polymorphic). A high degree of intra-specific similarity particularly in T. bicolor was established from the similarity matrix obtained by using the Dice index and represented in a UPGMA dendrogram and the principal components analysis (PCA). The comparison of this analysis with a previous morpho-agronomic evaluation of some T. grandiflorum individuals revealed that the groups generated on the basis of its agronomic and morphological traits were heterogeneous at molecular level. The obtained information will be used as a tool in strategies regarding maintenance, enrichment and use of the germplasm bank.
Key words: Theobroma grandiflorum, Theobroma bicolour, RAPD, molecular characterisation.
|
|||
|
|
|||
|
INTRODUCCIÓN
Al igual que el cacao (Theobroma cacao), el copoazú (T grandiflorum) y el maraco (T bicolor) son árboles tropicales originarios de la región amazónica cuya importancia dentro de los sistemas de producción agrícola radica en el potencial de sus frutos para obtener pulpas de aroma y sabor agradable que pueden ser utilizados en jugos, jaleas, helados, etc., o en la preparación de licores similares a los del cacao de buena calidad (cupulate y bacalate) a partir del proceso de fermentación de sus semillas, particularmente en la región amazónica en donde puede ser cultivado sin alterar el bosque original (Casas, 1995; Urano de Car-valho et al., 1999; Leal et al., 2000).
El banco de germoplasma ex situ del género Theobroma, custodiado por el Instituto Amazónico de Investigaciones Científicas (Sinchi), se encuentra localizado en San José del Guaviare (departamento del Guaviare, Colombia) y surge inicialmente como parte de una estrategia de soporte al desarrollo de sistemas productivos alternativos a los cultivos ilícitos, por medio de la cual se intenta generar opciones de producción sostenible ambiental y económicamente, con recursos genéticos caracterizados por material nativo, promisorio y accesibles al cultivador (Vargas y Argüelles, 2000).
Actualmente el banco contiene materiales de las especies T. bicolor (5 años de establecido) y T. grandiflorum (19 años de establecido). Debido a la antigüedad del banco de T grandiflorum, no existen los datos de pasaporte de los materiales y se presume su procedencia de tres regiones geográficas diferentes: Iquitos (Ecuador), Belém do Pará (Brasil) y San José del Guaviare (Colombia) (G. Vargas, comunicación personal). Los materiales fueron clasificados en ecotipos dependiendo de sus características morfológicas y/o agronómicas: 12 grupos para T. grandiflorum (3001 al 3012) y 4 para T bicolor
|
(MB1 a MB4), algunos de ellos poseen características agronómicas de interés para llevar a cabo su explotación comercial (Vargas y Argüelles, 2000; Vargas et al., 2002).
Debido a la relevancia y urgencia de utilizar estas especies en la región amazónica, se requiere un nivel más profundo de conocimiento sobre los materiales conservados para escoger aquellos que puedan ser de mayor interés en el enriquecimiento de los sistemas productivos, capaces de soportar la demanda para producción de licor de cacao de buena calidad.
El uso de marcadores moleculares para la caracterización de bancos de germoplasma es una herramienta útil y ventajosa en las labores de evaluación de la variabilidad genética, debido a que los descriptores morfológicos son sensibles a factores ambientales (Charters y Wilkinson, 2000). Se han descrito diversos tipos de marcadores moleculares: RFLP, SSR, AFLP, RAPD, cada uno con características propias, ventajas y desventajas (ver p. ej. Mueller y Wol-fenbarger, 1999). Su utilización es una alternativa viable si los términos de costo, tiempo, simplicidad y reproducibilidad son aceptables. Pueden generar un complemento a la información existente sobre los materiales con el fin de disminuir costos en el manejo del banco y determinar la posible necesidad de realizar nuevas colectas para su enriquecimiento.
La técnica RAPD (Random Amplified Polymorphic DNA) (Williams et al., 1990; Welsh & McCle-lland, 1990) se basa en la Reacción en Cadena de la Polimerasa (PCR). Utiliza cebadores cortos (usual-mente 10 pb) y condiciones de reacción poco astringentes para amplificar fragmentos discretos de ADN (Hansen et al., 1998). Ha sido utilizada en varios trabajos concernientes a la labor de curación de bancos de germoplasma, entre las que se encuentran los de cacao (Wilde, et al., 1992; Lerceteau, et al., 1997a; Lerceteau, et al., 1997b), posee varias de
|
||
|
|
|||
|
16
|
|||
|
|
|||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
CARACTERIZACIÓN MOLECULAR DE UN BANCO DE GERMOPLASMA DEL GÉNERO Theobroma
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
las características mencionadas anteriormente (costo, simplicidad, rapidez) y permite obtener información complementaria requerida, razón por la cual fue seleccionada para realizar la caracterización molecular, objeto de este trabajo.
MATERIALES Y MÉTODOS
Material Vegetal. Se utilizaron hojas jóvenes de 150 materiales: 145 pertenecientes al banco de germo-
|
plasma ex situ del género Theobroma (127 de T. grandiflorum y 17 de T. bicolor), de la estación experimental "El Trueno" del Instituto Amazónico de Investigaciones Científicas (Sinchi) en San José del Guaviare (departamento del Guaviare, Colombia), 4 muestras colectadas de árboles ubicados en Leticia y una en Guainía, que fueron utilizados como materiales externos al banco por ser de origen diferente. En la tabla 1 se presenta el sumario de los materiales utilizados para esta caracterización.
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Tabla 1. Listado de los materiales utilizados para la caracterización molecular del banco de germoplasma del género
Theobroma custodiado por el Instituto Sinchi. En el caso de los materiales de T. grandiflorum se muestra el número
del material seguido del grupo morfoagronómico (# - 30XX) dependiendo de si el material ha sido clasificado
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
17
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
|||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 15-24
|
|||
|
|
|||
|
Las hojas fueron colectadas sobre sílica gel y transportadas bajo condiciones refrigeradas hasta el laboratorio en donde fueron lavadas con agua, enjuagadas con etanol al 90%, secadas con una toalla de papel y maceradas en nitrógeno líquido hasta la obtención de un polvo fino, el cual fue almacenado a -70°C para posteriores análisis.
Extracción y cuantificación de ADN. Para la extracción de ADN se realizó una modificación del protocolo de Doyle y Doyle (1990). Se adicionaron a 0,2 g de hojas maceradas, 0,8 ml de bufferde extracción (CTAB 3%, NaCl 1,4M, 2-mercaptoetanol 0,2%, PVP-40 1%, EDTA 20 mM, Tris-HCl 100 mM, pH 8,0) y se dejó incubar a 65 °C durante 60 min. Posteriormente se realizó una extracción con 0,8 mL de cloroformo: alcohol isoamílico (24:1). El ADN se precipitó adicionando 0,6 mL de isopropanol sobre la fase acuosa recuperada por centrifugación a 1800 xg durante 10 min. El precipitado se centrifugó a 7000 xg durante 10 min. Se adicionó 1 mL de solución de acetato de amonio 10 mM en etanol al 76%, y se centrifugó nuevamente a 7000 xg durante 10 min. El pellet obtenido se secó al aire y se resuspen-dió en 50 |jL de buffer TE. Se adicionó 1 |jL de RNa-sa A (10 mg/mL) y se dejó incubar a 37 °C durante 30 min. El ADN fue cuantificado por fluorometría utilizando Hoescht dye en un fluorómetro DynaQuant 2000 (Hoefer), la calidad fue verificada por electrofo-resis en gel de agarosa al 0,8%, tinción con Bromuro de Etidio y visualización en luz UV.
Amplificación de ADN y electroforesis. Después de la evaluación de las concentraciones de ión Mg2+, dNTP y ADN, las amplificaciones fueron llevadas a cabo en Tris-HCl10 mM pH 8,5, KCl 50 mM, 200 |Jv1 de cada dNTP (Invitrogen), 0,8 \iM de cebador de 10 bases (Set R, Operon Technologies Inc), 1,25 U de Taq Polimerasa (Corpogen) y 50 ng de ADN genómi-co en un volumen final de 25 |jL. Las reacciones se realizaron en un termociclador modelo PTC-100 (MJ Research) programado para un paso inicial de denaturalización a 94 °C durante 3 min. seguido de 46 ciclos de 1 min. a 94 °C, 1 min. a 36 °C y 2 min. a 72 °C. Un paso final de 10 min. a 72 °C y enfriamiento a 4 °C. En cada grupo de reacciones se incluyeron controles positivos y negativos (ausencia de plantilla de ADN).
Los productos de reacción fueron separados por electroforesis en gel de agarosa al 1,8% en
|
buffer TBE 0,5X, durante 150 min. a 5 V/cm, para su visualización con luz UV, previa tinción con bromuro de etidio. Los geles fueron digitalizados utilizando un sistema de análisis Geldoc (Biorad) a partir de los cuales se realizó la lectura de bandas
Análisis de datos. Se construyó una matriz de presencias (1) y ausencias (0)a partir de los perfiles de bandas obtenidas para cada cebador con el fin de realizar la comparación entre las muestras. Se tuvieron en cuenta aquellas bandas que fueran fácilmente identificables y reproducibles. A partir de dicha matriz binaria se generó una matriz de semejanzas utilizando el algoritmo de Dice (1945): 2a/(2a+b+c), siendo a el número de bandas presentes en dos individuos (i,j); b el número de bandas presentes en i y ausentes en j; y c el número de bandas presentes enjy ausentes en i. La matriz de semejanzas fue representada en un dendrogra-ma generado por UPGMA (Unweighted pair-group method). A partir de la matriz de presencias y ausencias se realizó un análisis de componentes principales. Los análisis fueron realizados utilizando el programa NTSYSpc v.2.02j.
RESULTADOS Y DISCUSIÓN
Extracción de ADN. Se evaluaron inicialmente protocolos ampliamente reportados en la extracción de ADN de múltiples especies vegetales como el de Doyle y Doyle (1990), utilizado previamente para T. grandiflorum por Alves et al. (2003) y el de Dellaporta et al. (1980) reportado por Wilde et al. (1992) en T. cacao. Para T. grandiflorum se obtuvo un rendimiento de 4,25 |ug de ADN x g "1 y 3,00 ^g de ADN x g "1 respectivamente, los cuales no son satisfactorios para los propósitos requeridos.
Posteriormente, se probaron otras modificaciones reportadas sobre estos protocolos clásicos (Keb- Llanes et al., 2002; Stein y Raoult, 1992; Szmidt, 2004) y protocolos desarrollados particularmente para T. cacao (Perry et al., 1998; Couch y Fritz, 1990). En el caso del protocolo de Couch y Fritz (1990), se utilizó únicamente el procedimiento para obtención de pellets nucleares en un buffer a pH 6,0 y posteriormente se extrajo el ADN de los núcleos mediante el protocolo de Doyle y Doyle (1990) con el fin generar un procedimiento accesible a las condiciones de laboratorio pues involucraba pasos de purificación de ADN en gradientes de cloruro de cesio. Los resultados obtenidos con estos métodos
|
||
|
|
|||
|
18
|
|||
|
|
|||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
CARACTERIZACIÓN MOLECULAR DE UN BANCO DE GERMOPLASMA DEL GÉNERO Theobroma
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
no fueron tampoco aceptables, en la mayoría de los casos por su rendimiento con valores similares o menores a los obtenidos inicialmente o por la calidad del ADN obtenido (degradación) por la modificación del protocolo de Couch y Fritz (1990).
Las diferencias relacionadas con los protocolos reportados previamente para estas especies o su especie relacionada sugieren que las condiciones ambientales, incluyendo el manejo cultural de los materiales, puede ser un factor clave en el establecimiento de protocolos de extracción de ADN. Los resultados obtenidos sugerían que el control de la oxidación fenólica no era el paso relevante a superar, como se ha planteado para el caso particular de T. cacao (Fritz y Couch, 1990) y que ha sido observado posteriormente en nuestro laboratorio con el protocolo final obtenido; para ello se utilizó p-mercaptoe-tanol al 0,2% y PVP al 1% en los tampones de extracción de manera que se evitó eficientemente dicha oxidación en los procedimientos de extracción de ADN de T. grandiflorum y T. bicolor. Posteriormente, el paso crucial correspondía a la separación de proteínas y carbohidratos del ADN.
De acuerdo con lo anterior, se elevó la concentración del detergente CTAB del 2 al 3% en el buffer de extracción de Doyle y Doyle (1990) tal como lo sugieren dichos autores. Esta modificación permitió extraer ADN de T. grandiflorum y T. bicolor de manera eficiente, aumentándose la cantidad de ADN extraído por el método convencional de Doyle y Doyle (1990) de 4,25 \ig de ADN xg"1a 30,3 \ig de ADN x g-1 con esta modificación. Aumentar la cantidad de CTAB en valores mayores al 3% no mostró mejores resultados debido a que la manipulación de las
|
soluciones de extracción se torna más difícil por aumento en su viscosidad, lo que genera pérdidas.
El ADN obtenido fue de alta calidad, digerible y amplificable, apto para sus posteriores análisis. La utilización de volúmenes menores a los utilizados en el protocolo original ahorra en materiales y reactivos, y permite el procesamiento de hasta 48 muestras al día.
Selección de cebadores y adecuación de las condiciones de amplificación. Se probaron inicialmente 20 cebadores denominados OPR-01 a OPR-20 (set R, Operon Technologies Inc.) con el fin de evaluar su capacidad para generar patrones complejos de bandas en ADN extraído de los materiales 163-3007 de T. grandiflorum y MB3-6 de T. bicolor. Con excepción de los cebadores OPR-17 y OPR-18, todos los cebadores evaluados generaron patrones de bandeo entre 200 y 2000 pb con estos materiales.
Se seleccionó el primer OPR-12 para llevar a cabo el proceso de adecuación de las condiciones. Para esto se evaluaron los parámetros de concentración de ión Mg2+, de dNTP, y la relación de concentraciones de cebador y ADN por variación en la concentración de este último. La concentración de ión Mg2+ fue el parámetro que mayor influencia exhibió sobre el número y la calidad de las bandas resueltas por electroforesis.
Posteriormente se evaluaron en su capacidad de generar polimorfismos los 10 cebadores que generaron el mayor número de bandas (OPR-01, OPR-02, OPR-03, OPR-07, OPR-08, OPR-09, OPR-10, OPR-12, OPR-15 y OPR-20) con 6 materiales de T. grandiflorum y 4 de T. bicolor, y de éstos se utilizaron los 5 cebadores que mostraron mayor
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
19
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
||||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 15-24
|
||||
|
|
||||
|
número de bandas polimórficas para la caracterización molecular de los materiales del banco (tabla 2).
Caracterización molecular mediante RAPD. A partir de los 145 materiales analizados pertenecientes al banco de germoplasma, se evaluaron 114 bandas distribuidas así: 57 bandas para T. grandiflorum (84,2% polimórficas), 45 bandas para T. bicolor (26,7% polimórficas) y 12 bandas compartidas entre
 |
las dos especies (58.3% polimórficas). En la tabla 2 se presenta el número de bandas analizadas para cada uno de los cebadores con los que se evaluó la colección.
Los individuos externos presentaron patrones de bandeo similares a los del banco, con excepción de 2 bandas presentes exclusivamente en el material GOL1 y 1 banda exclusiva para GLS, ambos procedentes de Leticia (departamento de Amazonas, Colombia).
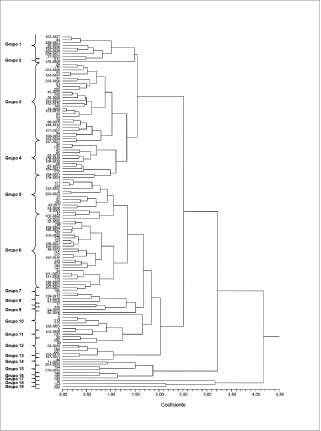
Como se observa en el dendrograma generado por UPGMA (figura 1) derivado de los valores de semejanza según Dice (1945), la totalidad de los materiales analizados se agrupó en un valor de semejanza de 0,23. Los materiales de la especie T. grandiflorum constituyen una primera rama agrupada con un valor de semejanza de 0,70; y los de T. bicolor la segunda rama agrupada en 0,93.
Estos valores son comparables a los presentados por Wilde et al. (1992). Para esto se construyó un dendrograma mediante UPGMA a partir de los valores de semejanza reportados por este autor, los cuales fueron calculados por el coeficiente de Dice (1945) utilizando marcadores RAPD para materiales de T. cacao. Los materiales de T. cacao utilizados se agruparon en valores cercanos a 0,65, y éstos, con respecto a un material de T. microcar-pum, en 0,32.
Si bien la totalidad de los individuos de T. grandiflorum analizados se agrupó en valores de 0,70, los materiales conservados en el banco lo hicieron sobre valores de semejanza cercanos a 0,75; el material denominado GG (proveniente del departamento de Guainía) y utilizado como externo al banco presentó menor semejanza con el resto de la colección. Los materiales provenientes de Leticia (departamento del Amazonas), se agruparon junto con los materiales provenientes del banco.
|
|||
|
|
||||
|
Figura 1. Dendrograma UPGMA (r =0,99439) derivado de los valores de semejanza estimada mediante el índice de Dice (1945) utilizando marcadores RAPD de los materiales del banco de germoplasma del género Theobroma.
|
A pesar de la alta semejanza intraes-pecífica encontrada, particularmente en T. bicolor, los resultados obtenidos por la
|
|||
|
|
||||
|
20
|
||||
|
|
||||
|
|
||||
|
CARACTERIZACIÓN MOLECULAR DE UN BANCO DE GERMOPLASMA DEL GÉNERO Theobroma
|
||||
|
|
||||
|
técnica RAPD permitieron la distinción del 99% de los materiales de la colección con base en los perfiles de bandas generados. Sólo dos materiales de T. bicolor (MB3-3 y MB3-5) presentaron patrones similares de bandeo.
La alta semejanza encontrada entre los materiales T. bicolor (0,93-1,00) posiblemente refleja el hecho de que los materiales tienen la misma procedencia ubicada en la zona geográfica del municipio de Calamar (departamento de Guaviare); sin embargo, el material BLS proveniente de Leticia (departamento del Amazonas), utilizado como contraste de los materiales conservados no mostró una separación evidente del conjunto de los individuos analizados. Esto muestra una segunda posibilidad: que la utilización de marcadores RAPD no es eficiente para detectar diferencias a nivel del genoma de esta especie debido a la baja frecuencia de los polimorfismos detectados y, por tanto, es necesaria la utilización de otro tipo de marcadores, por ejemplo AFLP (Amplified Fragment Length Polymorphism). Caso similar al de Perry et al. (1998), quien muestra que la utilización de marcadores RAPD para T. cacao, colectados en Malasia, fue poco satisfactoria para discriminar entre variedades de esta especie a diferencia de los marcadores AFLP.
|
conformación de 12 diferentes grupos (Vargas y Argüelles, 2000; Vargas et al., 2002). Sin embargo, la ausencia de agrupamientos claros a partir del cálculo de semejanza mediante el uso de marcadores RAPD (figura 1) respecto a los resultados de la caracterización morfoagronómica, muestra la heterogeneidad a nivel molecular de los 12 grupos mor-foagronómicos conformados previamente a este trabajo. Esto puede deberse a que los caracteres morfológicos y agronómicos que fueron tenidos en cuenta para la evaluación inicial del banco son: 1) influenciables por factores ambientales, o 2) no reflejan la variabilidad presente en el banco o 3) que no fueron suficientes para una evaluación exhaustiva de éste.
Con los datos obtenidos únicamente para los materiales de T. grandiflorum del banco de germoplasma se realizó un análisis de componentes principales con la finalidad de apreciar relaciones entre materiales que en el dendrograma no son evidentes y generar agrupamientos.
La representación en tres dimensiones de los componentes principales (figura 2), que corresponde al 74% de la variación total, muestra las posibles
|
|||
|
|
||||
|
Estos resultados difieren, seguramente por la selección de materiales evaluados, de los obtenidos previamente por Wilde et al. (1992) quien mostró que el uso de los marcadores RAPD para la caracterización de clones de cacao es una alternativa viable para la labor de curación de colecciones de germoplasma de esta especie, siendo utilizados posteriormente en trabajos como los de Lerceteau, et al. (1997a, 1997b). De esta manera, es de particular importancia para el caso de los materiales de T. bicolor, la realización de nuevas colectas de materiales pertenecientes a poblaciones ya sean cultivadas o silvestres, y comparar los datos obtenidos en este trabajo por marcadores RAPD con otro tipo de marcador molecular.
|
 |
|||
|
|
||||
|
Figura 2. Representación de los 3 componentes principales de variación (74%) obte-
Los trabajos de caracterización nidos por el análisis de componentes principales (ACP) de marcadores RAPD en 128 morfoagronómicos en varios materia- materiales de Theobroma grandiflorum del banco de germoplasma custodiado por el les de T. grandiflorum demostraron la Instituto Sinchi.
|
||||
|
|
||||
|
21
|
||||
|
|
||||
|
|
||||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 15-24
|
||||
|
|
||||
|
relaciones obtenidas en estos materiales. Con el fin de evidenciar con mayor claridad las relaciones entre individuos obtenidos mediante este análisis, se generó un dendrograma UPGMA a partir del cálculo de las distancias de cada pareja de puntos en la representación. Para esto se construyó previamente una matriz de distancias euclidianas a partir de los datos de los tres componentes principales representados. La necesidad de utilizar este procedimiento se derivó de la dificultad de generar los agru-pamientos en la representación tridimensional tanto
 |
por la naturaleza e información disponible sobre los individuos analizados (p. ej. datos de pasaporte), como por el número de puntos (individuos) sobre la representación.
En la figura 3 se presenta el dendrograma generado, el cual muestra en otro tipo de representación gráfica los resultados obtenidos mediante el análisis de componentes principales (figura 2). Con la información presentada de esta manera se clasificaron los materiales del banco dentro de 19 grupos, los cuales tampoco muestran relación con los obtenidos mediante la caracterización morfoagronómica. Se espera que el uso conjunto de los grupos obtenidos por metodologías diferentes sea un criterio de utilidad para la toma de decisiones en trabajos posteriores, bien sea en tareas relacionadas con el manejo del banco, o con miras a establecer un programa de mejoramiento genético para esta especie.
CONCLUSIONES
A partir del análisis de marcadores RAPD, se logró la caracterización de 145 materiales del banco de germoplasma del género Theobroma custodiado por el instituto Sinchi. Para esto fue necesario adecuar previamente las condiciones para la extracción de ADN y de generación de estos marcadores, con lo cual se asegura la aplicabilidad de la técnica en el laboratorio y su utilidad para responder al objetivo planteado inicialmente.
|
|||
|
|
||||
|
Figura 3. Dendrograma UPGMA derivado de la matriz de distancias euclidianas calculadas entre los puntos de la proyección de los 3 componentes principales de variación, obtenido mediante el ACP de marcadores RAPD para los materiales de Theobroma grandiflorum del banco de germoplasma custodiado por el Instituto Sinchi.
|
Los resultados presentados muestran que la técnica empleada fue útil en cuanto a la discriminación de los materiales conservados, particularmente en el caso de T. grandiflorum. Sin embargo, es conveniente comparar los resultados obtenidos
|
|||
|
|
||||
|
22
|
||||
|
|
||||
|
|
|||
|
CARACTERIZACIÓN MOLECULAR DE UN BANCO DE GERMOPLASMA DEL GÉNERO Theobroma
|
|||
|
|
|||
|
para T. bicolor con los generados mediante otro tipo de marcador molecular con el fin de evaluar la eficiencia del uso de RAPD para detectar diferencias a nivel del genoma de esta especie.
Si bien no se encontró una relación directa entre el análisis molecular con el obtenido por medio de la caracterización morfológica y agronómica, y no se encontraron indicios de diferenciación con las procedencias de los materiales, la implementación de la generación de marcadores RAPD, como complemento a la información que se tiene del banco, es una herramienta muy útil en la generación de criterios para su manejo, enriquecimiento y uso en tareas relacionadas con el manejo de estas especies.
AGRADECIMIENTOS
Los autores desean expresar sus agradecimientos a la doctora Esperanza Torres Rojas, subdirectora científica y tecnológica del Instituto Sinchi por su apoyo durante la realización de este trabajo. Igualmente al doctor Dairon Cárdenas y al grupo de trabajo del Herbario Amazónico por la consecución de los materiales utilizados como contrastes. Este trabajo se realizó en el marco del proyecto titulado "Oferta y potencialidades de un banco de germo-plasma del género Theobroma en el enriquecimiento de los sistemas productivos de la región amazónica" financiado con apoyo de Colciencias, contrato No. 582.
BIBLIOGRAFÍA
Alves, R. M.; Artero, A. S.; Sebbenn, A.; Figueira, A. 2003. Ma-ting system in a natural population of Theobroma gran-diflorum (Wild. ex Spreng.) Schum., by microsatellite markers. Genet. Mol. Biol. 3: 373-379.
Casas, A. 1995. Análisis de crecimiento del fruto y determinación del momento óptimo de cosecha del maraco (Theobroma bicolor H.B.K.). Tesis ingeniero agrónomo. Bogotá, Colombia: Universidad Nacional de Colombia. Facultad de Agronomía. 105 p.
Charters, Y. M. y Wilkinson, M. J. 2000. The use of self-pollinated progenies as "in-groups" for the genetic characterization of cocoa germplasm. Theor. Appl. Genet. 100: 160-166.
|
Couch, J. A.; Fritz, P. J. (1990). Isolation of DNA from plant high in polyphenolics. Plant. Mol. Biol. Rep. 8: 8-12.
Dellaporta, S. L.;Wood, J.; Hicks, J. B. 1983. A plant DNA mi-nipreparation: Version II. Plant. Mol. Biol. Rep. 1:19-21.
Dice, L. R. 1945. Measures of theamountof ecologicassocia-tion between species. Ecology. 26:297-302.
Doyle, J. J. y Doyle, J. L. 1990. Isolation of plant DNA from fresh tissue. Focus. 12: 13-15.
Hansen, M.; Halldén, C.; Torbjörn, S. (1998). Error rates and polymorphism frequencies for three RAPD protocols. Plant. Mol. Biol. Rep. 16:139-146.
Keb-Llanes, M.; González, G.; Chi-Manzanero, B.; Infante, D. (2002). A rapid and simple meted for small-scale DNA extraction in Agavaceae and other tropical plants. Plant. Mol. Biol. Rep. 20: 299a-299e.
Leal, F.; Serra, A.; Valderrama, E. 2000. El copoazú (Theobroma grandiflorum) Sterculiaceae, pariente del cacao. En: Memorias I congreso Venezolano del cacao y su industria. CONICIT. p. 268-274.
Lerceteau, E.; Quiroz, J.; Soria, J. Sophie, F. Pétiard, V.; Crou-zillat, D. 1997a. Genetic differentiation among Ecuado-rian Theobroma cacao L. accessions using DNA and morphological analyses. Euphytica. 95: 77-87.
Lerceteau, E.; Robert, T.; Pétiard, V.; Crouzillat, D. 1997b. Evaluation of the extent of genetic variability among Theobroma cacao accessions using RAPD and RFLP markers. Theor. Appl. Genet. 95: 10-19.
Mueller, U.G.; Wolfenbarger, L. L. 1999.AFLPgenotypingand fingerprinting. Trends. Ecol. Evol.10: 389-394.
Perry, M. D.; Davey, M. R.; Power, J. B.; Lowe, K. C.; Bligh, H. F. J.; Roach, P. S.; Jones, C. 1998. DNA isolation and AFLP™ genetic fingerprint of Theobroma cacao (L.). Plant. Mol. Biol. Rep. 16: 49-59.
Szmidt, A. 2004. MPGL-DNA extraction from recalcitrant plant. Última actualización: 06/04/2004. En: http://genetics.biology.kyushu-u.ac.jp/pgen/method /dnaextraction.htm
Stein, A. y Raoult, D. 1992. A simple method for amplification of DNA from paraffin-embedded tissues. Nucl. Acids. Res. 20: 5237-5238.
Urano de Carvalho, J. E.; Müller, C. H.; Benchimol, R. L.; Kuozo, A.; Alves, R. M. 1999. Copoasu [Theobroma grandiflorum (Willd. Ex Spreng.) Shum.]: cultivo y utilización. Belém, Brasil: Tratado de cooperación amazónica. 152 p.
Vargas Ávila, G.; Argüelles Cárdenas, J. 2000. Clasificación y caracterización morfoagronómica del germoplasma de cinco especies frutales amazónicas. Instituto Amazónico de Investigaciones Científicas (Sinchi), San Jose del Guaviare, Colombia. 28 p.
|
||
|
|
|||
|
23
|
|||
|
|
|||
|
|
|||
|
REVISTA COLOMBIANA DE BIOTECNOLOGÍA VOL.VI No. 2 Diciembre 2004 15-24
|
|||
|
|
|||
|
Vargas Ávila, G.; Suárez Suárez, S.; Cárdenas López, D. 2002. Descripción taxonómica de nueve especies frutales establecidas en el banco de germoplasma del Instituto Sinchi. San José del Guaviare, Colombia. 44
P-
Welsh, J.; McClelland, M. 1990. Fingerprinting genomes using PCR with arbitrary primers. Nucl. Acids. Res. 18: 7213-7218.
|
Wilde, J.; Waugh, R.; Powell, W. 1992. Genetic fingerprinting of Theobroma clones using randomly amplified poly-morphic DNA markers. Theor. Appl. Genet. 83: 871-877.
Williams, J. G. K.; Kubelik, A. R.; Livak, K. J.; Rafalski, J. A.; Tingey, S. V. 1990. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers. Nucl. Acids. Res. 18: 6531-6535.
|
||
|
|
|||
 |
|||
|
|
|||
|
24
|
|||
|
|
|||
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2004 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).


















