Las proteínas B3 y PDI aisladas de plaquetas humanas se unen con el rotavirus ECwt in vitro
β3 and PDI proteins isolated from human platelets bind with ECwt rotavirus in vitro
Palabras clave:
proteínas, plaquetas, rotavirus, electroforesis. (es)proteins, blood platelets, rotavirus, electrophoresis. (en)
Antecedentes. En la actualidad, no se dispone de integrina β3 de manera comercial y la PDI comercial tiene costos muy altos, lo cual dificulta el acceso a estas dos proteínas para realizar estudios conducentes a establecer si β3 y PDI interactúan con cepas de rotavirus silvestres. Objetivo. Explorar una metodología que permitiese aislar las proteínas β3 y PDI a partir de plaquetas humanas para generar anticuerpos policlonales en conejo contra la integrina β3 y evaluar la interacción entre las proteínas PDI y β3 con el rotavirus ECwt.
Material y métodos. Mediante la técnica de electroforesis preparativa en condiciones reductoras, se separaron las proteínas de un lisado de plaquetas humanas y posteriormente se electroeluyeron. Mediante las técnicas de coinmunoprecipitación, "Western blotting" y ELISA de captura se analizó la interacción del rotavirus ECwt con las proteínas β3, y PDI.
Resultados. Las proteínas totales de un lisado de plaquetas humanas se separaron mediante electroforesis en condiciones reductoras, se identificaron las proteínas β3 y PDI en un segmento del gel, utilizando anticuerpos comerciales en "Western blotting" y luego se aislaron estas dos proteínas del resto del gel. Posteriormente las proteínas se electroeluyeron del segmento del gel y se analizó su pureza. La proteína β3 se utilizó para generar anticuerpos policlonales en conejo. Igualmente, β3 y PDI eluidas se incubaron con el rotavirus ECwt y mediante la técnica de co-inmunoprecipitación y ELISA de captura encontramos que estas dos proteínas se unen in vitro. Esta misma unión se observó cuando se incubó las vellosidades aisladas de intestino delgado de ratón lactante con el rotavirus.
Conclusión. Se logró purificar parcialmente a partir de plaquetas humanas, utilizando electroforesis preparativa, cantidades relativamente altas de proteína β3 y PDI. El aislamiento de estas proteínas nos permitió generar un anticuerpo policlonal contra β3 y establecer que β3 y PDI se unen in vitro, luego de incubar las proteínas aisladas con el rotavirus ECwt, e in vivo, después de incubar el rotavirus con las vellosidades aisladas del intestino delgado de ratón lactante de la cepa ICR.
Background. Commercial integrin β3 is currently not available and commercial PDI is too expensive, which is making access difficult to these proteins needed for conducting experiments aimed at the establishment of possible interactions between integrin β3 and PDI and wild type rotavirus strains.
Objective. To explore a methodology allowing isolation of proteins β3 and PDI from human platelets to be used as antigens in the generation of rabbit polyclonal antibodies useful in the assessment of interactions between these proteins and rotavirus ECwt.
Materials and methods. Proteins β3 and PDI from human platelet lysates were separated using preparative electrophoresis under reducing conditions and then eluted. Interactions of these proteins with rotavirus ECwt were analyzed using co-immunoprecipitation, Western blotting and capture ELISA.
Results. Proteins from human platelet lysates were separated by preparative electrophoresis under reducing conditions. The identification of proteins β3 and PDI present in a gel slice was performed through their reaction with commercial antibodies in a Western blotting analysis. Protein purity was established after electroelution from a gel slice. Polyclonal antibodies against protein β3 were generated in rabbit. Incubation of eluted proteins β3 and PDI with rotavirus ECwt showed in co-immunoprecipitation and ELISA assays that these proteins bound virus in vitro. The same binding was showed to occur when rotavirus was incubated with isolated small intestinal villi from suckling mice.
Conclusions. Relatively high amounts of proteins β3 and PDI were partially purified from human platelets by preparative electrophoresis. The isolation of these proteins allowed the generation of polyclonal antibodies against β3 in addition to the establishment of the in vitro interaction of proteins β3 and PDI with rotavirus ECwt. This interaction was also demonstrated in vivo after incubating the virus with isolated small intestinal villi from suckling mice.
INVESTIGACIÓN ORIGINAL
LAS PROTEÍNAS β3 Y PDI AISLADAS DE PLAQUETAS HUMANAS SE UNEN CON EL ROTAVIRUS ECwt IN VITRO
β3 and PDI proteins isolated from human platelets bind with ECwt rotavirus in vitro
Diana Mayorga1, Linda Rubio1 Carlos A Guerrero-Fonseca2, Orlando Acosta-Losada3
1Estudiante de Biología, Universidad Distrital Francisco José de Caldas, Facultad de Ciencias y Educación.2PhD, MSc, MD, Profesor Asociado, Unidad de Bioquímica, Facultad de Medicina, Universidad Nacional de Colombia, Bogotá.
3PhD, MSc, MD, Profesor Asociado, Unidad de Bioquímica, Facultad de Medicina, Universidad Nacional de Colombia, Bogotá. Correspondencia: caguerrerof@unal.edu.co
Recibido:20100308 Enviado a pares: 20100427 Aceptado publicación: 20100514
Resumen
Antecedentes. En la actualidad, no se dispone de integrina β3 de manera comercial y la PDI comercial tiene costos muy altos, lo cual dificulta el acceso a estas dos proteínas para realizar estudios conducentes a establecer si β3 y PDI interactúan con cepas de rotavirus silvestres. Objetivo. Explorar una metodología que permitiese aislar las proteínas β3 y PDI a partir de plaquetas humanas para generar anticuerpos policlonales en conejo contra la integrina β3 y evaluar la interacción entre las proteínas PDI y β3 con el rotavirus ECwt.
Material y métodos. Mediante la técnica de electroforesis preparativa en condiciones reductoras, se separaron las proteínas de un lisado de plaquetas humanas y posteriormente se electroeluyeron. Mediante las técnicas de coinmunoprecipitación, "Western blotting" y ELISA de captura se analizó la interacción del rotavirus ECwt con las proteínas β3, y PDI.
Resultados. Las proteínas totales de un lisado de plaquetas humanas se separaron mediante electroforesis en condiciones reductoras, se identificaron las proteínas β3 y PDI en un segmento del gel, utilizando anticuerpos comerciales en "Western blotting" y luego se aislaron estas dos proteínas del resto del gel. Posteriormente las proteínas se electroeluyeron del segmento del gel y se analizó su pureza. La proteína β3 se utilizó para generar anticuerpos policlonales en conejo. Igualmente, β3 y PDI eluidas se incubaron con el rotavirus ECwt y mediante la técnica de co-inmunoprecipitación y ELISA de captura encontramos que estas dos proteínas se unen in vitro. Esta misma unión se observó cuando se incubó las vellosidades aisladas de intestino delgado de ratón lactante con el rotavirus.
Conclusión. Se logró purificar parcialmente a partir de plaquetas humanas, utilizando electroforesis preparativa, cantidades relativamente altas de proteína β3 y PDI. El aislamiento de estas proteínas nos permitió generar un anticuerpo policlonal contra β3 y establecer que β3 y PDI se unen in vitro, luego de incubar las proteínas aisladas con el rotavirus ECwt, e in vivo, después de incubar el rotavirus con las vellosidades aisladas del intestino delgado de ratón lactante de la cepa ICR.
Palabras clave: proteínas, plaquetas, rotavirus, electroforesis.
Mayorga D, Rubio L, Guerrero-Fonseca CA, Acosta- Losada O. Las proteínas β3 y PDI aisladas de plaquetas humanas se unen con el rotavirus ECwt in vitro. rev.fac.med. 2010; 58: 115-127.
Summary
Background. Commercial integrin β3 is currently not available and commercial PDI is too expensive, which is making access difficult to these proteins needed for conducting experiments aimed at the establishment of possible interactions between integrin β3 and PDI and wild type rotavirus strains.
Objective. To explore a methodology allowing isolation of proteins β3 and PDI from human platelets to be used as antigens in the generation of rabbit polyclonal antibodies useful in the assessment of interactions between these proteins and rotavirus ECwt.
Materials and methods. Proteins β3 and PDI from human platelet lysates were separated using preparative electrophoresis under reducing conditions and then eluted. Interactions of these proteins with rotavirus ECwt were analyzed using co-immunoprecipitation, Western blotting and capture ELISA.
Results. Proteins from human platelet lysates were separated by preparative electrophoresis under reducing conditions. The identification of proteins β3 and PDI present in a gel slice was performed through their reaction with commercial antibodies in a Western blotting analysis. Protein purity was established after electroelution from a gel slice. Polyclonal antibodies against protein β3 were generated in rabbit. Incubation of eluted proteins β3 and PDI with rotavirus ECwt showed in co-immunoprecipitation and ELISA assays that these proteins bound virus in vitro. The same binding was showed to occur when rotavirus was incubated with isolated small intestinal villi from suckling mice.
Conclusions. Relatively high amounts of proteins β3 and PDI were partially purified from human platelets by preparative electrophoresis. The isolation of these proteins allowed the generation of polyclonal antibodies against β3 in addition to the establishment of the in vitro interaction of proteins β3 and PDI with rotavirus ECwt. This interaction was also demonstrated in vivo after incubating the virus with isolated small intestinal villi from suckling mice.
Key word: proteins, blood platelets, rotavirus, electrophoresis.
Mayorga D, Rubio L, Guerrero-Fonseca CA, Acosta- Losada O. β3 and PDI proteins isolated from human platelets bind with ECwt rotavirus in vitro. rev.fac.med. 2010; 58: 115-127.
Introducción
Los rotavirus constituyen la primera causa de gastroenteritis aguda en niños menores de cinco años, presentando la más alta frecuencia de hospitalizaciones entre los seis y los 24 meses de edad. La deshidratación ocasionada es la causa más significativa de muerte en los infantes de países en desarrollo, fluctuando la mortalidad a nivel mundial entre 454.000 y 705.000 (1, 2). Se considera que la infección es especifica de especie, dado que no se ha documentado la transmisión de rotavirus entre el hombre y animales; sin embargo, las cepas aisladas de humanos tienen una alta homología genética con las cepas aisladas de animales (3) y al co-infectar una célula con dos cepas distintas de rotavirus, la progenie viral resultante es una población de virus que contiene diferentes combinaciones de los diferentes segmentos genómicos parentales. Recientemente se introdujeron dos vacunas (RotaTeq, Rotarix) (2,4); sin embargo, el desarrollo de estas vacunas (virus vivos atenuados) ha suscitado algunos interrogantes sobre su costo, eficacia, seguridad (5) y riesgos de transmisión (6).
Los rotavirus pertenecen a la familia Reoviridae, carecen de cubierta lipídica (7,8) y contienen un genoma de dsRNA distribuido en 11 segmentos. Las proteínas estructurales (VP1-VP4, VP6 y Vp7) de la partícula viral se disponen en tres capas concéntricas (9-12). Las partículas virales con las tres capas proteicas concéntricas (TLPs) son infecciosas, mientras que aquellas no infecciosas poseen sólo dos capas (DLPs).
Se ha propuesto que el receptor funcional para rotavirus corresponde a un complejo de varias macromoléculas que incluye, entre otras, las integrinas avβ3 (13,14,15), a2b1 (16-20), axb2 (17,18) y la proteína de choque térmico HSC70 (14,21-24), presente en la superficie de células MA104. Recientemente, en un estudio realizado en nuestro laboratorio (25) se encontró que la proteína disulfuro isomerasa (PDI) también participa en los mecanismo de entrada de los rotavirus a células MA104, sugiriendo la existencia de posibles vías alternativas para la entrada de los rotavirus a diferentes células hospederas. Por otra parte, las plaquetas humanas expresan las integrinas aVβ3 y la aIIbβ3 en una buena cantidad (80.000 copias aproximadamente) (26). De igual forma, la proteína chaperona PDI, también se encuentra en buena cantidad sobre la membrana de las plaquetas y está implicada en la agregación plaquetaria (27).
En el presente trabajo nos propusimos examinar si la alta concentración de las proteínas β3 y PDI en plaquetas facilita su aislamiento a través de las técnicas de electroforesis preparativa y electroelución, con el objeto de obtener antígeno para generar anticuerpos contra la integrina β3 y utilizar la proteína PDI para estudiar posibles interacciones con la partícula del rotavirus ECwt. Adicionalmente, analizamos la interacción del rotavirus ECwt con integrina β3 y PDI de las vellosidades aisladas del intestino delgado de ratón lactante ICR.
Material y métodos
Animales, virus, anticuerpos y plaquetas
Se utilizaron ratones lactantes de 10-12 días de la cepa ICR obtenidos del bioterio del Instituto Nacional de Salud (Bogotá, Colombia) que se sacrificaron mediante el método de dislocación cervical (28). El rotavirus empleado fue la cepa ECwt o EDIM tipo silvestre de ratón, donada por el Dr. M. Franco del Instituto de Genética de la Pontificia Universidad Javeriana (Bogotá, Colombia).
Se usaron anticuerpos (Ac) comerciales policlonales contra las proteínas β3 (SC6627, Santa Cruz) y PDI (SC17222. Santa Cruz). Se utilizaron anticuerpos policlonales anti-β3 generados en nuestro laboratorio (conejos), utilizando un antígeno β3 aislado de plaquetas, y contra proteínas estructurales del rotavirus ECwt. Las plaquetas vencidas fueron donadas por el Hospital la Samaritana (Bogotá, Colombia). Las bolsas con plaquetas se mantuvieron congeladas a -70°C y se descongelaron solo para su uso.
Lisado de plaquetas y preparación de la muestra para electroforesis
Aproximadamente 8.7 x 1010 plaquetas suspendidas en plasma (50 ml) se lisaron con RIPA (150 mM NaCl, Nonident-40 1%, DOC 0.5%, SDS 0.1%, 50 mM Tris-HCl, pH 8) a 37 °C por 20 min. El precipitado de 2760 x g fue descartado y el sobrenadante sometido a precipitación con polietilenglicol 8000 al 10% en agitación suave toda la noche. El precipitado se colectó a 13800 x g por 30 min y se resuspendió en buffer de Laemmli (15 ml) en condiciones reductoras y desnaturalizantes (0.065 M Tris-HCl, pH 6.8; 2% (p/v) SDS; 5% (v/ v) 2 beta-mercaptoetanol; 2 mM EDTA; 10% (v/ v) glicerol y 0.1% azul de bromofenol, seguido de incubación en agua hirviendo por 3 min), alternativamente, el precipitado en el mismo buffer no fue incubado en agua hirviendo.
Electroforesis SDS-PAGE
El sistema SDS-PAGE fue como se describió previamente (29). Tanto el gel de concentración (5%) como de separación (10%) se suplementaron con 1mM EDTA. La electroforesis se condujo a 20 mA por lámina de gel (8 x 7 x 0.75 cm). Los geles se tiñeron con azul de Coomassie G-260 al 0.15%. Para propósitos analíticos se aplicaron muestras (1, 2, 4, 8, 16 µl) del lisado de plaquetas al gel de 12 bolsillos. Para fines preparativos se utilizó un gel de un solo bolsillo con un de espesor de 1.5 mm.
Western blotting (WB)
Las proteínas de un segmento (0.5 cm de ancho) del gel se transfirieron a una membrana de PVDF previamente activada con metanol por 15 segundos. La transferencia se hizo en condiciones semi-secas a 32mA durante 10 horas o a 300 mA por 2 horas, según lo recomendado por el fabricante (minicamara BIORAD). La membrana se bloqueó con leche descremada al 5%, se agregó anticuerpo primario contra β3 o PDI (0.2 µg/ml, Santa Cruz). Como anticuerpo secundario se utilizó un conjugado con peroxidasa (0.1 µg/ml, Santa Cruz). Se visualizó con Luminol (Santa Cruz) o con Amino-etilcarbazol (AEC) (0.25 mg/ml) en buffer de acetato de sodio 50 mM, pH 5.2 y peroxido de hidrógeno al 0.04%. Las posiciones de las bandas identificadas de β3 o PDI en el "blot" fueron utilizadas para determinar los sitios de corte en el gel preparativo. Los segmentos de gel (0.5 cm) cortados fueron conservados a -20°C hasta su electroelución.
Electroelución
Los segmentos de gel se electroeluyeron en buffer (25 mM Tris, 192 mM glicina y SDS 0.1%) a 300 V durante cinco horas, a temperatura no mayor a 50 °C dentro de la cámara. La muestra electroeluida se recolectó para semi-cuantificarse en SDS-PAGE comparando con bandas de albumina de concentración conocida.
Producción del anticuerpo policlonal anti-β3
La proteína β3 electroeluida fue sometida nuevamente a electroforesis en gel en condiciones desnaturalizantes. La banda del gel correspondiente a la integrina β3 fue cortada, macerada en PBS e inoculada (1mg) cada 15 días, tres veces, vía subcutánea, a un conejo Nueva Zelanda. Quince días después de la última aplicación, el conejo se sacrificó. La sangre se mantuvo a de 37°C, por 30 minutos y luego se dejó toda la noche a 4°C. El suero recuperado se mezcló con glicerol 1:1 (v/v), con azida de sodio al 0.02% y se almacenó a -20°C hasta su uso.
Alquilación de proteínas
Para estabilizar los grupos sulfidrilo de la integrina β3 y de PDI, se empleo la técnica de Lane (1978). Las proteínas fueron mezcladas con 1/3 de 0.06 M (v/v) Tris-HCl, pH 8.8, SDS 2%, 2 mM EDTA, glicerol 10% y 1/50 (v/v) de 0.25 M ditiotreitol recién preparado. Las muestras fueron hervidas durante tres minutos, enfriadas y mezcladas con 1/20 (v/v) de iodoacetamida 0.25M y mantenidas a 50°C por 15 minutos. Las proteínas fueron analizadas en SDS-PAGE como se indicó arriba.
Análisis de la unión in vitro de las proteínas electroeluidas β3 y PDI con el virus ECwt
Se incubó el electroeluido de β3 o PDI con TLPs de la cepa rotaviral ECwt, preincubada con tripsina (1µg/ml) toda la noche a 4°C. A cada uno de los complejos β3-ECwt o PDI-ECwt, se le adicionó anticuerpo policlonal anti-ECwt, generado en conejo en el laboratorio y se incubó 1 h a 37°C en agitación suave. Luego se adicionó proteína A-Sefarosa (Sigma, 5µl/100 µl) al complejo virus-proteína-anticuerpo. El inmunoprecipitado se lavó con PBS y fue analizado en SDS-PAGE y "Western blotting". A la membrana se le adicionó anticuerpo anti-PDI (0.2 µg/ml, Santa cruz) y anti-β3 generados en nuestro laboratorio (1:1000). Como anticuerpo secundario se empleó un conjugado con peroxidasa (0.1 µg/ml, Santa Cruz) y se reveló con Luminol (Santa Cruz) o AEC.
La interacción de β3 o PDI con TLPs se examinó también con ELISA de captura. Como anticuerpo de captura se utilizó anti-ECwt generado en cobayo (1:250). Se bloqueó con leche descremada al 5% y see adicionó el complejo ECwt-β3 o ECwt-PDI, en diferentes concentraciones. Como anticuerpo primario se utilizó anti- PDI o anti-β3 y como anticuerpo secundario un conjugado con peroxidasa (0.3 µg/ml, Santa Cruz). Se reveló con peroxidasa en buffer de citrato, utilizando el sustrato (OPD). Se leyó a 492 nm.
Aislamiento de vellosidades intestinales
Para la extracción de las vellosidades del intestino delgado de ratón lactante, se siguió el protocolo estandarizado y desarrollado en nuestro laboratorio (29). Brevemente, se sacrificaron los animales por dislocación cervical, se retiró el intestino, se lavó con medio de cultivo, se corto en fragmentos de 5mm y se extrajeron las vellosidades con 5 ml de MEM con antibiótico/ antimicótico y 1.5 mM EDTA. Las células se mantuvieron a 4°C hasta realizar la infección con rotavirus.
Evaluación de la unión in vivo de las proteínas integrina β3 y PDI en vellosidades intestinales con el virus ECwt
Para determinar la unión del rotavirus ECwt a PDI y β3 de las vellosidades aisladas del intestino delgado, el rotavirus se incubó durante 45 minutos a 4°C con las vellosidades, luego se incubaron las vellosidades a 37°C durante 0, 30 y 60 min e inmediatamente fueron lisadas con buffer RIPA modificado (150 mM NaCl, Nonident- 40 1%, DOC 0.5%, 2 mM de DTT, 1% Tritón X-100, 50 mM Tris-HCl, pH 8) y se incubaron a 37°C durante 1 hora. Se co-inmunoprecipitó adicionando anticuerpo policlonal anti-ECwt, se incubó 1 h a 37°C y se continuó como se describió antes.
Resultados
Aislamiento de las proteínas β3 y PDI a partir de plaquetas humanas por las técnicas de electroforesis y electroelución.
Mediante la técnica de electroforesis en condiciones reductoras, se separaron las proteínas a partir de un lisado de plaquetas. Para identificar las bandas del gel preparativo correspondientes a PDI y β3, un segmento del gel se cortó y con este se realizo un "Western blotting" (WB). En la figura 1A se observa un segmento del gel preparativo representativo, donde se aprecian las bandas identificadas por tinción con azul de Coomassie y en el WB se puede identificar las bandas obtenidas con Acs específicos contra β3 y PDI, con un peso molecular aproximado de 90 y 55 kDa, respectivamente. Con esta técnica las bandas se cortaron del gel preparativo y se guardaron congeladas a -20°C hasta su electroelución.
Las bandas recuperadas de los diferentes geles preparativos se electroeluyeron y los diferentes electroeluidos se analizaron por electroforesis en condiciones desnaturalizantes y reductoras con el objeto de determinar su pureza
figura 1B. Se observa que las muestras correspondientes a electroeluidos de segmentos procedentes de geles preparativos tanto de PDI como de β3 tienen varias bandas de diferente peso molecular, siendo más comunes las tres bandas ubicadas por encima de 45 kDa. En general, la banda mayoritaria, designada como β3, es la ubicada inmediatamente a la altura de 70 kDa (β3 posee un peso molecular aproximado de 87- 90 kDa) mientras que en el caso de la electroelución de PDI, la banda predominante es la ubicada entre 70 kDa y 45 kDa (PDI posee un peso molecular aproximado de 55-57 kDa).Otro método utilizado para analizar la contaminación de los electroeluidos fue realizar una alquilación de las proteínas siguiendo el método de Lane. Como se observa en la figura 1C, los electroeluidos denominados PDI y β3 continúan presentando tres bandas: una prominente, a la altura de 55 kDa en el electroeluido denominado PDI, y una a la altura de 90 kDa en el electroeluido denominado β3. Sin embargo, las tres bandas se observan en todos los electroeluidos analizados, aunque varía la intensidad de cada una de las bandas.
Las bandas proteínicas que difieren de las masas moleculares esperadas (para cada una de las proteínas extraídas) puede deberse a una posible contaminación de otras proteínas, para el caso de las bandas relacionadas con PDI, o una degradación de estas durante el proceso de electroelución, en el caso de las bandas relacionadas con β3. Para diferenciar estas dos posibilidades, se realizo un "Western blotting", utilizándose, como anticuerpo primario anti-PDI y anti-β3 comerciales (0.2 ug/ml, Santa Cruz).
Como se muestra en la figura 2, de las proteínas electroeluidas pertenecientes a PDI, en el WB figura 2-B puede visualizarse 2 bandas de un peso molecular alrededor de los 55 kDa. Mientras que en el caso de las bandas relacionadas con β3, se observa, una única banda la cual posee una masa molecular aproximada de 90 kDa.
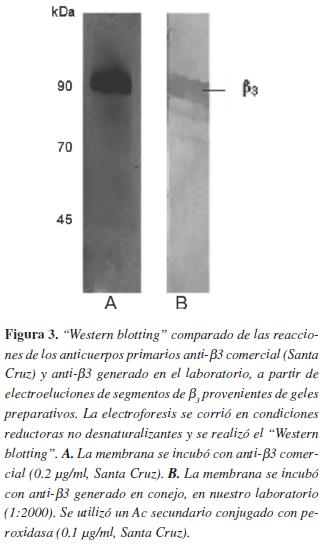
Este resultado sugiere que tanto en PDI como en β3 existe contaminación con otras proteínas, dado que las demás bandas de menor intensidad no las detectan los anticuerpos.Posteriormente, se quiso observar la diferencia que existe en el reconocimiento del anticuerpo anti-β3 generado en conejo (utilizando como antígeno la proteína electroeluida de un segmento del gel preparativo) y el anticuerpo anti-β3 de origen comercial (Santa cruz). Como se observa en la figura 3 , el anticuerpo comercial reconoce una sola banda, mientras que el generado en el laboratorio reconoce varias bandas, aunque principalmente reconoce la banda del mismo peso molecular que la observada con el anticuerpo comercial figura 3B.
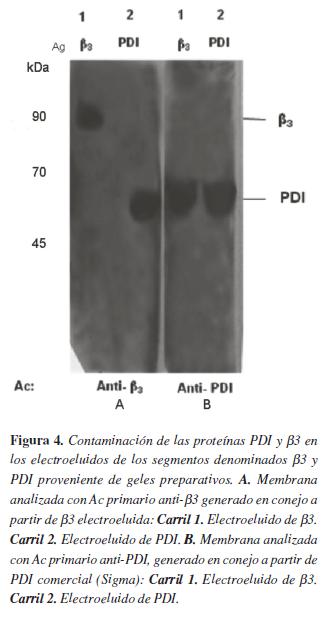
Los electroeluidos de los segmentos denominados β3 están contaminados con la proteína PDI, mientras que los electroeluidos de los segmentos denominados PDI no están contaminados con la proteína β3Para conocer si las bandas relacionadas con aquello denominado como electroeluido de β3 o PDI están contaminadas entre sí, es decir, si β3 se encuentra contaminada con PDI o viceversa, se decidió realizar un WB. Para esto, el electroeluido denominado PDI se examinó con el anticuerpo anti-β3, generado en conejo (utilizando como antígeno la proteína β3 electroeluida en este trabajo). El electroeluido denominado β3 se analizó con anticuerpo anti-PDI comercial (0.2 ug/ml, Santa cruz).
Como se observa en la figura 4-A, carril 2, en el electroeluido procedente del segmento denominado (antígeno) Ag PDI, el anticuerpo anti- β3 detecta una banda a la altura de PDI; mientras que en la figura 4-B, carril 1, el electroeluido del segmento denominado Ag β3, el anticuerpo anti-PDI no detecta banda alguna a la altura de 90 kDa, pero si una banda de 55 kDa que corresponde a PDI. Esto sugiere que el electroeluido denominado Ag β3 está contaminado con la proteína PDI. Igualmente sugiere que la fracción denominada Ag PDI no está contaminada con la proteína β3, por lo menos de manera evidente con esta técnica.
Las proteínas electroeluidas PDI y β3 se unen in vitro con el virus ECwt Para poder determinar si existe una unión de las proteínas electroeluidas β3 o PDI, con el rotavirus ECwt, se realizaron dos pruebas inmunoenzimaticas: inmunoprecipitacion y ELISA.
Para la inmunoprecipitacion se incubó PDI y/o β3 con TLPs de la cepa rotaviral ECwt (la cual se encontraba activada con tripsina) y luego se co-inmunoprecipitó con anticuerpo anti-rotavirus. Como se observa en la figura 5-A, el rotavirus ECwt co-inmunoprecipita con β3 y PDI, sugiriendo que estas proteínas se unen con el rotavirus en un sistema in vitro. Sin embargo en el carril 1 se observa una banda a la altura de 55 kDa que puede corresponder a PDI. Lo anterior es explicable si se tiene en cuenta que el electroeluido denominado β3 está contaminado con la proteína PDI. En la figura 5-B se mezclaron las dos proteínas con ECwt y se repitió el experimento. Esta figura muestra que el rotavirus se une con las proteínas cuando se encuentran "solas" o cuando se encuentran mezcladas.
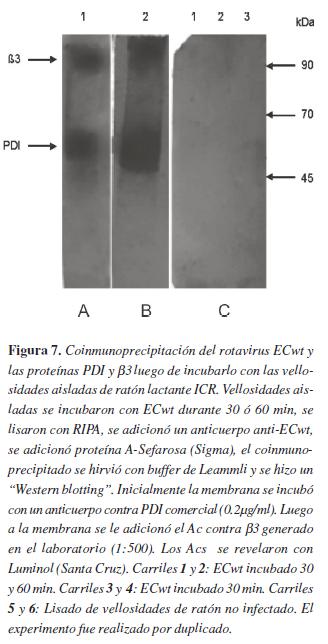
Este mismo objetivo se analizó empleando la técnica de ELISA de captura. El anticuerpo que se unió a la placa de ELISA fue anti-ECwt obtenido de cobayo en dilución 1:250. Como primer anticuerpo se usó anti-PDI generado en conejo, en dilución 1:1000 y como anticuerpo secundario, se empleó anti-conejo HRP (0.13 µg/ml, Santa cruz). Cabe aclarar que en nuestro laboratorio se toma las absorbancias inferiores a 0.1, como no confiables (línea de corte), una vez substraída la absorbancia de base del control. Como se observa en la figura 6, no hay cambios significativos en la absorbancia cuando se incuban diferentes concentraciones de virus con distintas concentraciones de proteínas β3 o PDI, se podría asumir en principio que dentro de los limites de las concentraciones de virus, de PDI o β3 utilizadas es posible que se haya conseguido una saturación en la interacción, lo que podría explicar la limitada variación en los valores de ELISA. Esto sugiere que estamos en un punto de saturación en el cual, al aumentar la concentración de proteína y disminuir la del virus o viceversa, la unión neta como promedio,se mantiene. Este resultado apoya lo observado en la coinmunoprecipitación, sugiriendo que la unión entre las proteínas PDI o β3 con el rotavirus ECwt es real y no un artefacto. Sin embargo, este resultado no permite hallar la concentración límite de proteína que se une al rotavirus, ni la concertación limite de rotavirus que se une a las proteínas. El rotavirus ECwt se une in vivo con las proteínas PDI y β3 de las vellosidades aisladas de ratón lactante ICRPara determinar si el virus ECwt se une in vivo a las proteínas PDI y β3, el rotavirus se incubó durante 30 y 60 minutos a 37°C con vellosidades aisladas de intestino de ratones lactantes ICR. Transcurridos estos tiempos las células se lisaron con RIPA, después se co-inmunoprecipitó utilizando un anticuerpo anti-ECwt, luego proteína A-Sefarosa y se realizó un "Western blotting".
En la figura 7 se observan dos bandas de aproximadamente 90 y 55 kDa en los carriles que relacionan la coinmunoprecipitación luego de incubar las vellosidades con el virus durante 30 o 60 min, sugiriendo que el rotavirus ECwt se une in vivo a PDI y β3.
DiscusiónLa integrina β3 se ha relacionado como molécula receptora para rotavirus, junto con a2b1, a4b1, axb2, a4b1, gangliósidos, HSC70 y recientemente PDI, principalmente en la línea celular MA104. Sin embargo, no se ha determinado si estas moléculas tienen el mismo papel de receptor in vivo en el enterocito con rotavirus silvestres, es decir, rotavirus no adaptados a cultivos de laboratorio. En la actualidad, no se dispone de integrina β3 de manera comercial y la PDI comercial tiene costos muy altos, lo cual dificulta el acceso a estas dos proteínas para realizar estudios conducentes a establecer si β3 y PDI interactúan con cepas de rotavirus silvestres. Por esta razón, se quiso explorar una metodología que permitiese obtener las proteínas a partir de plaquetas humanas, dado que estas dos proteínas se encuentran en forma relativamente abundante, respecto a otros tejidos.
Para aislar y purificar β3 y PDI a partir de plaquetas, examinamos las condiciones de corrido por electroforesis SDS-PAGE, específicamente el grado de separación del componente proteico de las plaquetas. Como se puede observar en la figura 1, no hay una buena separación de proteínas de las plaquetas cuando se corren en condiciones reductoras sin hervir la muestra o reductoras hirviendo la muestra. Esto se puede deber a que las plaquetas tienen alto componente de azucares en las glicoproteínas necesarias para la adherencia en la formación del trombo.
Se conoce que las proteínas altamente glicosiladas muestran un patrón de corrido en forma de barrido o presentan diferentes pesos moleculares en diferentes corridos electroforéticos. Estos cambios se presentan porque la proteína pierde azúcares en el proceso de extracción, tiene diferente grado de glicosilación o se asocia de manera diversa entre las moléculas. Igualmente, el patrón de corrido puede alterarse cuando las proteínas presentan grupos sulfidrilo, a pesar de usar agentes reductores como el 2-beta-mercaptoetanol, dado que estas pueden reasociarse durante el corrido electroforético o no reducir todos los puentes de disulfuro. Este fenómeno puede presentarse aún cuando la muestra se hierve, en presencia de SDS y 2-beta-mercaptoetanol. β3 es una proteína altamente glicosilada e igualmente PDI y β3 son proteínas con una relativa abundancia de puentes disulfuro.
Lo anterior determina que las proteínas con estas características bioquímicas, cuando se aíslan a partir de electroforesis preparativa su pureza no sea buena y en términos generales sea más bien una purificación parcial. Dicha purificación parcial se evidencia en la figura 1, donde se analiza el electroeluido luego de alquilarlo. Esta técnica permite reducir permanentemente el azufre de los grupos tiol, al modificarlo de manera covalente. Esta contaminación también se evidencia en las figuraS 2-3-4 que muestran diferencia en el patrón de bandas de β3 reconocidas por los anticuerpos comerciales y el generado en el laboratorio a partir de la proteína β3 aislada en nuestro trabajo. Igualmente, se evidencia que el electroeluido denominado β3 está contaminado con PDI, aunque PDI, aparentemente, no está contaminada con β3, pero sí con otras proteínas como se observa en la figura 4.
Sin embargo, aislar proteínas por electroforesis preparativa permite obtener cantidades relativamente altas de proteína y de fácil separación, como se puede observar en las figuraS 2 Y 3, que muestra que los segmentos identificados, cortados del gel preparativo y electroeluidos contienen las proteínas β3 y PDI dado que éstas fueron identificadas por los anticuerpos comerciales El aislamiento de estas proteínas nos permitió establecer que β3 y PDI se unen in vitro con el rotavirus silvestre de ratón ECwt. Esta conclusión la obtenemos de la figura 5, dado que al incubar las dos proteínas de manera individual o como mezcla con el rotavirus se coinmunoprecipitan con el rotavirus. Esta unión se corrobora en la técnica de ELISA de captura luego de incubar las proteínas con el rotavirus y adicionar el complejo al anticuerpo adherido a la placa figura 6 . Otra técnica empleada para confirmar que hay unión entre el rotavirus y las proteínas β3 y PDI fue la co-inmunoprecipitación de β3 y PDI luego de incubar el rotavirus con las vellosidades aisladas del intestino delgado de ratón lactante de la cepa ICR. Esta unión in vivo figura 7 , confirma lo observado en la figura 5-6, descartando que dicho fenómeno sea un artefacto, en especial por la función de chaperona de PDI. La unión in vivo de PDI y β3 a la partícula rotaviral fortalece el modelo de ingreso de rotavirus a la célula hospedera que involucra la proteína VP7 de rotavirus y la integrina avβ3, así como la implicación de la PDI en el proceso de entrada del rotavirus a la célula (25).
Conclusión
El presente trabajo contribuye originalmente al aislamiento y purificación de cantidades adecuadas de las proteínas PDI y β3, útiles en el estudio de las interacciones del rotavirus con las moléculas de superficie de la célula hospedera. En forma particular, la interacción de PDI y β3 con las proteínas estructurales del rotavirus sugiere la participación de reacciones de óxidoreducción que actuarían sobre los enlaces de disulfuro con la consecuente inducción de cambios conformacionales en las proteínas virales. Disponer de cantidades apreciables de las proteínas facilitaría enormemente los estudios básicos de los mecanismos de entrada del rotavirus y la posibilidad de la interferencia terapéutica experimental de este proceso de entrada.
Agradecimientos
El soporte financiero para realizar este proyecto fue otorgado por la División de Investigación Bogotá (DIB) código 9082 Universidad Nacional de Colombia.
Referencias
1. Parashar U, Bresee J, Glass R. Rotavirus and severe childhood diarrhea. Emerg Infect Dis. 2006; 12: 304- 306.
2. Panatto D, Amicizia D, Ansaldi F, Marocco A, Marchetti F, Bamfi F, Giacchino R, Tacchella A, Del Buono S, Gasparini R. Burden of rotavirus disease and cost- effectiveness of universal vaccination in the Province of Genoa (Northern Italy). Vaccine. 2009; [In press].
3. Nakagomi O, Nakagomi T. Genetic diversity and similarity among mammalian rotaviruses in relation to interspecies transmission of rotavirus. Arch Virol. 1991; 120:43-55.
4. Wu CL, Yang YC, Huang LM, Chen KT. Cost-effectiveness of childhood rotavirus vaccination in Taiwan. Vaccine. 2009; 27: 1492-1499.
5. Parez N. Rotavirus gastroenteritis: Why to back the development of new vaccines? Comp Immunol Microbiol Infect Dis. 2008; 31: 253-269.
6. Anderson EJ. Rotavirus vaccines: viral shedding and risk of transmission. Lancet Infect Dis. 2008; 8: 642- 649.
7. Pesavento JB, Crawford SE, Estes MK, Prasad BV. Rotavirus proteins: structure and assembly. Curr Top Microbiol Immunol. 2006; 309: 189-219.
8. Chen JZ, Settembre E, Aoki S, Dormitzer P, Bellamy R, Harrison S, Grigorieff N. High-Resolution Electron Microscopy of a Rotavirus Particle. Biophys J. 2009; 96 (Suppl 1): p 413ª.
9. Arias CF, Romero P, Alvarez V, López S. Trypsin activation pathway of rotavirus infectivity. J Virol. 1996; 70: 5832-5839.
10. Ludert JE, Krishnaney AA, Burns JW, Vo PT, Greenberg HB. Cleavage of rotavirus VP4 in vivo. J Gen Virol. 1996; 77: 391-395.
11. Benureau Y, Huet JC, Charpilienne A, Poncet D, Cohen J. Trypsin is associated with the rotavirus capsid and is activated by solubilization of outer capsid proteins. J Gen Virol. 2005; 86: 3143-3151.
12. Zarate S, Espinosa R, Romero P, Méndez E, Arias CF, López S. The VP5 domain of VP4 can mediate attachment of rotaviruses to cells. J. Virol. 2000; 74: 593-599.
13. Casola A, Estes MK, Crawford SE, Ogra PL, Ernst PB, Garofalo RP, Crowe SE. Rotavirus infection of cultured intestinal epithelial cells induces secretion of CXC and CC chemokines. Gastroenterology. 1998; 114:947-55.
14. Chouchane F, Lotfi S, Sawasdikosol R, Simpson M, Kindt T. Heat-shock proteins expressed on the surface of human T cell leukemia virus tipe-I-infected cell lines induce autoantibodies in rabbits. J. Infect. Dis 1994; 169:253-259.
15. Ciarlet M, Crawford S, Cheng E, Blutt S, Rice D, Bergelson J. Estes M. VLA-2 (alpha2Beta1) integrin promotes rotavirus entry into cells but is not necessary for rotavirus attachment. J. Virol 2002; 76: 1109-1123.
16. Ciarlet M, Crawford SE, Estes MK. Differential infection of polarized epithelial cell lines by sialic aciddependent and sialic acid-independent rotavirus strains. J Virol. 2001; 75:11834-50.
17. Clark MA, Blair H, Liang L, Brey RN, Brayden D, Hirst BH. Targeting polymerised liposome vaccine carriers to intestinal M cells. Vaccine 20: 2002; 208-217.
18. Conrad ME, Umbreit JN, Peterson RD, Moore EG, Harper KP. Function of integrin in duodenal mucosa uptake of iron. Blood 1993; 81: 517-521.
19. Coulson BS, Londrigan SL, Lee DJ. Rotavirus contains integrin ligand sequences and a disintegrin-like domain that are implicated in virus entry into cells. Proc Natl Acad Sci U S A. 1997; 94:5389-94.
20. Cuadras MA, Feigelstock DA, An S, Greenberg HB. Gene expression pattern in Caco-2 cells following rotavirus infection. J Virol. 2002; 76:4467-82.
21. Delmas O, Gardet A, Chwetzoff S, Breton M, Cohen J, Colard O, Sapin C, Trugnan G. Minireview: Different ways to reach the top of a cell. Analysis of rotavirus assembly and targeting in human intestinal cells reveals an original raft-dependent, Golgi-independent apical targeting pathway. Virology. 2004; 327:157- 161.
22. Estes M. Rotaviruses and their replication. In: Knipe DM and Howley PM (ed) Fields virology, 4th ed. Lippincott- Raven publishers. Philadelphia, 2001; pp 1747-1785.
23. Fleming FE, Graham KL, Taniguchi K, Takada Y, Coulson BS. Rotavirus-neutralizing antibodies inhibit virus binding to integrins alpha 2 beta 1 and alpha 4 beta 1. Arch Virol 2007; 152:1087-101.
24. Geyra A, Uni Z, Gal-Garber O, Guy D, Sklan D. Starving Affects CDX Gene Expression during Small Intestinal Development in the Chick. J. Nutr. 2002; 132:911-917.
25. Calderon MN, Acosta O, Guerrero CA, Guzman F. Protein disulfide isomerase activity is involved in rotavirus entry to MA104 cells. Proceedings of the XIV International Congress of Virology, Istanbul. 2008; pp. 163-164.
26. Fang J, Kairbaan H, Bryon D, Hynes R, White G, Wilcox D. Therapeutic expression of the platelet-specific integrin, aII β3, in a murine model for Glanzmann thrombasthenia. Blood. 2005; 106: 2671-2679.
27. Essex DW, Li M. Protein disulphide isomerase mediates platelet aggregation and secretion. Br J Haematol. 1999; 104: 448-454.
28. Beaver B, Reed W, Leary S, Mckiernan B, Bain F, et al. Report of the AVMA Panel on Euthanasia. J Am Vet Med Assoc. 2001; 218: 669-696.
29. Santana Y. Participación de la proteína de choque térmico HSC70, la integrina av β3 y la proteína disulfuro isomerasa en el proceso de entrada del rotavirus homólogo Ecwt y heterólogos RRV y Wa en cultivos primarios de células intestinales de ratón lactante. 2009. Tesis de Maestría, Universidad Nacional de Colombia.
Referencias
Parashar U, Bresee J, Glass R. Rotavirus and severe childhood diarrhea. Emerg Infect Dis. 2006; 12: 304- 306.
Panatto D, Amicizia D, Ansaldi F, Marocco A, Marchetti F, Bamfi F, Giacchino R, Tacchella A, Del Buono S, Gasparini R. Burden of rotavirus disease and cost- effectiveness of universal vaccination in the Province of Genoa (Northern Italy). Vaccine. 2009; [In press].
Nakagomi O, Nakagomi T. Genetic diversity and similarity among mammalian rotaviruses in relation to interspecies transmission of rotavirus. Arch Virol. 1991; 120:43-55.
Wu CL, Yang YC, Huang LM, Chen KT. Cost-effectiveness of childhood rotavirus vaccination in Taiwan. Vaccine. 2009; 27: 1492-1499.
Parez N. Rotavirus gastroenteritis: Why to back the development of new vaccines? Comp Immunol Microbiol Infect Dis. 2008; 31: 253-269.
Anderson EJ. Rotavirus vaccines: viral shedding and risk of transmission. Lancet Infect Dis. 2008; 8: 642- 649.
Pesavento JB, Crawford SE, Estes MK, Prasad BV. Rotavirus proteins: structure and assembly. Curr Top Microbiol Immunol. 2006; 309: 189-219.
Chen JZ, Settembre E, Aoki S, Dormitzer P, Bellamy R, Harrison S, Grigorieff N. High-Resolution Electron Microscopy of a Rotavirus Particle. Biophys J. 2009; 96 (Suppl 1): p 413ª.
Arias CF, Romero P, Alvarez V, López S. Trypsin activation pathway of rotavirus infectivity. J Virol. 1996; 70: 5832-5839.
Ludert JE, Krishnaney AA, Burns JW, Vo PT, Greenberg HB. Cleavage of rotavirus VP4 in vivo. J Gen Virol. 1996; 77: 391-395.
Benureau Y, Huet JC, Charpilienne A, Poncet D, Cohen J. Trypsin is associated with the rotavirus capsid and is activated by solubilization of outer capsid proteins. J Gen Virol. 2005; 86: 3143-3151.
Zarate S, Espinosa R, Romero P, Méndez E, Arias CF, López S. The VP5 domain of VP4 can mediate attachment of rotaviruses to cells. J. Virol. 2000; 74: 593-599.
Casola A, Estes MK, Crawford SE, Ogra PL, Ernst PB, Garofalo RP, Crowe SE. Rotavirus infection of cultured intestinal epithelial cells induces secretion of CXC and CC chemokines. Gastroenterology. 1998; 114:947-55.
Chouchane F, Lotfi S, Sawasdikosol R, Simpson M, Kindt T. Heat-shock proteins expressed on the surface of human T cell leukemia virus tipe-I-infected cell lines induce autoantibodies in rabbits. J. Infect. Dis 1994; 169:253-259.
Ciarlet M, Crawford S, Cheng E, Blutt S, Rice D, Bergelson J. Estes M. VLA-2 (alpha2Beta1) integrin promotes rotavirus entry into cells but is not necessary for rotavirus attachment. J. Virol 2002; 76: 1109-1123.
Ciarlet M, Crawford SE, Estes MK. Differential infection of polarized epithelial cell lines by sialic aciddependent and sialic acid-independent rotavirus strains. J Virol. 2001; 75:11834-50.
Clark MA, Blair H, Liang L, Brey RN, Brayden D, Hirst BH. Targeting polymerised liposome vaccine carriers to intestinal M cells. Vaccine 20: 2002; 208-217.
Conrad ME, Umbreit JN, Peterson RD, Moore EG, Harper KP. Function of integrin in duodenal mucosa uptake of iron. Blood 1993; 81: 517-521.
Coulson BS, Londrigan SL, Lee DJ. Rotavirus contains integrin ligand sequences and a disintegrin-like domain that are implicated in virus entry into cells. Proc Natl Acad Sci U S A. 1997; 94:5389-94.
Cuadras MA, Feigelstock DA, An S, Greenberg HB. Gene expression pattern in Caco-2 cells following rotavirus infection. J Virol. 2002; 76:4467-82.
Delmas O, Gardet A, Chwetzoff S, Breton M, Cohen J, Colard O, Sapin C, Trugnan G. Minireview: Different ways to reach the top of a cell. Analysis of rotavirus assembly and targeting in human intestinal cells reveals an original raft-dependent, Golgi-independent apical targeting pathway. Virology. 2004; 327:157- 161.
Estes M. Rotaviruses and their replication. In: Knipe DM and Howley PM (ed) Fields virology, 4th ed. Lippincott- Raven publishers. Philadelphia, 2001; pp 1747-1785.
Fleming FE, Graham KL, Taniguchi K, Takada Y, Coulson BS. Rotavirus-neutralizing antibodies inhibit virus binding to integrins alpha 2 beta 1 and alpha 4 beta 1. Arch Virol 2007; 152:1087-101.
Geyra A, Uni Z, Gal-Garber O, Guy D, Sklan D. Starving Affects CDX Gene Expression during Small Intestinal Development in the Chick. J. Nutr. 2002; 132:911-917.
Calderon MN, Acosta O, Guerrero CA, Guzman F. Protein disulfide isomerase activity is involved in rotavirus entry to MA104 cells. Proceedings of the XIV International Congress of Virology, Istanbul. 2008; pp. 163-164.
Fang J, Kairbaan H, Bryon D, Hynes R, White G, Wilcox D. Therapeutic expression of the platelet-specific integrin, aII β3, in a murine model for Glanzmann thrombasthenia. Blood. 2005; 106: 2671-2679.
Essex DW, Li M. Protein disulphide isomerase mediates platelet aggregation and secretion. Br J Haematol. 1999; 104: 448-454.
Beaver B, Reed W, Leary S, Mckiernan B, Bain F, et al. Report of the AVMA Panel on Euthanasia. J Am Vet Med Assoc. 2001; 218: 669-696.
Santana Y. Participación de la proteína de choque térmico HSC70, la integrina av β3 y la proteína disulfuro isomerasa en el proceso de entrada del rotavirus homólogo Ecwt y heterólogos RRV y Wa en cultivos primarios de células intestinales de ratón lactante. 2009. Tesis de Maestría, Universidad Nacional de Colombia.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2010 Revista de la Facultad de Medicina

Esta obra está bajo una licencia Creative Commons Reconocimiento 3.0 Unported.
-