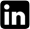La proteína PthB de Xanthomonas axonopodis pv. manihotis es autoactiva en ensayos de doble hibrido
LA PROTEÍNA PTHB DE Xanthomonas axonopodis pv. Manihotis ES AUTOACTIVA EN ENSAYOS DE DOBLE HÍBRIDO
The PthB Protein from Xanthomonas axonopodis pv. Manihotis is an Autoactive in Yeast Two-Hybrid Assays
JULIANA GIL1, Bióloga; LAURA BOHÓRQUEZ2, Bióloga; LUISA B. CASTIBLANCO3, M.Sc.; ADRIANA BERNAL3, M.Sc.; CAMILO LÓPEZ4, Ph. D. 1 Universidad de California-Davis, California, Estados Unidos. 2 Corpogen, Bogotá, Colombia. 3 Departamento de Biología, Universidad de los Andes, Bogotá, Colombia. 4 Departamento de Biología (edificio 421), Universidad Nacional de Colombia, Bogotá, Colombia. celopezc@unal.edu.co
Presentado 2 de febrero de 2010, aceptado 16 de febrero de 2011, correcciones 17 de febrero de 2011.
RESUMEN
La bacteriosis vascular de yuca producida por la bacteria Xanthomonas axonopodis pv. manihotis (Xam) es una enfermedad limitante para la producción de yuca. Dentro de los primeros factores de patogenicidad identificados en esta bacteria se encuentra el gen PthB. La proteína PthB pertenece a la familia de efectores PthA/AvrBs3, que se caracterizan por presentar dominios NLS (Nuclear Localization Signal) y un dominio AAD (Acidic Activation Domain), lo cual sugiere que estas proteínas actúan como factores de transcripción. La identificación de las proteínas de yuca que interactúan con PthB permitiría dar luces sobre la función de esta proteína en la patogenicidad de esta bacteria. En este trabajo se clonó PthB en una fusión traduccional con el BD (Binding Domain) del factor de transcripción GAL4. Después de transformar este constructo en una cepa de levadura, se observó autoactivación de los genes reporteros, incluso a concentraciones altas de 3-AT. La eliminación del primer, segundo o de los dos NLS y del AAD no eliminaron la capacidad de autoactivación de los genes reporteros mediada por PthB. Estos resultados indican la imposibilidad de su utilización en un tamizaje de una librería de ADNc de yuca para identificar las proteínas que interactúan con PthB.
Palabras clave: yuca, bacteriosis vascular, doble hibrido, PthB.
ABSTRACT
Cassava bacterial blight disease is caused by the gram-negative bacteria Xanthomonas axonopodis pv. manihotis (Xam), and constitutes one of the most important constraints for cassava production. One of the first determinants of pathogenicity identified in this bacterium is the PthB gene. The PthB protein belongs to the PthA/AvrBs3 family, characterized by the presence of Nuclear Localization Signal (NLS) and Acidic Activation (AAD) domains, suggesting that these proteins are transcription factors. The identification of cassava proteins interacting with PthB could give insights about the function of this protein in the pathogenicity of this bacterium. In this work we cloned PthB in the yeast two hybrid expression vector pLAW10, generating a fusion protein with the Binding Domain (BD) of the transcription factor GAL4. In this work, PthB was cloned in a translational fusion with Gal4-BD (DNA Binding Domain). After transforming this construct into a yeast strain, autoactivation of the reporter genes was observed, even at the highest concentrations of 3-AT. The deletion of the first, second or both NLS and the AAD did not eliminate the ability of autoactivation of PthB. These results show the impossibility of using PthB to screen a cassava cDNA library to identify the proteins interacting with PthB.
Key words: cassava, cassava bacterial blight, yeast two-hybrid, PthB.
INTRODUCCIÓN
La bacteria gram negativa Xanthomonas axonopodis pv. manihotis (Xam), es el agente causal de la bacteriosis vascular de la yuca (Manihot esculenta) y puede ocasionar pérdidas de hasta el 100% de su producción (López et al., 2006). Esta bacteria se encuentra distribuida en el área tropical de todo el mundo donde se cultiva yuca, comprometiendo el principal suministro de calorías para más de un billón de personas (FAOSTAT, 2008). La yuca es uno de los cultivos principales de seguridad alimentaria a nivel mundial. Adicionalmente, sus subproductos tienen una gran gama de aplicaciones industriales, incluyendo la producción de bioetanol a partir del almidón (El-Sharkawy, 2004).
La bacteriosis vascular se caracteriza por tener un primer ciclo de infección epifítico (Restrepo et al., 2004) en donde la bacteria se reproduce para después penetrar, durante la época lluviosa a través de estomas y heridas, al tejido vascular. Los síntomas principales que se observan son manchas angulares, marchitamiento, producción de exudados en los tallos y finalmente muerte de la planta (Restrepo et al., 1999).
Para lograr una colonización exitosa dentro de las células vegetales, las diferentes especies de bacterias fitopatógenas han desarrollado proteínas efectoras que son inyectadas al citoplasma de las células vegetales a través del sistema de secreción tipo tres (T3SS, del inglés Type Three Secretion System; Jones y Dangl, 2006). Estas proteínas efectoras, a través de diferentes mecanismos, suprimen la primera rama de inmunidad vegetal conocida como PTI (del inglés PAMP Trigger Immunity; Zipfel, 2008). Sin embargo, las plantas en su proceso co-evolutivo con patógenos, desarrollaron proteínas de resistencia capaces de reconocer ya sea de manera directa o indirecta proteínas efectoras o el efecto que ellas tienen sobre otras proteínas de la planta, tales como la fosforilación o degradación . En este caso, dicho reconocimiento desencadena una respuesta hipersensible que consiste en muerte celular programada que detiene la multiplicación del patógeno y resulta en fenotipos resistentes en las plantas (Jones y Dangl, 2006).
La mayoría de efectores no muestran características conservadas entre sí y presentan una gran diversidad no solo a nivel de secuencia sino también a nivel de funciones bioquímicas (Bent y Mackey, 2007). Una excepción la constituyen los efectores de la familia PthA/AvrsBs3, los cuales se caracterizan por poseer tres dominios. En la región central de la proteína se encuentra el primer dominio compuesto por una serie de 34 aminoácidos repetidos varias veces y en donde el número y orden de repeticiones determina la especificidad de la cepa a una variedad particular de hospedero (Buttner y Bonas, 2003). En la región C-terminal se encuentra una señal de localización nuclear o NLS (del inglés Nuclear Localization Signal), la cual esta implicada en la translocación de la proteína al núcleo de las células vegetales (Marois et al., 2002; Van den Ackerveken et al., 1996). Así mismo, en esta región se encuentra un dominio acídico de activación de la transcripción (AAD, del inglés Acidic Activation Domain; Römer et al., 2007; Kay et al., 2007). Adicionalmente, en el extremo N-terminal parece encontrarse una señal putativa de translocación mediante el T3SS, pero cuya estructura no se ha definido (Schornack et al., 2006).
Estudios recientes han mostrado que la proteína AvrBs3 de Xanthomonas campestris pv. vesicatoria, es capaz de interactuar con proteínas de la familia α-importina en células de pimentón (Szurek et al., 2001). De igual manera se ha determinado que AvrBs3 se une directamente al ADN, en la región promotora de genes blanco para inducir su expresión (Kay et al., 2007). En el caso de una interacción compatible (susceptibilidad), la inducción de genes mediada por AvrBs3 produce hipertrofia celular (Kay et al., 2007). En el caso de una interacción incompatible (resistencia), la proteína AvrBs3 se une al promotor del gen de resistencia Bs3, lo que produce una respuesta de defensa que detiene la colonización del patógeno (Römer et al., 2007). Recientemente se ha determinado que la secuencia de los 34 aminoácidos y el número de estas unidades repetitivas representan un código que permite predecir la secuencia de ADN a la cual el tipo de efectores AvrBs3 es capaz de unirse (Boch et al., 2009).
En Xam se ha logrado identificar una proteína perteneciente a la familia AvrBs3 y se ha denominado PthB (Restrepo et al., 1999; Restrepo et al., 2004; Castilblanco et al., comunicación personal). PthB parece ser crucial para la patogenicidad de Xam. Un fragmento de ADN correspondiente a la secuencia codificante para la proteína PthB, ha sido empleado como sonda de hibridación para determinar la diversidad y estructura de las poblaciones de Xam (Restrepo et al., 1999). A pesar de su importancia, la función y actividad fisiológica y molecular de PthB como proteína determinante en la patogenicidad de Xam dentro de las células de yuca aún no se conoce. Con miras a ganar una mayor información sobre la función de PthB es importante identificar las proteínas con las cuales PthB interactúa dentro de las células vegetales. La estrategia de doble híbrido se constituye en una de las mejores alternativas para identificar las proteínas de yuca que interactúan con PthB. Sin embargo, para realizar este tipo de estudios es necesario primero determinar si la proteína PthB no presenta autoactivación intrínseca en estudios de doble híbrido.
MATERIALES Y MÉTODOS
CLONACIÓN DE PTHB EN SISTEMA GATEWAY
La clonación de PthB en el sistema Gateway® (Invitrogen, Carlsbad, CA) se llevó a cabo en el vector pENTR/D-TOPO. Para clonar los productos de PCR en este vector se incluyeron cuatro nucleótidos en el cebador sentido (CACC). Los cebadores para la amplificación de PthB se diseñaron a partir de la secuencia reportada en GenBank (N° AF 012325), mediante el programa Primer3 (Rozen y Skaletsky, 2000). La secuencia del cebador sentido N-terminal, NF fue 5’-CACCATGGATCCCATTCGTCCG-3’. La secuencia del cebador antisentido C-terminal, CR fue -ACTAGTTCACTGAGGAAATAGCTCCAT-3’. Para llevar a cabo la amplificación de PthB se empleó como ADN molde el plásmido F3 (pF3; provisto por Valerie Verdier del Institut de Recherche pour le Developpement, IRD, Francia), el cual contiene un fragmento de 5,4 kb, en el que se encuentra PthB. Las reacciones de amplificación se realizaron en un volumen final de 50 µL, las cuales contenían 0,4 µM de cada cebador, 0,4 mM de dNTPs, 2 mM de MgSO4, 1 unidad de enzima Platinum® TaqHighFidelity DNA polimerasa (Invitrogen, Carlsbad, CA), 1X de buffer de PCR, dimetilsulfóxido (DMSO) al 3% de concentración final. La cantidad utilizada de pF3 fue de aproximadamente 150 ng. Las reacciones de PCR se llevaron a cabo en un termociclador iCycler (BioRad®) y se utilizó el siguiente protocolo de amplificación: 1) desnaturalización inicial a 94°C por dos minutos, 2) 35 ciclos de a) desnaturalización a 94 °C por 30 segundos, b) alineamiento de los cebadores a 52 °C por 30 segundos, c) extensión a 68 °C por 6 minutos. 10 µL del producto de amplificación se corrieron en un gel de agarosa, se realizó la limpieza de los restantes 40 µL de cada producto mediante kit de purificación de ADN (Promega, Madison, WI). El producto de PCR de la amplificación de PthB se clonó en el vector pENTR/D-TOPO. La reacción de clonación se realizó con 2 µL de producto de PCR, 1 µL de solución salina, 1 µL del vector. Se transformaron bacterias E. coli quimiocompetentes OneShot Top 10® (Invitrogen, Carlsbad, CA), siguiendo el protocolo de choque térmico (Sambrook et al., 1989).
Para confirmar la presencia del inserto de interés se tomaron algunas colonias en los clones obtenidos y se realizó un PCR de colonia. A partir de los clones positivos se realizó extracción de ADN plasmídico mediante lisis alcalina (Sambrook et al., 1989). El inserto fue parcialmente secuenciado y transferido por reacción LR y posterior BP, según las instrucciones del fabricante al vector de entrada pDONR207 (Invitrogen, Carlsbad, CA). Los clones fueron digeridos con BamHI, al igual que el plásmido pF3, siguiendo las instrucciones del fabricante (New England biolabs). Posterior a la digestión se realizó ligación empleando el fragmento de 3 kb liberado de pF3 con el producto de digestión del clon proveniente de PCR en pDONR207, previa defosforilación del clon proveniente del producto de PCR en pDONR207 y pF3 previa defosforilación empleando la CIP (New England biolabs). La reacción de ligación fue transformada por choque térmico en bacterias E. coli quimiocompetentes OneShot Top 10® (Invitrogen, Carlsbad, CA), siguiendo el protocolo de choque térmico (Sambrook et al., 1989). Los clones obtenidos se seleccionaron, se amplificaron y se realizó la secuenciación completa de este nuevo clon en la Universidad de California-Davis.
CONSTRUCCIÓN DE LA CARNADA DEL SISTEMA DOBLE HÍBRIDO CON PTHB
A partir del clon pDONR207:PthB se realizó una reacción LR (Invitrogen, Carlsbad, CA) a los vectores de expresión pLAW10 y pLAW11, siguiendo la metodología descrita anteriormente. Las reacciones LR fueron confirmadas por PCR de colonia y secuenciación parcial del inserto empleando las condiciones descritas en la sección anterior. Los vectores pLAW10 y pLAW11 son vectores de doble híbrido derivados de pGBkT7 y pGADT7 respectivamente (Clontech, 2009). En estos vectores el sitio de multiclonaje ha sido remplazado por el cassette de recombinación Gateway con los sitios de reconocimiento para las enzimas LR. pGBkT7 (Clontech, 2009) y por ende pLAW10 fueron diseñados de tal manera que permiten la inserción del fragmento de interés como una fusión N-terminal al dominio de unión al ADN, BD (del inglés Binding Domain) proveniente del factor de transcripción GAL4. Este vector presenta además resistencia a kanamicina (50 µg/mL), un gen de la ruta biosintética del triptófano (TRP), como marcador en levadura (Trp-) y un tag c-Myc. pLAW11 fue construido a partir de los vectores pGAD con epítope HA (Clontech, 2009). Los vectores pGAD y pLAW11 permiten la inserción del gen de interés en fusión N-terminal al dominio de activación de GAL4, AD (del inglés Activation Domain), presenta además resistencia a ampicilina (100 µg/mL) y un gen (LEU2) de la ruta biosintética de la leucina como marcador en levaduras (Leu –).
TRANSFORMACIÓN A LEVADURA
En este estudio se empleó la cepa Y187 (cortesía del Dr R. Michelmore, UC Davis) para introducir la proteína carnada, en nuestro caso PthB, que contiene el BD. El vector pLAW10:PthB se transformó en S. cerevisiae Y187 (MATα, ura3-52, his3-200, ade2-101, trp1-901,leu2-3, 112, gal4Δ, met–, gal80Δ, URA3::GAL1UAS-GAL1TATA-lacZ) según el protocolo descrito en el Manual de Clontech. La otra cepa de levadura empleada fue AH109 en la que se cotransformaron (por apareamiento) la construcción pLAW10:PthB y pLAW11 vacío. AH109 es una cepa MATa con mutaciones por deleción en los genes, trp1-901, leu23, 112, ura3-52, his3-200, HIS3, ADE2, lacZ, trp1 y leu2. Para el apareamiento se emplearon 0,4 µg de ADN plasmídico, 50 µg de esperma de salmón denaturado (Invitrogen, Carlsbad, CA, USA) y 50 µL de células competentes de Y187 (conteniendo previamente la construcción pLAW10:PthB) recién preparadas. A cada evento de transformación se le adicionó 0,5 mL de solución PEG/LiAc se incubó 30 minutos a 30 °C con continua agitación, se le añadieron 20 µL de DMSO y se incubó 15 minutos a 42 °C con agitación continua. Las levaduras se resuspendieron en 1 mL de H2O deionizada estéril, se sembraron en medio sólido y se incubaron tres días a 30 °C. En la selección de las células transformadas se empleó medio SD sin leucina y sin triptófano (SD-W-L). La ausencia de leucina en el medio se empleó como selección para la presencia de pLAW11 que contiene el AD del factor de transcripción mientras que la ausencia de triptófano en el medio servirá para seleccionar la presencia de pLAW10 y de las construcciones hechas a partir de éste. Las levaduras resultantes se sembraron en medio SD/-Trp-Leu-His a diferentes concentraciones de 3AT (3-Amino-1,2,4-triazole): 0 mM, 5 mM, 10 mM, 20 mM, 30 mM, 50 mM, 75 mM, 100 mM y se incubaron por ocho días a 30 °C.
Las deleciones de los dominios eucariota presentes en la región C-terminal de PthB se produjeron por mutagénesis sitio dirigida empleando la estrategia de Overlap Extension PCR (OE-PCR; Higuchi et al., 1988). Las reacciones de PCR se llevaron a cabo empleando Taq polimerasa de alta fidelidad (Platinum Pfx DNA polymerase, Invitrogen). El cebador del extremo 3’ poseía un sitio de restricción para la enzima AscI. La región N-terminal fue amplificada a partir de pF3 empleando cebadores de la región codificante y adicionando la secuencia CACC y el sitio de corte XhoI en el cebador del extremo 5’ para la clonación en pENTR/D-TOPO (Invitrogen), siguiendo las condiciones descritas anteriormente. Las diferentes versiones de la región C-terminal fueron clonadas en pGEM T-easy (Promega), siguiendo las instrucciones del fabricante. La región central que contiene las unidades repetitivas fue obtenida a partir de pF3 por digestión con SphI y XhoI y ligada a la región N-terminal digerida con las mismas enzimas. Las versiones C-terminal fueron extraídas de pGEM T-easy con XhoI y AscI y ligadas con las región que contiene las repeticiones y el domino N-terminal que se encontraba en pENTR/D-TOPO, ensamblando de esta manera las versiones completas del gen PthB. Las construcciones resultantes obtenidas en pENTR/D-TOPO fueron transferidas mediante una reacción LR a los vectores de doble híbrido para posteriormente transformar levaduras como se describió anteriormente. Las levaduras resultantes se sembraron igualmente en medio SD/-Trp-Leu-His a diferentes concentraciones de 3AT: 0 mM, 5 mM, 10 mM, 20 mM, 30 mM, 50 mM, 75 mM, 100 mM y se incubaron 8 días a 30 °C.
RESULTADOS Y DISCUSIÓN
AMPLIFICACIÓN DE PTHB Y CLONACIÓN EN EL SISTEMA GATEWAY
Las características de PthB, tales como su gran tamaño (3,6 kb), la presencia de la región central compuesta de varias unidades repetitivas (Figura 1A) y su relativo alto contenido de GC, dificultan su amplificación mediante estrategias de PCR convencionales. Para lograr la amplificación de PthB a partir de pF3 se debieron evaluar diferentes concentraciones de cebadores, emplear tiempos de extensión largos y sobre todo incluir DMSO en la mezcla de reacción. Finalmente se logró obtener amplificación correcta de un fragmento de aproximadamente 3,6 kb (Figura 1B), que permitió posteriormente su clonación en el vector pENTR/D-TOPO. Posterior a la transformación se obtuvieron > 2 x 102 colonias. Se escogieron 10 clones, que crecieron en el medio selectivo. Para confirmar la presencia del inserto, inicialmente se realizó la amplificación de la región N-terminal mediante un PCR directo sobre colonias, con la cual se comprobó que todos los clones seleccionados eran positivos. La segunda amplificación confirmatoria se realizó sobre la región C-terminal empleando el ADN plasmídico, pero fue positiva solamente para dos clones. La secuencia obtenida para uno de los clones fue de muy baja calidad, por lo tanto no se tuvo en cuenta para análisis posterior. Mientras que las secuencias para el clon restante (pENTR/D-TOPO:PthB2) fueron de buena calidad y permitieron determinar que este clon contiene una versión pero que contiene solamente 8,5 repeticiones con respecto a las 13,5 repeticiones en la secuencia de PthB reportada en GenBank. La pérdida en algunas de las unidades repetitivas puede deberse a problemas durante la amplificación de PthB debido a la utilización de DMSO que puede estar causando mutaciones y a que la actividad de corrección de la enzima Taq High Fidelity DNA polimerasa no es suficiente para la amplificación y corrección de la larga secuencia de la región repetitiva (≈1275pb) de PthB. A pesar de que el clon pENTR/D-TOPO:PthB2 no contiene la versión completa de PthB, sí está en un vector compatible con el sistema Gateway, a partir del cual se puede realizar fácilmente la introducción de PthB a otros vectores Gateway. La versión PthB2 fue transferida mediante una reacción LR al vector pDONR207, obteniendo la construcción pDONR207:PthB2.
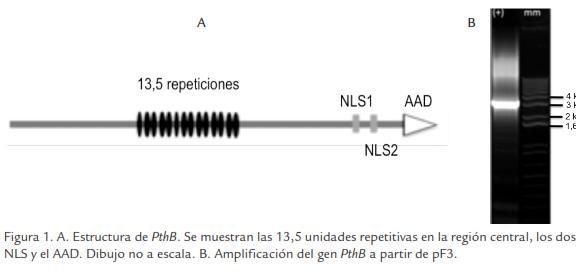
Para contar con una versión de PthB completa en el sistema Gateway se digirió pDON R207:PthB2 y pF3 con BamHI, lo cual permite liberar solamente la región central que contiene las unidades repetitivas. Las bandas producto de la digestión correspondiente a pDONR207 conteniendo la región C-terminal y N-terminal de PthB (tamaño aproximado de 3,6 kb: 3 kb del vector y aproximadamente 600 pb de los extremos N y C-terminal) y la región de las unidades repetitivas de pF3 incluyendo la región complementaria de los extremos N y C-terminal (fragmento de aproximadamente 3 kb; Figura 2) fueron eluídas del gel y empleadas en una reacción de ligación para reconstruir de esta forma el gen PthB completo en el vector pDONR207:PthB. Finalmente se obtuvieron clones que fueron secuenciados, obteniéndose uno de ellos en el cual se pudo demostrar que el gen estaba completo, con la región N-terminal, las unidades repetitivas y la región C-terminal con los motivos NLS y AAD. Este clon fue denominado pDONR207:PthB.
ENSAYOS DE AUTOACTIVACIÓN EN EL SISTEMA DOBLE HÍBRIDO
El gen PthB fue transferido de pDONR207:PthB al vector de expresión pLAW10 mediante una reacción LR. El vector pLAW10 posee el BD del factor de transcripción GAL4. De esta manera se produce una proteína de fusión entre PthB y el BD del factor de transcripción GAL4. La construcción pLAW10:PthB fue transformada a la cepa de levadura AH109 la cual ha sido transformada previamente con el vector pLAW11 vacío. El vector pLAW11 posee la secuencia codificante para el AD del factor de transcripción GAL4. Las levaduras obtenidas crecieron en medio SD sin triptófano y leucina, indicando que las levaduras incorporaron correctamente los plásmidos pLAW10 y pLAW11, los cuales poseen los genes capaces de sintetizar estos aminoácidos (Figura 3A). Levaduras sin contener los vectores fueron incapaces de crecer en el medio (Figura 3B).

En un tamizaje para la búsqueda de interactores con PthB se introduce en las levaduras el vector pLAW11, conteniendo insertos provenientes de una librería de ADNc de yuca. En el caso de que exista una interacción se restablecerá un factor de transcripción activo que permitirá la transcripción del gen reportero, en cuyo caso sería el gen que codifica para una proteína implicada en la biosíntesis de histidina, el cual está mutado en el genoma de la levadura AH109. Sin embargo, esta es una mutación débil y en algunos casos las levaduras son capaces de crecer en un medio sin histidina, obteniéndose en consecuencia un número alto de falsos positivos. Para evitar este inconveniente se suele emplear 3AT en el medio, el cual es un inhibidor competitivo de la ruta de biosíntesis de histidina. Las levaduras AH109 conteniendo pLAW10:PthB + pLAW11:vector vacío, se pusieron a crecer en medio SD sin triptófano, sin leucina y sin histidina, con crecientes concentraciones de 3AT. Como se observa en la Figura 4, las levaduras crecieron incluso a altas concentraciones de 3AT (75 y 100 mM; Figura 4). Estos resultados indican que PthB es autoactivo en ensayos de doble hibrido de levadura y que posiblemente por las características de esta proteína (motivos NLS y AAD), se produce activación constitutiva del gen reportero para la biosíntesis de histidina. Existen reportes en que se ha establecido que la proteína fusionada al BD cuando posee determinadas características, tales como presencia de dominios de activación y alta concentración de residuos ácidos, produce auto activación (Szurek et al., 2001; Sobhanifar, 2003). Esto significa que en los experimentos de doble híbrido, la proteína posee la capacidad de reclutar por sí sola, sin un dominio de activación proveniente de la proteína fusión, la maquinaria de transcripción lo que permite la expresión del gen reportero.

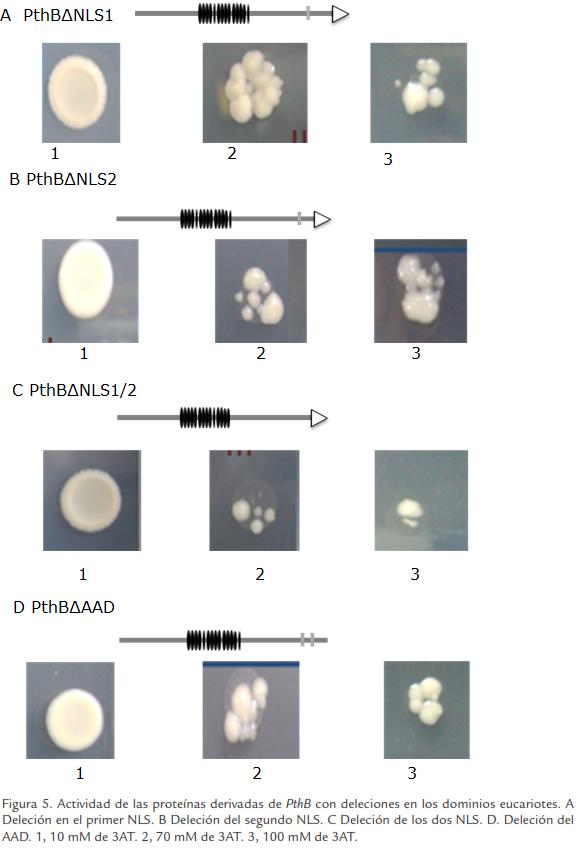
En vista de la naturaleza autoactiva de PthB se decidió realizar una serie de deleciones en el gen PthB para determinar la pérdida de la capacidad de autoactivación y de esta manera proceder con el tamizaje de la librería de ADNc. Para probar si los dominios eucarióticos en el extremo C-terminal de PthB juegan un papel en la autoactivación, se realizaron cuatro versiones diferentes de la proteína: sin el primer motivo NLS (PthBΔNLS1), sin el segundo (PthBΔNLS2), sin ninguno de los dos (PthBΔNLS1/2) y sin el dominio de activación transcripcional (PthBΔAAD; Figura 5). Estas versiones se clonaron en pLAW10 y se introdujeron a la levadura AH109. Como se observó en los primeros experimentos, las levaduras fueron capaces de crecer en medio sin histidina y a altas concentraciones de 3AT (Figura 5), indicando que la capacidad de autoactivación de PthB no depende de los dominios eucarióticos presentes en el extremo C-terminal.
Los trabajos previos de doble híbrido llevados a cabo empleando AvrBs3 permitieron demostrar que ésta proteína también presenta autoactivación y que la región responsable corresponde a una región que cubre aproximadamente los primeros 100 aminoácidos de la proteína (Szureket al., 2001). Los miembros de la familia AvrBs3 presentan un grado alto de conservación a nivel de secuencia, diferenciándose principalmente en el número de unidades repetitivas en la región central (Buttner y Bonas, 2003). Este alto grado de conservación sugiere que en PthB, al igual que en AvrBs3, sea la región del N-terminal la responsable de la autoactivación.
Las características propias de los miembros de la familia AvrBs3, tales como presencia de dominios NLS y AAD, sugieren que este tipo de proteínas se comportan como factores transcripcionales. Recientemente se ha confirmado esta hipótesis a través de la demostración que la proteína AvrBs3 se une a secuencias de ADN específicas en el promotor de genes blanco para permitir la represión o expresión de genes para el beneficio de la bacteria (Kay et al., 2007). De este modo es posible que PthB también juegue un papel similar a AvrBs3. En consecuencia, el desafío prioritario actual es identificar los promotores de los genes blanco a los cuales PthB se une para regular la expresión génica. El empleo de estrategias de genómica funcional como los microrarreglos permitirán identificar qué genes son expresados o reprimidos en plantas de yuca, cuando se compara el perfil de expresión entre plantas inoculadas con una bacteria con y sin PthB. De igual manera, con la liberación reciente del genoma completo de yuca, mediante herramientas de bioinformática es posible predecir promotores blanco de PthB. Alternativamente se puede emplear el sistema de un híbrido para determinar las secuencias de ADN con las cuales PthB puede interactuar.
AGRADECIMIENTOS
Este proyecto fue financiado parcialmente con el apoyo de Fundación para la Promoción de la Investigación y la Tecnología (Banco de la República) proyecto 2.049, del IFS (InternationalFoundation for Science) y DIB (División de Investigación de la Universidad Nacional de Colombia-sede Bogotá), ficha quipú 20101007581. Gracias especiales a Luis Williams y Richard Michelmore de la Universidad de California (Davis) por el entrenamiento en doble híbrido y por poner a nuestra disposición las instalaciones de su laboratorio.
BIBLIOGRAFÍA
BENT AF, MACKEY D. Elicitors, Effectors, and R Genes: The New Paradigm and a Lifetime Supply of Questions. Annu Rev Physiol. 2007;45:399-436.
BOCH J, SCHOLZE H, SCHORNACK S, LANDGRAF A, HAHN S, KAY S, LAHAYE T, NICKSTADT A, BONAS U. Breaking the Code of DNA Binding Specificity of TAL-Type III Effectors. Science. 2009;326:1509-1512.
BUTTNER D, BONAS U. Common infection strategies of plant and animal pathogenic bacteria. Curr Opin Plant Biol. 2003;6:312-319.
CLONTECH. http://www.clontech.com/CO/Home?sitex=10028:22372:US.
EL-SHARKAWY MA. Cassava biology and physiology. Plant Mol Biol. 2004;56:481-501.
HIGUCHI R, KRUMMEL B, SAIKI R. A general method of in vitro preparation and specific mutagenesis of DNA fragments: study of protein and DNA interactions. Nucl Acids Res. 1988;16:7351-7367.
FAOSTAT. FAO database. Food and Agriculture Organization of theUnited Nations. 2008. Rome, Italy. http://faostat.fao.org/site/567/default.aspx.
JONES JD, DANGL JL. The plant immune system. Nature. 2006;444:323-329.
KAY S, HAHN S, MAROIS E, HAUSE G, BONAS U. A Bacterial Effector Acts as a Plant Transcription Factor and Induces a Cell Size Regulator. Science. 2007;318:648-651.
LÓPEZ C, RESTREPO S, VERDIER V. Limitaciones en la Bacteriosis Vascular de la Yuca: Nuevos Avances. Act biol Colom. 2006;11:21-45.
MAROIS E, VAN DEN ACKERVEKEN G, BONAS U. The xanthomonas type III effector protein AvrBs3 modulates plant gene expression and induces cell hypertrophy in the susceptible host. Mol Plant Microbe Interact. 2002;15:637-646.
RÖMER P, HAHN S, JORDAN T, STRAUSS T, BONAS U, LAHAYE T. Plant pathogen recognition mediated by promoter activation of the pepper Bs3 resistance gene. Science. 2007;318:645-648.
RESTREPO S, VALLE T, VERDIER V. Assessing genetic variability among Brazilian strains of Xanthomonas axonopodis pv. manihotisthrough RFLP and AFLP analyses. Can J Microbiol 1999;45:754-763.
RESTREPO S, VELEZ CM, VERDIER V. Genetic structure and population dynamics of Xanthomonas axonopodis pv. manihotis in Colombia from 1995 to 1999. Appl Environ Microbiol. 2004;70:255-261.
ROZEN S, SKALETSKY HJ. Primer3 on the WWW for general users and for biologist programmers. In: Krawetz S, Misener S (eds) Bioinformatics Methods and Protocols: Methods in Molecular Biology. Humana Press, Totowa, NJ; 2000. p. 365-386.
SAMBROOK J, FRITSH EF, MANIATIS T. Molecular cloning: a laboratory manual. New York: Cold Spring Harbor Laboratory Press; 1989.
SCHORNACK S, MEYER A, RÖMER, P, JORDAN T, LAHAYE T. Gene-for-gene-mediated recognition of nuclear-targeted AvrBs3-like bacterial effector proteins. J Plant Physiol. 2006;163:256-272.
SOBHANIFAR S. Yeast Two Hybrid Assay: A Fishing Tale. BioTeach Journal. 2003;1:81-87.
SZUREK B, MAROIS E, BONAS U, VAN DEN ACKERVEKEN G. Eukaryotic features of the Xanthomonas type III effector AvrBs3: protein domains involved in transcriptional activation and the interaction with nuclear import receptors from pepper. Plant J. 2001;26:523-534.
VAN DEN ACKERVEKEN G, MAROIS E, BONAS U. Recognition of the bacterial avirulence protein AvrBs3 occurs inside the host plant cell. Cell. 1996;87:1307-1316.
ZIPFEL C. Pattern-recognition receptors in plant innate immunity. Curr Opin Immunol. 2008;20:10-16.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2011 Acta Biológica Colombiana

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
1. La aceptación de manuscritos por parte de la revista implicará, además de su edición electrónica de acceso abierto bajo licencia Attribution-NonCommercial-ShareAlike 4.0 (CC BY NC SA), la inclusión y difusión del texto completo a través del repositorio institucional de la Universidad Nacional de Colombia y en todas aquellas bases de datos especializadas que el editor considere adecuadas para su indización con miras a incrementar la visibilidad de la revista.
2. Acta Biológica Colombiana permite a los autores archivar, descargar y compartir, la versión final publicada, así como las versiones pre-print y post-print incluyendo un encabezado con la referencia bibliográfica del articulo publicado.
3. Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
4. Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos institucionales, en su página web o en redes sociales cientificas como Academia, Researchgate; Mendelay) lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).