Necesidad de un Centro Nacional de Bioinformática y Biología Computacional para Colombia
Necesidad de un Centro Nacional de Bioinformática y Biología Computacional para Colombia
The need for a Colombian bioinformatics and computational biology centre
Lyda Raquel Castro1 , Ricardo Gutiérrez2 , Marco Cristancho3
1Grupo de investigación en evolución y sistemática molecular, Intropic, Universidad del Magdalena, Santa Marta, Colombia. 2División de Ciencias Básicas, Grupo de investigación en química y biología, Universidad del Norte, Atlántico, Colombia. 3Centro Nacional de Investigación de Café (Cenicafe), Caldas, Colombia.
Recibido: febrero 26 de 2010 Aprobado: junio 17 de 2010
Resumen
Los principales y más revolucionarios avances de la biología en este siglo se han derivado de la información proveniente de genomas completos de diferentes organismos. Los descubrimientos que se derivan de la genómica están generando un nuevo paradigma en la biología, sustituyendo la era de la biología centrada en los genes por aquella centrada en los genomas. Este nuevo concepto es base para desarrollos de gran potencial e impacto social en diferentes áreas como la medicina, la agricultura y la industria. El éxito en el desarrollo de métodos de última generación para la secuenciación de genomas, la proteómica y todas las omicas, ha contribuido al surgimiento de nuevas posibilidades para el análisis de la enorme cantidad de datos que se están generando mediante el uso de herramientas computacionales, dando origen a una nueva rama de estudio conocida como bioinformática o biología computacional.
Este trabajo hace una revisión general del desarrollo de la bioinformática y la biología computacional en Colombia. Inicialmente, a modo de comparación, describimos el desarrollo de esta ciencia en otros países latinoamericanos que son reconocidos en el área. Finalmente, se discuten los principales aspectos que van a jugar un papel importante en el futuro de esta ciencia en nuestro país, y que además justifican la necesidad de crear un centro nacional de bioinformática y biología computacional.
Abstract
The main, most revolutionary advances in biology during this century have arisen from information being provided from revealing different organisms complete genomes. The discoveries deriving from genomics are leading to a new paradigm in biology, the era of gene-centred biology being substituted for that centred on genomes. Such new concepts areas such as medicine, agriculture and industry. The successful development of latestgeneration methods for genome sequencing, proteomics and all the other -omics has contributed towards an enormous amount of genetic data being produced that needs to be analyzed using computational tools. This has led to the development of a new area called bioinformatics or computational biology.
The present work gives a general overview of the development of bioinformatics and computational biology in Colombia. The justification for the establishment of a National bioinformatics and computational biology centre is discussed, as well as some aspects that will be crucial for developing this science in Colombia.
Breve definición e historia
La reciente revolución de la genómica y la bioinformática ha invadido el mundo entero de una manera sorprendentemente rápida. En muchas conversaciones y escritos, inclusive de personas no científicas, el dilema del genoma humano incluyendo el análisis de la variación genética, el acceso a la información genética y el alcance que implica conocer esta secuencia, es tema de debate (Ouzounis y Valencia, 2003).
En las dos últimas décadas, el almacenamiento de datos biológicos en bases de datos públicas se ha vuelto extremadamente común, y estas bases de datos han crecido a una velocidad enorme (Gibas y Jamebeck, 2001). Para ser más explícitos respecto a la tasa de crecimiento de la información, en agosto del 2000, la base de datos Gen- Bank contenía 8,214.000 secuencias (Benson et al., 2000), mientras que para agosto del 2009 este número había aumentado a 108431.692 (Benson et al., 2010). Once millones de secuencias fueron agregadas en el 2009. Desde su comienzo, GenBank ha crecido exponencialmente y continúa haciéndolo, doblando su tamaño aproximadamente cada 35 meses. Los genomas completos representan un área de la base de datos con alta tasa de crecimiento. En este momento GenBank contiene más de 1000 genomas completos de bacteria y archae, de los cuales 30% fueron depositados durante el 2009. El número de genomas eucariotas asciende a 380 WGS de ensamblajes disponibles (Benson et al., 2010).
Como resultado de esta enorme cantidad de información, los computadores se han vuelto indispensables para analizar los datos biológicos. La bioinformática se ha definido, entonces, como la aplicación de las técnicas computacionales para entender, organizar y analizar la información asociada a las macromoléculas (Luscombe et al., 2001).
La biología computacional, en cambio, abarca más allá de la bioinformática, podríamos definirla como todos los aspectos de la biología computacional aplicados a las diferentes áreas de la biología en todas sus escalas, desde moléculas y células hasta poblaciones y ecosistemas. La investigación en biología computacional comprende entonces cualquier cosa, desde abstracción de las propiedades de un sistema biológico en un modelo físico o matemático, hasta la implementación de nuevos algoritmos para el análisis de datos o el desarrollo de bases de datos con herramientas web para acceso público (Young, 2003). En comparación con la década pasada, en este momento todas las áreas de la biología están siendo beneficiadas e influenciadas por las ciencias computacionales (Gribskov, 2005).
La historia de la investigación en bioinformática y biología computacional inicia desde la década de los cincuenta, donde aparecen los trabajos de Watson y Crick sobre la estructura del ADN (Franco et al., 2008; Ouzuonis y Valencia, 2003; Richon, 2001). En 1986, el térmico genómica apareció por primera vez (Lee y Lee, 2000), y en 1988 se funda el Centro Nacional de Informática y Biotecnología (NCBI) inicialmente bajo el nombre de Instituto Nacional del Cáncer. En este mismo año se inicia el proyecto de secuenciación del genoma humano el cual finaliza en el 2003 (Venter et al., 2001). El proyecto genoma humano acelera el desarrollo de varias técnicas y tecnologías y le da fuerza al campo de las omicas, entre ellas las proteómica, la transcriptómica y la metabolómica. A partir de la cantidad de información generada en estos proyectos se da impulso al campo de la bioinformática y biología computacional, y con esto a la era de la posgenómica, caracterizada por aplicar un enfoque holístico al estudio de los sistemas biológicos utilizando múltiples herramientas analíticas.
Bioinformática en otros países latinoamericanos
Los grandes avances en métodos de secuenciación de DNA, genomas y proteomas han contribuido al desarrollo de la biología computacional como una herramienta que nos permite analizar la enorme cantidad de información que se está generando. En los países más desarrollados los avances y las aplicaciones de estas herramientas crecen exponencialmente.
La revista PLoS Computational Biology publicó una serie de artículos en donde se hace una revisión de la situación de algunos países latinoamericanos como México, Brasil, Argentina, Cuba, Costa Rica y, recientemente, Colombia. Cabe resaltar los avances de Argentina, México y sobre todo Brasil, quienes han participado en grandes proyectos de genómica y proteómica.
Hay varios grupos en Argentina trabajando en el área de bioinformática y biología computacional. En la ciudad de Rosario, por ejemplo, se encuentra el centro de biología computacional, este centro es una iniciativa reciente del Instituto de Biología Molecular y la Facultad de Ciencias Exactas e Ingeniería de la Universidad Nacional del Rosario, y se encuentra hoy en día muy interesado en el diseño de algoritmos para la detección de algunas proteínas que tiene capacidad de interacción con el ADN. En cuanto al área de análisis de datos de microarrays, este centro está interesado en el diseño de algoritmos de clasificación basado en los códigos de corrección de errores. También se encuentra el grupo de bioinformática del Instituto de Biotecnología de la Universidad General de San Martín. Este grupo ha estado involucrado en la secuenciación del genoma de Trypanosoma cruzi desde 1997. También está trabajando en la secuenciación de diferentes bacterias patogénicas como Brucella abortus y Campylobacter fetus, y hospedadores comunes de bacterias como Tupaia belangeri (Bassi et al., 2007).
El primer gran proyecto de secuenciación en México fue realizado en el CIFN y consistió en la determinación de una secuencia de nucleótidos de 370-kb pertenecientes al plásmido simbiótico Rhizobium etli. Este proyecto fue la base para lograr el soporte del Consejo Nacional de Ciencia y Tecnología (Conacyt) para otro proyecto titulado Desarrollo de las ciencias genómicas en México: el genoma de Rhizobium etli como sistema modelo, el cual permitió generar la secuenciación del primer genoma completo para México y fue un paso clave en el desarrollo de las ciencias genómicas en este país. Actualmente hay una gran variedad de proyectos en genómica y posgenómica en las diferentes instituciones y centros de investigación mexicanos, además de que fueron creadas instituciones específicas para trabajar en esta área como el Laboratorio de Genómica para la Biodiversidad de México creado en el 2005. Interesante de recalcar es que la mayoría de los proyectos de investigación están siendo financiados con recursos públicos mexicanos (Palacios y Collado-Vides, 2007).
Por su parte, el gobierno brasilero ha mostrado gran interés en avanzar en el área de la biotecnología y la biología computacional. En 1997 se invirtieron recursos humanos y financieros en tecnología de punta para secuenciación. No se creó un laboratorio centralizado para el secuenciamiento de genomas, sino una red de laboratorios con este propósito. El ensamble de genomas se centralizó en una plataforma localizada en la universidad Unicamp en Campinas. El resultado de este esfuerzo fue el mapa genético de Xillela fastidiosa publicado en Nature. Los genomas de las bacterias Xanthomonas citri, Xanthomonas campestris, Lifsonia xyli, que afectan a la agricultura brasilera también fueron descifrados, además de contribuir, a través de redes internacionales, al mapeo del genoma del banano, el café y el arroz. En relación con la genómica animal, Brasil ha participado en la secuenciación del genoma de la vaca y del cerdo. A su vez, ha participado en iniciativas de secuenciación de genomas de interés medico, como es el genoma de Anopheles darlingi (mosquito vector de la malaria). Brasil, al igual que México, cuenta con una sólida asociación nacional de bioinformática y biología computacional, además de varios centros de investigación especializados en el área (Neshich, 2007).
Bioinformática en Colombia
En Colombia, la investigación en bioinformática es todavía bastante incipiente, sin embargo, hay bastante potencial en el área, con grupos de investigación sólidos que trabajan en bioinformática o áreas relacionadas (Restrepo et al., 2009). Reconocer que tenemos investigaciones e investigadores de alto nivel ha permitido pensar en la necesidad que tiene el país de invertir en centros con alta tecnología de secuenciación, y en la necesidad de un centro de biología computacional. Las gestiones para la creación de este centro de bioinformática y biología computacional se inician gracias a las ayudas que prometió Bill Gates en una carta enviada al presidente de la República el pasado 8 de mayo de 2009, fruto de una propuesta presentada por José Fernando Isaza, rector de la Universidad Jorge Tadeo Lozano, y del trabajo desarrollado por el colombiano Orlando Ayala, vicepresidente senior del grupo de desarrollo de mercados emergentes de Microsoft.
La importancia de la construcción de este centro radica en que considerando que en la época actual la bioinformática no solo se restringe al análisis de los datos moleculares, la integración de los datos de biodiversidad constituye uno de los aspectos de la investigación y el desarrollo en los cuales los grupos de bioinformática pueden encontrar un conjunto de problemas interesantes y pertinentes para resolver, ya que aunque Colombia es uno de los países con mayor biodiversidad del mundo, enfrenta el reto de iniciar varios frentes de acción de forma sistemática y coordinada, para consolidar el conocimiento completo de dicha biodiversidad. La bioinformática ofrece las herramientas y los conceptos para sistematizar ese conocimiento (Barreto, 2008).
Si bien en el país los centros de bioinformática y biología computacional son escasos y limitados, existen en Colombia universidades públicas y privadas como la Universidad de Antioquia, Universidad del Valle, Universidad Nacional de Colombia, Universidad de los Andes y la Universidad del Cauca, las cuales han creado centros de investigación que han estado generando datos relevantes a nivel molecular, además, también existen diversos institutos tales como Cenicafe, Cenicaña, Corpoica, CIAT y el Instituto Alexander von Humboldt que realizan investigaciones en el área de la genética y la biología molecular con diferentes aplicaciones (Restrepo et al., 2009).
Cenicafe es una institución asociada a la Federación Nacional de Cafeteros la cual ha realizado estudios en el área de la bioinformática, tales como el desarrollado por Rivera et al. (2008), denominado Sistema de información para el manejo de datos moleculares en café, en el cual se muestra cómo fue el desarrollo de un sistema de información de datos genómicos (LIMS), basado en su mayoría en herramientas libres. Este sistema está especializado para el manejo de información relacionada con secuencias de EST, BAC y Microsatélites de varias especies de café, la broca Hypothenemus hampei y el hongo Beauveria bassiana. Para el análisis de esta información se crearon pipelines específicos para proceder con el agrupamiento, análisis y anotación de las secuencias. Con esta información se generó un modelo relacional de bases de datos para su almacenamiento, se diseñaron interfaces web con motores de búsqueda especializados, y se incorporaron herramientas para despliegue gráfico de ensamblajes de genes, anotaciones, datos estadísticos y otra información relacionada.
En esta misma línea, Cristancho et al. (2006), en su trabajo Development of a bioinformatics platform at the Colombia National Research Center-Cenicafé, muestra cómo el Centro desarrolló una plataforma de Bioinformática basada en web, que funciona como un recurso de información genómica para el café y otros organismos de estudio en Cenicafe. La plataforma de bioinformática incluye un Sistema Integrado de Administración de Laboratorio (LIMS), la implementación de wEMBOSS, instrumentos desarrollados en el lenguaje perl en Cenicafé para el análisis de datos, InterproScan para la búsqueda y anotación de dominios de proteínas, y la implementación de wBLAST y wNetBLAST, entre otros instrumentos disponibles. El principal elemento del sistema es la adaptación del SOL Genomics Network (SGN), un sistema de bases de datos desarrollado en la Universidad de Cornell para el almacenamiento y análisis de EST, marcadores moleculares y secuencias BAC.
Por su parte, la Universidad Nacional ha realizado investigaciones como la presentada en el documento ENKI-DB: sistema de información taxonómica y molecular de especies propias de la biodiversidad colombiana, desarrollado por Barreto et al. (2006), en el cual los autores desarrollan un software accesible vía Internet, donde se tiene acceso inmediato a toda la información taxonómica y molecular presente en diversas bases de datos para especies propias de la biodiversidad colombiana exclusivamente. Hasta la fecha, en ENKI-DB se han logrado enlazar 10.808 registros de especies propias de la biodiversidad colombiana. Aunque la información puede ser encontrada de manera independiente de cada una de las bases de datos que conforman el software, el valor agregado de ENKI-DB es que los datos son accesibles desde una sola interfaz, de manera integrada y depurada.
Finalmente, la Universidad del Valle, en su Escuela de Ingeniería de Sistemas y Computación, ha realizado investigaciones en temas relacionados con la biología computacional y la bioinformática, como el presentado por Tischer et al. (2007), titulado Genezilla-Graphics: un software para la predicción de genes, o el presentado por Cuaran y Tischer (2007) titulado, Una aproximación probabilística para la predicción de elementos de promotores humanos, además de estas publicaciones, la Escuela publicó un libro con las memorias del Segundo Seminario Internacional de Genómica, Proteómica, Bioinformática y Biología de Sistemas, llevado a cabo en el mes de octubre del año 2006 en la Universidad del Cauca.
Investigación en el área o áreas relacionadas: potenciales usuarios del Centro
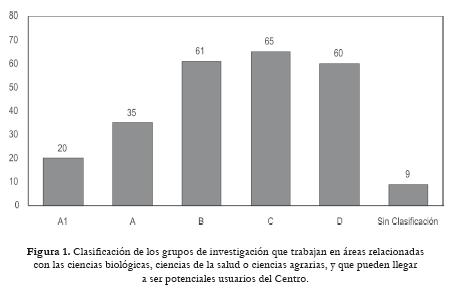
Cualquier investigador, tanto nacional como internacional, puede ser potencial usuario de un Centro Nacional de Bioinformática y Biología Computacional. Es difícil pensar que investigadores internacionales (con excepción de los de algunos países vecinos que no posean un centro propio) utilicen los servicios del Centro pero, sin duda alguna, cualquiera podría ser usuario de la información almacenada en el mismo. Basándonos en la base de datos de Colciencias nos damos cuenta que en Colombia hay 250 grupos de investigación que trabajan en áreas que incluyen las ciencias agrarias, ciencias biológicas o ciencias de la salud, entre otras, y que, aunque no necesariamente trabajen en el área de la biología molecular, secuenciación de genes o genomas, son potenciales usuarios del centro. Como tal, un centro en biología computacional, no solo implica la aplicación de ciencias computacionales para el análisis de información proveniente de genes o proteínas, sino que incluye la utilización de herramientas computacionales para cualquier tipo de análisis o almacenamiento de datos biológicos. De los 250 grupos detectados en la base de datos de Colciencias, 55 son grupos con clasificación A1 o A. Estos grupos generalmente sobresalen no solo a nivel nacional sino también internacional por su productividad y la calidad de sus investigadores. Sin embargo, la mayoría de los grupos de investigación en el área, o áreas relacionadas, están clasificados como grupos C o D, es decir, corresponden a grupos con un bajo índice de productividad (fig. 1). Todos ellos pueden ser usuarios potenciales del centro. Además, es de suponer que el centro podría servir de motor o catalizador para que aquellos grupos que necesitan fortalecerse y hacerse competitivos puedan hacerlo en un tiempo menor y alcanzar así los estándares de productividad que marca el contexto internacional.
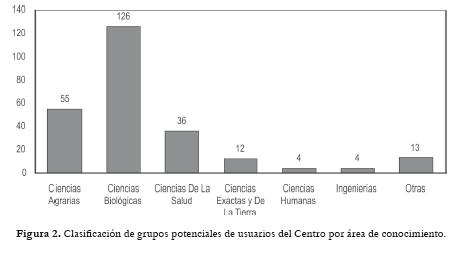
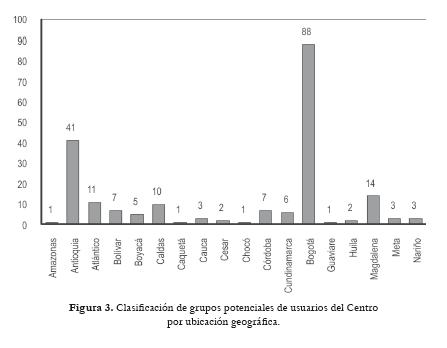
En cuanto a área del conocimiento, los grupos potenciales usuarios del centro trabajan principalmente en ciencias agrarias, ciencias biológicas, o ciencias de la salud (figura 2). Además, se observa claramente que un alto porcentaje de los grupos pertenecen a centros de investigación y universidades ubicadas en la capital del país, Bogotá. El departamento de Antioquia también cuenta con un número sustancial de grupos de investigación y está seguido de Valle (figura 3).
Oferta de posgrados en el área o áreas relacionadas
En este momento se ofrecen algunos posgrados en áreas cercanamente relacionadas con la biología computacional como pueden ser la biotecnología, la biología molecular o la genética. Por ejemplo, la Universidad Tecnológica de Pereira ofrece maestría presencial en Biología Molecular y Biotecnología, la Universidad Nacional de Colombia con sede en Medellín ofrece maestría presencial en Biotecnología, y la Universidad Nacional de Colombia con sede en Bogotá ofrece un doctorado en Biotecnología.
Sin embargo, como tal, no se ofrece una maestría o doctorado con el nombre específico de bioinformática o biología computacional. En este momento la Universidad de los Andes está ofreciendo maestría y doctorado con énfasis en bioinformática en el Departamento de Ciencias Biológicas, y la Universidad Tecnológica de Pereira tiene también una maestría con énfasis en bioinformática en la Facultad de Ingeniería.
La creciente investigación en el área a nivel internacional y, a menor escala, a nivel nacional, nos demuestra la necesidad de formar estudiantes en el área específica de biología computacional, que se conviertan no solo en potenciales usuarios del centro sino también en potenciales investigadores del mismo. Actualmente en Colombia tenemos algunos doctores (la mayoría formados en el exterior) con énfasis en bioinformática que hacen una masa crítica base para el desarrollo de lo que sería el Centro de Bioinformática y Biología Computacional, y a su vez son soporte para posgrados nacionales en el área.
Recursos para la investigación en bioinformática en Colombia
Como se mencionó, una de las claves para el éxito y el desarrollo de la bioinformática en países como México y Brasil sin mirar obviamente lo que pasa en países más desarrollados, es el compromiso e interés del gobierno para inversión en investigación.
Colciencias, recientemente nombrado como Departamento Administrativo de Ciencia, Tecnología e Innovación, con el propósito de proveer herramientas de última generación para conocer, proteger y valorar nuestra biodiversidad, apoyó la conformación del primer Centro Nacional de Secuenciación Genómica en Colombia. Este Centro será una infraestructura física robusta para llevar a cabo proyectos sobre información genética de recursos biológicos colombianos. Con el Centro Nacional se podrá responder a la demanda de secuenciación genómica de los sectores que la requieren a nivel nacional e internacional. Su sede será la Universidad de Antioquia.
Ahora, Colciencias invertirá recursos nacionales e internacionales en un Centro de Bioinformática y Biología Computacional. La convocatoria para el diseño del modelo organizacional del Centro fue otorgada a las universidades del Norte y del Magdalena, con asesoramiento por reconocidos investigadores en el área tanto a nivel nacional como internacional. Lo importante es que están los recursos para la creación del centro.
Adicionalmente, Colciencias acaba de entregar 1.285 becas a profesionales colombianos que adelantarán estudios de formación de alto nivel tanto en Colombia como en el exterior. Se espera que muchos de estos estudiantes puedan ser potenciales usuarios e investigadores del Centro (Colciencias, 2009).
La captación de recursos para la investigación en esta área, tenemos que ser realistas, no puede depender únicamente de lo que tenga Colciencias. Muchos de los recursos (posiblemente la mayoría) tendrán que venir del exterior, en este sentido es importante conocer y pertenecer a redes y asociaciones tanto a nivel nacional como internacional, que faciliten la interlocución entre centros y grupos de investigación y fomenten el trabajo colaborativo y la captación de recursos. Ejemplo de estas redes incluyen la EMBnet, RIBO y Soibio, especializadas en bioinformática, donde instituciones como la Universidad Nacional de Colombia ha participado desde hace más de cinco años.
Conclusiones
Aunque existen ejemplos de investigaciones y grupos de investigación que trabajan en el área de la bioinformática en Colombia, hay todavía un gran camino por recorrer para consolidar esta disciplina en el país. Como en otros países de América Latina, la mayoría de los investigadores en Colombia que trabajan en el área de las ciencias computacionales no están familiarizados con las ciencias biológicas, y, de la misma manera, la mayoría de los biólogos tienen conocimientos limitados de programación y modelamiento computacional. Ningún programa a nivel de pregrado o de posgrado contempla realmente un currículo integrado de estas disciplinas. Como consecuencia, actualmente son pocas las oportunidades de nuestros estudiantes para desarrollarse en el área de la biología computacional.
La creación de un centro nacional en bioinformática y biología computacional es una estrategia para apoyar y fomentar la investigación en esta área. Esta estrategia debe estar conectada con un reajuste en los currículos de los programas de ciencias biológicas para permitir una mayor inclusión de cursos obligatorios de ciencias computacionales. La necesidad de entrenar a los biólogos en los métodos computacionales es inminente (Lewitter, 2007). Por otro lado, es necesaria la creación de redes y asociaciones en biología computacional, y la integración de esfuerzos entre los científicos nacionales que conocen y han o están trabajando en esta área. Aunque la investigación en bioinformática y biología computacional en Colombia es aún muy incipiente, ya se está desarrollando una masa crítica de investigadores. Una prueba de ello es la organización del Primer Congreso Colombiano de Biología Computacional que se llevará a cabo en Bogotá en marzo del 2011.
Adicionalmente, entre las estrategias del gobierno debe estar el apoyo al entrenamiento de estudiantes nacionales en reconocidos centros y universidades internacionales en el área de biología computacional. También es indispensable fomentar la colaboración entre grupos de investigación nacionales y grupos de bioinformática avanzados en otros países. En este contexto, es importante la financiación de proyectos donde haya participación mínima de dos grupos nacionales y uno internacional. Dentro de las alianzas entre grupos de investigación es indispensable la presencia de investigadores de áreas biológicas y de ingeniería. Los desarrollos recientes exitosos que se han presentado en bioinformática en el país como los de la Universidad de los Andes, la Universidad Nacional y Cenicafe han incluido este tipo de alianzas.
Agradecimientos
Este trabajo se realizó gracias a la financiación de Colciencias y la Universidad del Norte.
Referencias bibliográficas
2 Barreto, E. 2008. Bioinformática: una oportunidad y un desafio. Rev Colombi Biotecnol 10 (1): D132- D138.
3 Bassi, S., Gonzalez V., Parisi, G. 2007. Computational Biology in Argentina. PLoS Comput Biol 3 (12): e257. doi:10.1371/journal.pcbi.0030257.
4 Benson, D. A., Karsch-Mizrachi, I., Lipman, D. J., Ostell, J., Rapp, B. A., Wheeler, D. L. 2000. GenBank. Nucleic Acids Res 28 (1): D15-D8.
5 Benson, D. A., Karsch-Mizrachi, I., Lipman, D. J., Ostell J., Sayers, E. W. 2010. GenBank. Nucleic Acids Res 38 (1): 46-51.
6 Colciencias. 2009. Convocatoria para estudios de doctorado en Colombia o en el exterior 2010 y pasantías de investigación. Disponible en: http://www.colciencias.gov.co/ Fecha de consulta: 3 de mayo de 2010, fecha actualización: 3 de mayo de 2010.
7 Cristancho, M., Rivera, L., Orozco, C., Chalarca A., Mueller, L. 2006. Development of a bioinformatics platform at the Colombia National Research Center – Cenicafé. 21st International Conference on Coffee Science, Montpellier, France.
8 Cuaran, M., Tischer, I. 2007. Una aproximación probabilística para la predicción de elementos de promotores humanos. V Congreso Internacional - VIII Congreso Colombiano de Genética. Colomb Med 39 (2).
9 Franco, M. L., Cediel, J. F., Payan, C. 2008. Breve historia de la bioinformática. Colomb Med 39: 117-120.
10Gibas, C., Jamebeck, P. 2001. Bioinformatic Computer Skills. Ed. OReilly.
11 Gribskov, M. 2005. An Open Forum for Computational Biology. PLoS. Comput Biol 1 (1): e5. doi:10.1371/ journal.pcbi.0010005.
12 Lee, P. S., Lee, K. H. 2000. Genomic Analysis. Curr Opin Biotechnol 11: 171-175.
13 Lewitter, F. 2007. Moving Education Forward. PLoS. Comput Biol 3 (1): e19. doi:10.1371/journal.pcbi. 0030019.
14 Luscombe, M. N., Greenbaum D., Gerstein, M. 2001. What is bioinformatics? An introduction and overview. Yearbook of Medical Informatics. Stuttgart, New York, Geneva: Schattauer.
15 Neshich, G. 2007. Computational biology in Brazil. PLoS Comput Biol 3 (10): e185. doi:10.1371/journal. pcbi.0030185.
16 Ouzounis, C. A., Valencia, A. 2003. Early bioinformatics: the birth of a disciplinea personal view. Bioinformatics 19 (17): 2176-2190.
17 Palacios, R., Collado-Vides, J. 2007. Development of genomic sciences in Mexico: a good start and a long way to go. PLoS Comput Biol 3 (9): e143. doi:10.1371/journal.pcbi.0030143.
18 Restrepo, S., Pinzón, A., Rodríguez-R, L. M., Sierra, R., Grajales, A. et al. 2009. Computational Biology in Colombia. PLoS Comput Biol 5 (10): e1000535. doi:10.1371/journal.pcbi.1000535.
19 Rivera, L. F., Orozco, C. E., Chalarca, A., Gaitán A. L., Cristancho, M. A. 2008. Sistema de información para el manejo de datos moleculares en café: 1. Desarrollo y uso de herramientas. Rev Acad Colomb Cienc 32 (124): 317-324.
20 Richon. 2001. A short history of bioinformatics. Network Science Corporation, ISSN 1092-7360.
21Tischer, I., Moreno, P., Cuaran, M., Bedoya, O., Garreta, L., Cabezas I., Bedoya, O. 2007. Genezilla-Graphics: un software para la predicciòn de genes. Memorias V Congreso Internacional y VIII Colombiano de Genética.
22 Venter, C., Adams, M. D., Meyers, E. W. 2001. The sequence of the human genome. Science 291: 1304- 1351.
23 Young, P. G. 2003. Exploring Genomes. New York: Freeman and Company.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2010 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).