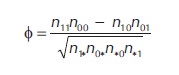Generación de anticuerpos policlonales para la detección de la variante genotípica GIII de PVY, en cultivos de tomate de árbol y papa de Colombia
Palabras clave:
anticuerpos policlonales, ELISA, potyvirus, RT-PCR, virus Y de la papa, policlonal antibodies, Potato virus Y (es)Resumen: El potyvirus PVY es uno de los agentes causales más frecuentemente asociados a problemas virales en cultivos de papa y tomate de árbol en Colombia. Dada la importancia económica de las enfermedades causadas por PVY y a la necesidad de generar material de siembra certificado por su sanidad viral, es fundamental la generación de herramientas de diagnóstico que permitan la detección temprana de este virus. En este trabajo se reporta la obtención de anticuerpos policlonales específicos, útiles para la detección del genotipo III de PVY (GIII), una de las tres variantes que recientemente han sido reportadas en cultivos de papa y tomate de árbol de la región Andina de Colombia. Como antígeno, se utilizó un péptido sintético diseñado a partir de la región variable del extremo N-terminal del gen de la cápside viral. La sensibilidad de los anticuerpos fue evaluada mediante pruebas de ELISA y dot-blot utilizando péptidos sintéticos. Se realizó una prueba piloto para validar el uso de los anticuerpos a partir de plantas sintomáticas y asintomáticas obtenidas de una región donde confluyen cultivos de ambas solanáceas, encontrándose que los anticuerpos generados ofrecen mayores niveles de detección que los anticuerpos comerciales comúnmente utilizados para detectar los serotipos PVY-O,C y PVY-N de este virus.
Palabras clave: anticuerpos policlonales; ELISA; potyvirus; RT-PCR; virus Y de la papa
Abstract: PVY is one of the potyvirus more frequently associated with viral infections in tomato and tamarillo crops in Colombia. Due to the economic impact of PVY and the need to certify seeds as virus-free it is important to develop diagnostic tools that allow its premature detection. In this work, the obtention of antibodies detecting the genotype III of PVY is reported. This genotype is one of the three PVY variants infecting tamarillo and potato in the Andean region of Colombia. The N-terminal variable region of the coat protein was chosen as antigen for antibody production. The sensibility of these antibodies was tested by ELISA and dot-blot using synthetic peptides. A pilot test was performed on symptomatic and non-symptomatic plants from a mixed orchard of tamarillo and potato. The generated antibodies showed higher detection levels than the commercial antibodies commonly used to detect the PVY-O,C and PVY-N serotypes.
Key words: ELISA; policlonal antibodies; potyvirus; RT-PCR, Potato virus Y
ARTÍCULO CORTO
Generación de anticuerpos policlonales para la detección de la variante genotípica GIII de PVY, en cultivos de tomate de árbol y papa de Colombia
Generation of policlonal antibodies for detection of the PVY genotypic variant GIII in tamarillo and potato crops from Colombia
Yuliana Gallo García1, Mauricio Marín Montoya2, Pablo Gutiérrez Sánchez3.
1 Investigadora, MSc, Laboratorio de Microbiología Industrial, Facultad de Ciencias, Universidad Nacional de Colombia Sede Medellín. ymgallo@gmail.com
2 Profesor Asociado, PhD, Laboratorio de Biología Celular y Molecular, Facultad de Ciencias, Universidad Nacional de Colombia Sede Medellín. mamarinm@unal.edu.co
3 Profesor Asociado, PhD, Laboratorio de Microbiología Industrial, Facultad de Ciencias, Facultad de Ciencias, Universidad Nacional de Colombia Sede Medellín. paguties@unal.edu.co
Recibido: febrero 13 de 2012 Aprobado: junio 29 de 2012
Resumen
El potyvirus PVY es uno de los agentes causales más frecuentemente asociados a problemas virales en cultivos de papa y tomate de árbol en Colombia. Dada la importancia económica de las enfermedades causadas por PVY y a la necesidad de generar material de siembra certificado por su sanidad viral, es fundamental la generación de herramientas de diagnóstico que permitan la detección temprana de este virus. En este trabajo se reporta la obtención de anticuerpos policlonales específicos, útiles para la detección del genotipo III de PVY (GIII), una de las tres variantes que recientemente han sido reportadas en cultivos de papa y tomate de árbol de la región Andina de Colombia. Como antígeno, se utilizó un péptido sintético diseñado a partir de la región variable del extremo N-terminal del gen de la cápside viral. La sensibilidad de los anticuerpos fue evaluada mediante pruebas de ELISA y dot-blot utilizando péptidos sintéticos. Se realizó una prueba piloto para validar el uso de los anticuerpos a partir de plantas sintomáticas y asintomáticas obtenidas de una región donde confluyen cultivos de ambas solanáceas, encontrándose que los anticuerpos generados ofrecen mayores niveles de detección que los anticuerpos comerciales comúnmente utilizados para detectar los serotipos PVY-O,C y PVY-N de este virus.
Palabras clave: anticuerpos policlonales, ELISA, potyvirus, RT-PCR, virus Y de la papa.
Abstract
PVY is one of the potyvirus more frequently associated with viral infections in tomato and tamarillo crops in Colombia. Due to the economic impact of PVY and the need to certify seeds as virus-free it is important to develop diagnostic tools that allow its premature detection. In this work, the obtention of antibodies detecting the genotype III of PVY is reported. This genotype is one of the three PVY variants infecting tamarillo and potato in the Andean region of Colombia. The N-terminal variable region of the coat protein was chosen as antigen for antibody production. The sensibility of these antibodies was tested by ELISA and dot-blot using synthetic peptides. A pilot test was performed on symptomatic and non-symptomatic plants from a mixed orchard of tamarillo and potato. The generated antibodies showed higher detection levels than the commercial antibodies commonly used to detect the PVY-O,C and PVY-N serotypes.
Key words: ELISA, policlonal antibodies, potyvirus, RT-PCR, Potato virus Y
Introducción
Potato virus Y (PVY) es la especie tipo del género Potyvirus (Potyviridae) (Fauquet et al., 2005). Este virus afecta un amplio número de especies de la familia Solanaceae y dependiendo del cultivar, la variante viral y las condiciones medio ambientales, puede inducir un rango de síntomas que incluyen mosaicos, moteados, enanismos, necrosis e incluso la muerte de sus hospedantes. Este virus se dispersa principalmente por áfidos de manera no persistente, por propagación asexual y por contacto mecánico (Shukla et at., 1994). PVY tiene genoma de ARN de cadena sencilla positiva de aproximadamente 9700 nt que codifica para una poliproteína de 3061 aminoácidos (Riechmann et al., 1992), que sufre un proceso de proteolisis para generar 10 proteínas maduras, con diferentes funciones de replicación, transporte y diseminación del virus (Urcuqui-Inchima et al., 2001).
Los aislamientos de PVY provenientes de papa han sido históricamente clasificados en tres razas: PVYN, PVYO y PVYC, de acuerdo a las reacciones sistémicas y locales que induce en Nicotiana tabacum y Solanum tuberosum(Miczynski, 1963). Otro grupo denominado PVYZ, fue propuesto por Jones (1990) para incluir algunos aislamientos encontrados inicialmente en Gran Bretaña, que sobrepasaban la respuesta hipersensible conferida contra PVYC y PVYO por la presencia en los hospedantes de los genes Nc y Nytbr, respectivamente (Blanco-Urgoiti et al., 1998). La raza PVYN incluye dos subgrupos correspondientes a las variantes PVYW (también conocida como PVYN:O ) y PVYNTN (Kahn et al., 1963). PVYNTN se caracteriza por su habilidad para inducir anillos necróticos en tubérculos de papa, enfermedad conocida como PTNRD por sus siglas en inglés (Potato Tuber Necrotic Ringspot Disease) y que puede ocasionar pérdidas hasta del 100% en este cultivo (Kogovsek et al., 2008). PVYN:O es definido por su reacción positiva con anticuerpos que reconocen el serotipo PVY-O y la inducción de síntomas de necrosis de venas en tabaco (Hu et al., 2011). Así mismo, dos tipos de PVYNTN, uno recombinante y otro no recombinante, han sido identificados (Nie y Singh, 2003). El primero se conoce como la variante Europea (Eu), mientras que el segundo es referido como la variante Norteamericana (NA) de PVYNTN. Ambas variantes reaccionan contra anticuerpos específicos para el serotipo PVY-N (Hu et al., 2009)
A pesar de la gran diversidad de variantes de PVY reportadas, es posible diferenciar dos serotipos principales entre todas ellas: PVY-O,C que incluye los miembros de las razas PVYO, PVYC, PVYZ, y PVYW, y el serotipo PVYN, que contiene los aislamientos de las razas PVYN y PVYNTN (Ellis et al., 1977). El anticuerpo monoclonal MAb2 desarrollado por McDonald et al. (1994), puede identificar el serotipo-PVY-O,C sin reacción cruzada con aislamientos del serotipo-PVY-N; mientras que el anticuerpo 1F5, reportado por Ellis et al. (1996) puede identificar todos los aislamientos del serotipo PVY-N, aunque algunos aislamientos de la raza PVYO pueden también reaccionar con éste anticuerpo. Aparentemente una sustitución E29G en la secuencia de la cápside viral es la responsable de las diferencias entre ambos serotipos (Chikh et al., 2007).
Adicionalmente a las razas de PVY obtenidas en papa, se han identificado diferentes variantes en otros hospedantes como Capsicum spp., S. lycopersicum y N. tabacum (Singh et al., 2008). Algunos de estos aislamientos son incapaces de desarrollar infecciones sistémicas en papa y presentan diferencias notables en las secuencias de sus genomas, especialmente en la región que codifica para la cápside viral (Romero et al., 2001). Esta situación implica diferencias en las respuestas de dichos aislamientos frente a anticuerpos desarrollados para la detección de los serotipos de PVY provenientes de papa, tal como lo demostraron Ounouna et al. (2002) para cepas de PVY afectando Capsicum spp. en Túnez.
Con base en análisis de secuencias de la cápside viral, en Colombia se han identificado al menos tres genotipos diferentes de PVY (denominados GI, GII y GIII) afectando cultivos de papa y tomate de árbol (S. betaceum) (Gilet al., 2011; Jaramillo et al., 2011). Adicionalmente, Ayala et al. (2010), con base en secuenciación del gen de la cápside viral, identificaron un nuevo Potyvirus afectando plantas de tomate de árbol obtenidas en el departamento de Antioquia, para el cual propusieron el nombre de Tamarillo leaf malformation virus (TaLMV). En dichos trabajos, los genotipos I y II de PVY incluían aislamientos obtenidos de ambos hospedantes y presentaban altos niveles de afinidad filogenética con las razas PVYN y PVYNTN de diferentes países del mundo. El genotipo III solo se encontró en tomate de árbol y tiene una relación distante con las cepas representativas de las diferentes razas conocidas de PVY. En un estudio posterior utilizando un mayor número de cepas, Henao et al. (2011) encontraron aislamientos de PVY (GIII) en cultivos de papa en Colombia. Con base en estos resultados y en el hecho que en Colombia, es frecuente encontrar explotaciones comerciales de ambos cultivos en las mismas zonas agroecológicas, se ha sugerido la existencia de transmisión cruzada de PVY entre estos hospedantes (Jaramillo et al., 2011).
Debido al incremento en la incidencia y severidad de las enfermedades virales causadas por PVY en cultivos de tomate de árbol y papa en Colombia, es fundamental la generación de herramientas de diagnóstico para apoyar los programas de manejo integrado de enfermedades, especialmente los esquemas de vigilancia cuarentenaria y de generación de material de siembra certificado por su sanidad viral. Pese a la existencia de anticuerpos comerciales para la detección de este virus, observaciones previas realizadas por nuestro grupo y basadas en comparaciones de los niveles de sensibilidad de pruebas de ELISA con anticuerpos genéricos contra Potyvirus y específicos para PVY, han advertido que estos últimos no siempre son efectivos en la detección de las variantes de PVY presentes en Colombia, en particular para aquellas con el genotipo III (GIII). En este trabajo se reporta la obtención de anticuerpos específicos para PVY (GIII) y su utilización en pruebas de detección basadas en ELISA,dot-blot y flujo lateral. Estos anticuerpos fueron generados a partir de la inoculación de un péptido sintético diseñado con base en la identificación de una región antigénica de la región N-terminal de la proteína de la cápside de cepas representativas de dicho genotipo.
Materiales y métodos
Colección de material vegetal
Las muestras de las plantas de tomate de árbol y papa con síntomas de mosaicos presumiblemente asociados a la infección por PVY, fueron obtenidas en el corregimiento de Santa Elena, Medellín (06°07′08′′N, 74°55′45′′W) y utilizadas en el proceso de estandarización de las pruebas serológicas desarrolladas en este trabajo. Para la realización de la prueba piloto de detección de PVY, se obtuvieron en total 20 muestras de tejido foliar de papa y 20 muestras de tomate de árbol en diferentes cultivos de los municipios de La Ceja (06°32′10′′N, 75°42′21′′W) y La Unión (05°58′38′′N, 75°21′54′′W) (Antioquia, Colombia).
Producción de anticuerpos
La región con mayor potencial antigénico para la síntesis de péptidos fue obtenida mediante un análisis de hidrofilicidad siguiendo el método de Hopp y Woods (1981) e implementado en el software BioEdit 6.0.6 (Hall, 1999). El péptido PVY(p) fue sintetizado mediante el método de fase sólida y verificado por espectrometría de masas y HPLC. Los anticuerpos fueron producidos por la compañía GenScript (NJ, EEUU), en dos conejos utilizando 5 mg del péptido sintético conjugado con hemocianina KLH. Se adicionó una cisteína C-terminal para facilitar la conjugación a la hemocianina. Los anticuerpos fueron purificados por afinidad y resuspendidos en PBS pH 7.4, obteniéndose una cantidad total de 36.28 mg. El título de anticuerpos fue determinado mediante ELISA utilizando 100 µl de péptido a una concentración de 4 mg/ml por pozo, en PBS pH 7.4.
ELISA
Se utilizaron anticuerpos específicos producidos contra el péptido diseñado para PVY. La concentración mínima para la detección se determinó a partir de diluciones seriadas del péptido a partir de una concentración inicial de 4 mg/ml. La especificidad de los anticuerpos fue establecida mediante comparación con los controles positivos de la compañía Agdia (Indiana, EE.UU.) para la detección genérica de Potyvirus, el virus PVY y anticuerpos específicos contra TaLMV (Gallo et al., 2011). Para los anticuerpos anti-PVY(p) y anti-TaLMV se utilizó una dilución de anticuerpos 1:512000 a partir de un stock de 1 mg/ml de anticuerpos. Como antígeno se utilizaron 100 µl de péptido a una concentración de 4 mg/ml por pozo, en PBS pH 7.4. Para los anticuerpos comerciales se utilizó la concentración sugerida por el fabricante. Para la detección en plantas se tomaron 100 mg de tejido foliar macerados con el buffer general de extracción (1.3 g de sulfito de sodio, 20 g de PVP, 0.2 g de azida de sodio, 2 g de albúmina de huevo, 20 g de Tween 20, en 1000 ml de PBST 1X pH 7.4) y se siguió el procedimiento estándar de DAS-ELISA (Matthews, 1993). Cada prueba incluyó el péptido como control positivo y el buffer de extracción como control negativo. Para el revelado se utilizaron anticuerpos anti-conejo producidos en cabra conjugados a fosfatasa alcalina (Bio-Rad, EEUU). La prueba fue considerada positiva si la lectura de absorbancia a 405 nm era superior al doble del control negativo, siguiendo el criterio de Matthews (1993).
dot-blot
Se adicionaron 100 µl de péptido (4 mg/ml) a una membrana de nitrocelulosa que fue bloqueada posteriormente con leche descremada en polvo al 5% en buffer TBS-T 1X (150 mM NaCl, 10 mM Tris-HCl, 0.1% Tween 20, pH 7.4) durante 1 h a temperatura ambiente con agitación. Para el revelado, se incubó la membrana durante tres horas a 4 ?C, con una dilución 1:1000 del anticuerpo primario anti-PVY(p) diluido en TBS-T. Luego de realizar tres lavados con TBS-T 1X, se llevó a cabo la incubación del segundo anticuerpo anti-conejo conjugado con fosfatasa alcalina con las mismas condiciones y concentraciones descritas anteriormente. La detección se realizó utilizanto el kit alkaline phosphatase conjugate substrate de Bio-Rad, siguiendo las recomendaciones del fabricante.
Flujo lateral
El oro coloidal fue preparado según el procedimiento descrito por Dawei et al. (2009). El anticuerpo conjugado se preparó mediante adición de 1.5 ml del anticuerpo a 100 ml de la solución de oro coloidal pH 9.0. Luego de incubar toda la noche a 4°C, la mezcla fue centrifugada a 10000 gravedades durante 40 min. El pellet fue resuspendido en 10 ml de carbonato de sodio 2 mM, 1% BSA y 0.1% de azida de sodio, pH 9.2 y almacenado a 4°C. Las pruebas de flujo lateral se construyeron siguiendo el protocolo de Dawei et al. (2009). La almohadilla de la muestra se saturó con 100 mM de PBS pH 7.4, 0.1% Tween-20 y 1% (w/v) BSA; se dejó secar a 60°C y se almacenó a temperatura ambiente. La almohadilla conjugada se trató con 0.1% Tween-20 por 24 h. La almohadilla conjugada seca se sumergió en una solución de anticuerpos marcados con oro coloidal diluidos en proporcion1:1 (v/v) en 20 mM PBS, 8% sacarosa y 1% BSA. A la membrana de nitrocelulosa se le adicionó 1 µl de anticuerpo anti-conejo en la zona control y 1 µl de péptido a una concentración de 1mM en la zona de prueba; se secó a 37°C por 30 min y posteriormente se bloqueo con BSA 2% durante 30 min a 37°C.
Prueba piloto de detección de PVY
Con el fin de evaluar la utilidad de los anticuerpos generados para detectar el genotipo III de PVY en Colombia, se colectaron 20 muestras de papa y 20 de tomate de árbol de diferentes cultivos de una zona de los municipios de La Unión y La Ceja, donde confluyen ambos cultivos. Tres de las plantas de papa y cinco de tomate de árbol eran visualmente asintomáticas para enfermedades virales. Como control negativo se utilizaron plantas de papa criolla (S. phureja, Pcr) y papa capira (S. tuberosum, Pca) producidas mediante cultivo in vitro de meristemos y certificadas como libres de virus (Pca-vi y Pcr-vi). Los resultados se compararon mediante ELISA con los obtenidos mediante el uso de los anticuerpos comerciales anti-poty y anti-PVY, realizándose un análisis estadístico de correlación utilizando el coeficiente phi (Φ) de Pearson definido con la siguiente expresión:
donde n11 corresponde al total de muestras positivas para ambas pruebas, n10 y n01 al total de muestras positivas para una de las pruebas, n00 al número de muestras negativas para ambas pruebas, n1* al total de muestras positivas para la primera prueba, n0* al total de muestras negativas para la primera prueba, n*0 al total de muestras negativas para la segunda prueba y n*1 el total de muestras positivas para la segunda prueba.
Confirmación mediante RT-PCR de PVY
Con el fin de confirmar la presencia de la variante PVY (GIII) y de otras presentes en las muestras de papa y tomate de árbol inicialmente utilizadas para estandarizar las metodologías de detección serológica, se realizó RT-PCR y secuenciación del gen de la cápside. Similarmente, cuatro de las muestras que resultaron positivas en las pruebas serológicas con los anticuerpos genéricos para Potyvirus, fueron evaluadas mediante RT-PCR.
El ARN molde se obtuvo mediante la utilización del kit RNeasy plant mini (Qiagen, California, USA). Las reacciones de RT-PCR se realizaron en dos pasos, utilizando los cebadores dirigidos al gen de la cápside viral: PVYCPF_BamHI (5'-CGCGGATCCGGAAATGACACAATTGATG-3') y PVYCPR_HindIII (5'-CCCAAGCTTTCACATGTTCTTGACTCC-3'). Estos cebadores fueron diseñados con base en las secuencias de PVY de los tres genotipos del virus identificados en Colombia y reportadas por Gil et al. (2011) y Jaramillo et al. (2011). Cada reacción de RT se realizó en un volumen de 25 µl, incluyendo 3 µl de agua destilada estéril, 1.5 µl de PBST (0.5%), 1X de Buffer RT, 4 mM de MgCl2, 1 mM de dNTPs, 0.8 μM de cebador reverso (PVYCPR_HindIII) y de Oligo dT, 20 U de inhibidor de RNasa (Fermentas, Lituania), 10 U de enzima M-MuLV Transcriptasa Reversa (Fermentas) y 5 μl de ARN. La reacción se llevó a cabo a 37°C por 60 min y 75°C por 15 min. Las reacciones de PCR se realizaron en un volumen de 25 μl con 16.2 μl de agua destilada estéril, 1X de buffer de enzima, 1.8 mM de MgCl2, 0.2 mM de dNTPs, 0.2 μM de cada cebador, 1 U de Taq ADN polimerasa (Fermentas) y 2.5 μl de ADNc. El programa de PCR consistió en 95°C por 30 s, seguido de 35 ciclos de 95°C por 30 s, 52°C por 45 s, 72°C por 45s y una extensión final a 72°C por 5 min. Los amplicones del tamaño esperado fueron purificados directamente del gel para proceder a su secuenciación en ambos sentidos utilizando los mismos cebadores del RT-PCR. Las secuencias obtenidas fueron editadas mediante el software BioEdit 6.0.6, construyéndose consensos y confirmándose su identidad por comparación mediante BLASTN (Altschul et al., 1990). Finalmente se realizaron matrices de distancia con el método de Tajima-Nei (Tajima y Nei, 1984) y se identificaron los genotipos de PVY correspondientes. La tasa de variación por sitio fue modelada utilizando una distribución gama de cinco parámetros
Resultados y discusión
Producción de anticuerpos anti-PVY
Estudios serológicos realizados en Potyvirus con anticuerpos mono y policlonales dirigidos a la cápside viral, han demostrado que la región N-terminal de esta proteína contiene epítopes especie-específicos (Shukla et al., 1988), mientras que la región central es altamente conservada y contiene epítopes genéricos para todos los Potyvirus(Shukla et al., 1988; Jordan y Hammond, 1991). La región N-terminal es lo suficientemente variable para diferenciar serotipos dentro de la especie PVY; característica ampliamente utilizada para la discriminación de PVY-O,C y PVY-N (Chikh et al., 2007). Estas condiciones sustentaban la hipótesis de que era posible generar anticuerpos para el reconocimiento de variantes de PVY utilizando péptidos correspondientes a secuencias del extremo amino de la cápside viral y particularmente para la detección del PVY (GIII) encontrado en Colombia por Jaramillo et al. (2011). Para esto se compararon las secuencias de varios Potyvirus y de diferentes razas de PVY y se correlacionó este resultado con un análisis de hidrofilicidad siguiendo el método de Hopp y Woods (1981) (figura 1). Este método puede ser utilizado para predecir los determinantes antigénicos en la cápside, asumiendo que estos se encuentran expuestos en la superficie y corresponden a regiones hidrofílicas. En el caso de PVY, la región de mayor variabilidad corresponde a una de las de mayor antigenicidad y por lo tanto cumple con los requerimientos necesarios para la generación de anticuerpos específicos. Basados en este análisis, se diseño un péptido de 15 aminoácidos, PVY(p), con el cual se obtuvieron 36.28 mg de anticuerpos policlonales de conejo.
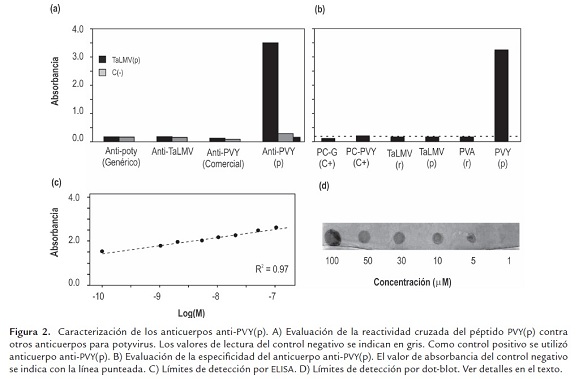
Para descartar la posibilidad de reacción cruzada del péptido PVY(p) y de esta forma confirmar la unicidad de la región seleccionada, se llevó a cabo una prueba de ELISA con tres anticuerpos diferentes utilizados en la detección de Potyvirus asociados a cultivos de papa y tomate de árbol: anti-poty (genérico, comercial), anti-PVY (comercial) y anti-TaLMV (Gallo et al., 2011), usando PVY(p) como antígeno (figura 2A). Dicha prueba demostró que, a excepción de los anticuerpos inducidos con el péptido PVY(p), ninguno reconoce la región N-terminal del PVY (GIII). Por otra parte, la especificidad de los anticuerpos anti-PVY(p) fue evaluada por ELISA utilizando los controles positivos de dos pruebas comerciales para la detección genérica de Potyvirus y PVY (PC-G y PC-PVY respectivamente), cápside recombinante de PVA (PVA(r)), un péptido N-terminal del virus TaLMV(p) y la proteína recombinante de una porción de cápside de TaLMV(r) (Gallo et al., 2011) y PVY(p). Tal como se esperaba, los resultados de la prueba indicaron que el anticuerpo anti-PVY(p) solo reconoce PVY(p) (figura 2B). De esta forma, ambas pruebas sugieren que los anticuerpos generados son específicos para el genotipo III del PVY y por tanto pueden ser utilizados como elemento diagnóstico para evaluar la presencia de este componente etiológico en cultivos de papa y tomate de árbol de Colombia y otros países.
Para conocer la sensibilidad de los anticuerpos en pruebas de ELISA y dot-blot se determinó la concentración mínima de péptido detectable. Mediante diluciones seriadas del péptido se encontró que éste puede ser detectado por los anticuerpos específicos anti-PVY(p) a concentraciones inferiores a 0.1 nM (figura 2C). Asumiendo que un Potyvirus está constituido por aproximadamente 2000 capsómeros (Urcuqui-Inchima, 2001), se estima que mediante pruebas de ELISA se pueden detectar concentraciones de hasta 10000 partículas virales de PVY por microlitro de muestra. En el caso de detección en membranas mediante dot-blot, el límite de detección correspondió a una concentración de 1 mM (figura 2D). Lo anterior equivale a una sensibilidad 10000 veces menor respecto a ELISA y un título viral aproximado de 108 de partículas virales por microlitro. Estos rangos de sensibilidad son muy similares a los obtenidos previamente para la detección del TaLMV (Gallo et al., 2011).
La sensibilidad en la detección de virus utilizando membranas puede ser mejorada significativamente mediante la utilización de anticuerpos marcados con oro coloidal en pruebas de flujo lateral. Este tipo de dispositivos tienen popularidad creciente al permitir el diagnóstico de virus por personal no calificado. Por esta razón, se construyó un prototipo de prueba de flujo lateral en formato competitivo. En estos dispositivos se fija el antígeno en una zona de prueba y un anti-anticuerpo específico en la zona de control (figura 3A). La prueba se considera positiva si el anticuerpo marcado en contacto con la muestra no es capaz de fijarse a la zona de prueba generando solamente una señal en la zona de control. La presencia de color tanto en las zonas de prueba como de control, indica que la prueba es negativa. Utilizando diferentes concentraciones de péptido PVY(p) se encontró que la prueba diseñada puede detectar la presencia de antígeno en la muestra a concentraciones superiores a 20 nM (figura 3B). El dispositivo de flujo lateral fue probado en una planta sana de papa obtenida mediante cultivo in vitro certificada por su sanidad viral y en dos plantas evaluadas como positivas para PVY mediante la prueba de ELISA. A pesar de que los pigmentos vegetales dificultaron ligeramente la lectura, fue posible discriminar las plantas infectadas de las libres de virus, sugiriendo la viabilidad de construir localmente dispositivos portátiles de detección de PVY utilizando anticuerpos como los aquí desarrollados (figura 3C).
Prueba piloto de detección
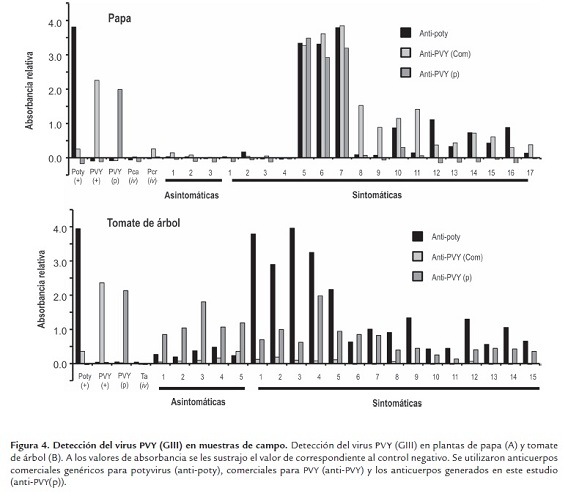
Detección de PVY (GIII) en plantas de papa. La utilidad de los anticuerpos generados fue evaluada en veinte plantas colectadas en diferentes cultivos de papa y contrastada con la detección con anticuerpos comerciales anti-poty y anti-PVY (figura 4A). El análisis de correlación utilizando el coeficiente de correlación de Pearson arrojó un valor de 0.79 entre la detección de Potyvirus con los anticuerpos genéricos y la prueba que utiliza anticuerpos comerciales para detectar diferentes serotipos de PVY. Lo anterior indica que en las muestras de papa obtenidas, existe una alta probabilidad de que PVY sea un componente principal de las infecciones causadas por Potyvirus en dichas plantas. En este sentido, Gil (2010), evaluó los niveles de incidencia de Potyvirus en cuatro departamentos cultivadores de papa de Colombia, detectando con pruebas de ELISA, la presencia dePotyvirus en el 72% de las muestras y encontrando mediante la utilización de RT-PCR en tiempo real, a partir de una submuestra de 40 de dichos materiales, que el 100% estaban infectadas con PVY.
Por otra parte, la correlación entre detección genérica de Potyvirus y de PVY (GIII) mediante los anticuerpos anti-PVY(p), arrojó un valor de 0.27; lo cual permitió plantear que dicho genotipo no es el que predomina en las muestras de papa obtenidas en el estudio. Este resultado es más claro aún si se tiene en cuenta la baja correlación (0.15) encontrada cuando se comparó la detección de los serotipos PVY-O,C y PVY-N lograda con los anticuerpos comerciales de PVY y aquellos desarrollados en esta investigación para PVY (GIII). De esta forma, tanto los anticuerpos genéricos para Potyvirus como los comerciales para PVY detectaron dichos virus en la mayoría de las plantas de papa sintomáticas (15 de 20), mientras que los anticuerpos anti-PVY(p) solo detectaron la presencia de PVY (GIII) en seis de las plantas (5-8, 10 y 11). Esta situación es un indicativo claro de la ocurrencia de coinfecciones de diferentes variantes de PVY y de otros Potyvirus en las muestras de papa analizadas. Hallazgos similares han sido reportados en múltiples estudios desarrollados en cultivos de diferentes países a partir de análisis tanto serológicos como moleculares. Así por ejemplo, Kogovsek et al. (2008) diseñaron una prueba de RT-PCR en tiempo real que permitió discriminar la presencia de diferentes genotipos de PVY en infecciones mixtas, encontrando la predominancia de cepas de la raza PVYNTN, aunque en algunas muestras se podían también encontrar aislamientos de PVYN y PVYO. Ounouna et al (2002), al evaluar la utilidad de anticuerpos monoclonales para la detección de variantes de PVY que afectan cultivos de Capsicum spp. en Túnez, determinaron la presencia de cepas del serotipo PVY-O,C y de un serotipo que no había sido identificado previamente en esta especie viral. Similarmente, Karasev et al. (2010), al realizar un estudio de varios años que pretendía evaluar los niveles de incidencia y diversidad de PVY en tubérculo semilla de papa distribuida en EEUU, encontraron además de las cepas clásicas de PVYN, PVYNTN, PVYN:O y PVYO, la presencia de la variante PVYO-O5, que siendo del serotipo PVY-O era detectada con anticuerpos monoclonales IF5, ampliamente utilizados para la detección del serotipo PVY-N por la organización norteamericana de protección de plantas (NAPPO).
Detección de PVY (GIII) en plantas de tomate de árbol. Para el caso de las muestras de tomate de árbol, todas las plantas evaluadas fueron positivas para la presencia de Potyvirus utilizando la prueba de ELISA con anticuerpos genéricos, incluso las cinco plantas asintomáticas. Sin embargo, los niveles de absorbancia obtenidos con los anticuerpos genéricos fueron claramente superiores para las plantas sintomáticas (figura 4B). Por otra parte, los anticuerpos comerciales para PVY detectaron la presencia de este virus en todas las plantas asintomáticas y en siete de las sintomáticas (1-5, 8 y 12). El coeficiente de correlación de Pearson entre ambas pruebas fue de 0.09, lo cual indica que existía una baja probabilidad de encontrar las cepas tradicionales de PVY detectadas por el kit comercial en las muestras de plantas de tomate de árbol estudiadas. En todos los casos que resultaron positivos, los títulos virales fueron muy cercanos al límite de detección de la prueba. Estos resultados concuerdan con los hallazgos de Ayala et al. (2010) quienes al realizar un análisis de la presencia de Potyvirus en siete zonas cultivadoras de tomate de árbol en Colombia, encontraron muy altos niveles de incidencia de estos virus con un promedio de 80%, estando también presente el TaLMV, una nueva especie de Potyvirus propuesta por estos autores. En dicho trabajo se indica además que los valores de absorbancia obtenidos con los anticuerpos comerciales para PVY fueron muy bajos en comparación con el control positivo y con los resultados de las pruebas con anticuerpos generales para Potyvirus, lo cual coincide plenamente con lo encontrado en el presente estudio. Cuando se utilizaron los anticuerpos propios para la detección de PVY (GIII), se encontró la presencia de esta variante en todas las plantas de tomate de árbol evaluadas, lo que se reflejó por un índice de correlación de 1.0 con respecto a la detección con anticuerpos genéricos para Potyvirus, reafirmándose así el hecho que existen genotipos de PVY con diferencias en sus perfiles serológicos y que por ende requieren de la generación de pruebas de diagnóstico específicas.
Confirmación mediante RT-PCR de PVY (GIII)
La utilización de la técnica RT-PCR con los cebadores PVYCPF_BamHI y PVYCPR_HindIII diseñados por nuestro grupo, permitió obtener amplicones del tamaño esperado de 850 pb (figura 5A). La secuenciación posterior confirmó la identidad de dichos amplicones como correspondientes al gen de la cápside de PVY, encontrándose los tres genotipos (GI, GII y GIII) reportados por Henao et al. (2011) y Jaramillo et al. (2011) en las muestras de papa y tomate de árbol utilizadas para la estandarización de las pruebas serológicas y en la prueba piloto. En lafigura 5B se presenta una matriz de distancia en donde se aprecia que la diferencia en las secuencias entre los genotipos I y II de PVY en Colombia corresponde al 5%, mientras que éstos presentan una distancia del 9% con respecto al PVY (GIII). PVY (GI y GII) presenta mayores niveles de identidad (>95%) para la región secuenciada con respecto a las cepas representativas de la raza PVYNTN, mientras que PVY (GIII) aparentemente está más relacionado con un genotipo representativo de la raza PVYN, aunque los niveles de identidad con éste fueron tan sólo del 93%. De gran interés será la realización de análisis filogenéticos que incluyan un alto número de cepas representativas de los tres genotipos de PVY encontrados en Colombia, así como su caracterización patogénica en papa y tabaco con el fin de identificar sus razas bajo el esquema tradicional aplicado para esta especie viral (Singh et al., 2008).
El éxito en el manejo de las enfermedades virales recae en dos aspectos fundamentales: a corto plazo, en la combinación de buenas prácticas culturales y del uso de material de siembra libre de virus y a largo plazo, en la obtención de materiales vegetales resistentes. Ambas estrategias requieren a su vez de la disponibilidad de herramientas diagnósticas de detección temprana que apoyen los programas de mejoramiento genético y de vigilancia cuarentenaria. Para el desarrollo de dichas herramientas, se requiere del conocimiento apropiado de la biología de los virus y muy particularmente de sus niveles de variación. La mayoría de los métodos de detección de virus de plantas comercialmente disponibles en el mundo, han sido diseñados a partir de variantes de importancia económica global, por lo que el desarrollo de métodos de diagnóstico para genotipos virales regionales debe ser abordado a nivel local. En este trabajo se ha demostrado la viabilidad de utilizar péptidos sintéticos para la producción de anticuerpos específicos útiles en la detección de variantes genotípicas de PVY. Este procedimiento difiere significativamente de las metodologías clásicas que requieren de la purificación del virus a partir de plantas infectadas los cuales son bastante laboriosos, costosos y no garantizan la producción de anticuerpos de alta especificidad. Se espera que los métodos de detección aquí desarrollados sean implementados por los organismos de sanidad vegetal, los gremios de productores y los asistentes técnicos, de manera que se apoyen los programas de certificación de material de siembra de papa y tomate de árbol en Colombia y en otros países donde se presente el PVY (GIII).
Conclusiones
Se generaron anticuerpos policlonales para la detección específica del PVY (GIII), uno de los genotipos que afecta los cultivos de tomate de árbol y papa en Colombia, desarrollándose tres esquemas de diagnóstico serológico: ELISA, dot-blot y flujo lateral.
A partir de una prueba piloto de detección de PVY, se detectó el PVY (GIII) en las 20 plantas de tomate de árbol evaluadas, independientemente de que presentaran síntomas visuales de infecciones virales. Así mismo, ésta variante fue detectada en seis de las plantas de papa, lo que sugiere la posible patogenicidad cruzada de dicho virus entre ambos hospedantes.
Agradecimientos
Esta investigación fue financiada por el Ministerio de Agricultura y Desarrollo Rural de Colombia (proyecto 090-2007S4527-87-08) y la Dirección de Investigaciones de la Universidad Nacional de Colombia - Sede Medellín.
Referencias bibliográficas
1 Altschul, S.F., Gish, W.; Miller, W., Myers, E.W., Lipman, D.J. 1990. Basic local alignment search tool. Journal of Molecular Biology. 215: 403-410.
2 Ayala, M., González, P., Gutiérrez, P.A., Cotes, J.M., Marín, M. 2010. Caracterización serológica y molecular dePotyvirus asociados a la virosis del tomate de árbol en Antioquia (Colombia). Acta Biológica Colombiana. 5: 143-162.
3 Blanco-Urgoiti, B., Sánchez, P., Pérez de San Román, C., Dopazo, J., Ponz, F. 1998. Potato virus Y group C isolates are a homogeneous pathotype but two different genetic strains, Journal of General Virology. 79: 2037-2042.
4 Chikh, M., Maoka, T., Natsuaki, K.T. 2007. A point mutation changes the serotype of a Potato virus Y isolate; genomic determination of the serotype of PVY strains. Virus Genes. 35: 359-367.
5 Dawei, L., Wei, S., Yang, H., Li, Y., Deng, A. 2009. A sensitive inmunochromatographic assay using colloidal gold-antibody probe for rapid detection of pharmaceutical indomethacin in water samples. Biosensors and Bioelectronics. 24: 2277-2280.
6 Ellis, P., Stace-Smith, R., de Villiers, G. 1977. Identification and geographic distribution of serotypes of Potato virus Y. Plant Disease. 81: 481-484.
7 Ellis P., Stace-Smith, R., Bowler, G., Mackenzie D.J. 1996. Production of monoclonal antibodies for detection and identification of strains of potato virus Y. Canadian Journal of Plant Pathology. 18:64-70.
8 Fauquet, C.M., Mayo, M.A., Maniloff, J., Desselberger, U., Ball, L.A. 2005. Virus taxonomy. Eighth Report of the International Committee on Taxonomy of Viruses. San Diego (EEUU): Elsevier Academic Press. 1259 p.
9 Shukla, D.D., Ward, C.W., Brunt, A.A. 1994. The Potyviridae. Wallingford (UK): CABI. 448 p.
10 Gallo, Y., Marín, M., Gutiérrez, P.A. 2011. Obtención de anticuerpos específicos para la detección del Tamarillo leaf malformation virus (TaLMV) en tomate de árbol". Acta Biológica Colombiana. 16:135-148.
11 Gil, J.F., Cotes, J.M., Marín, M. 2011. Incidencia de Potyvirus y caracterización molecular de PVY en regiones productoras de papa (Solanum tuberosum L.) de Colombia. Revista Colombiana de Biotecnología. 13: 85-93.
12 Hall, T.A. 1999. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucleic Acids Symposium Series. 41:9 5-98.
13 Henao, E., Gil, J.F., Marín, M. 2011. Identificación genotípica de variantes de los Potyvirus PVY y TaLMV en Colombia. Memorias del II Congreso Argentino de Fitopatología, Mar del Plata, Argentina. 1-3 de Junio de 2011.
14 Hopp, T.P., Woods R.K. 1981. Predictions of protein antigenic determinants from amino acid sequences.Proceedings of the National Academy of Sciences USA (PNAS). 78:3824-3828.
15 Hu, X., Nie, X., He, C., Xiong, X. 2011. Differential pathogenicity of two different recombinant PVYNTN isolates in Physalis floridana is likely determined by the coat protein gene. Virology Journal. 8: 1-5.
16 Hu, X.; He, C., Xiao, Y., Xiong, X., Nie, X. 2009. Molecular characterization and detection of recombinant isolates of Potato virus Y from China. Archives of Virology. 154: 1303-1312.
17 Jaramillo, M., Gutiérrez, P.A., Lagos, L.E., Cotes, J.M., Marin, M. 2011. Detection of a complex of viruses in tamarillo (Solanum betaceum) orchards in the Andean region of Colombia. Tropical Plant Pathology. 36: 150-159.
18 Jones, R.A.C. 1990. Strain group specific and virus specific hypersensitive reactions to infection withPotyviruses in potato cultivars. Annals of Applied Biology. 117: 93-105.
19 Jordan, R., Hammond, J. 1991. Comparison and differentiation of Potyvirus isolates and identification of strain-, virus-, subgroup-specific and Potyvirus group-common epitopes using monoclonal antibodies. Journal of General Virology. 72: 25-36.
20 Kahn, R.P., Monroe, R.O. 1963. Detection of the tobacco veinal necrosis strain of the Potato virus Y inSolanum cardenasii and S. andigenum introduced into the United States. Phytopathology. 53: 1356-1359.
21 Karasev, A.V., Nikolaeva, O.V., Hu, X., Sielaff, Z., Whitworth, J., Lorenzen, JH., Gray, S.M.. 2010. Potato virusY: A Case Study of PVYN Misidentification. American Journal of Potato Research. 87: 1-9.
22 Kogovsek, P., Gow, L., Pompe-Novak, M., Gruden, K., Foster, G.D., Boonham, N., Ravnikar, M. 2008. Single-step RT real-time PCR for sensitive detection and discrimination of Potato virus Y isolates. Journal of Virological Methods. 149: 1-11.
23 Matthews, R.E.F. 1993. Diagnosis of plant virus diseases. Boca Ratón: CRC Press. 374p.
24 McDonald, G., Kristjansson, G.T., Singh, R.P., Ellis, P.J., McNab, W. 1994. Consecutive ELISA screening with monoclonal antibodies to detect potato virus YN. American Potato Journal. 71: 175-83.
25 Miczynski, K.A. 1963. Studies on the Potato virus Y strains, their properties and occurrence in three potato varieties. Acta Biologica Cracoviensia. 6: 55-73.
26 Nie, X., Singh, R.P. 2003. Evolution of North American PVYNTN strain Tu 660 from local PVYN by mutation rather than recombination. Virus Genes. 26: 39-47.
27 Ounouna, H., Kerlan, C., Lafaye, P., Loukili, M.J., ElGaaied, A. 2002. Production of monoclonal antibodies against synthetic peptides of the N-terminal region of Potato virus Y coat protein and their use in PVY strain differentiation. Plant Pathology. 51: 487-494.
28 Riechmann, J.L., Laín, S., García, J.A. 1992. Highlights and prospects of Potyvirus molecular biology. Journal of General Virology. 73: 1-16.
29 Romero, A., Blanco-Urgoiti, B., Soto, M.J., Fereres, A., Ponz, F. 2001. Characterization of typical pepper-isolates of PVY reveals multiple pathotypes within a single genetic strain. Virus Research. 79: 71-80.
30 Shukla, D.D., Ward, C.W., Brunt, A.A. 1994. The Potyviridae. Wallingford (UK): CABI. 448 p.
31 Singh, R.P., Valkonen, J.P.T., Gray, S.M.,; Boonham, N., Jones, R.A.C.; Kerlan, C., Schubert, J. 2008. Brief review: The naming of Potato virus Y strains infecting potato. Archives of Virology. 153: 1-13.
32 Tajima F., Nei, M. 1984. Estimation of evolutionary distance between nucleotide sequences. Molecular Biology and Evolution. 1: 269-285.
33 Urcuqui-Inchima, S., Haenni, A.L., Bernardi, F. 2001. Potyvirus proteins: a wealth of functions. Virus Research. 74:157-175.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2012 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).