Establecimiento de un sistema diagnóstico para la detección de Ralstonia solanacearum y diferenciación genética utilizando marcadores moleculares RAPD
Establishing a diagnostic system for detecting Ralstonia solanacearum and genetic differentiation using RAPD molecular markers
Palabras clave:
marchitez bacteriana, moko, PCR-16S ADNr, ELISA-NCM, PCR-RAPD, bacterial withered, PCR-16S rADN (es)|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31________________________________________________________
|
|
|
|
Establecimiento de un sistema diagnóstico para la detección
de Ralstonia solanacearum y diferenciación genética
utilizando marcadores moleculares RAPD
Establishing a diagnostic system for detecting Ralstonia solanacearum and genetic differentiation using RAPD
molecular markers
Edisson Chavarro Mesa*, Jorge Evelio Ángel Díaz**
RESUMEN
Con el propósito de identificar de manera rápida y precisa el agente causal de la marchitez bacteriana de la papa (Ralstonia solanacearum) en tubérculos asintomáticos, se ha desarrollado una prueba diagnóstica basada en la reacción en cadena de la polimerasa (PCR) utilizando iniciadores específicos para amplificar una región 16S rDNA de 292 pb. Para ello se aisló la bacteria a partir de tubérculos de papa y frutos de plátano, empleando técnicas de cultivo, prueba inmunológica y molecular ELISA-NCM y PCR, respectivamente. En tubérculos asintomáticos la PCR detectó la presencia de R. solanacearum en contraste con la ELISA-NCM que no detectó al patógeno. El análisis de polimorfismos de amplificación al azar de ADN (RAPD) permitió diferenciar y agrupar las procedencias de R. solanacearum por regiones geográficas y razas bacterianas, sugiriendo que existen diferencias entre las colectas de acuerdo con su lugar de origen, indicando alta variabilidad genética. Los resultados mostraron que la PCR es una prueba sensible y específica para la detección de R. solanacearum y por lo tanto puede ser implementada como un método de control del patógeno en programas de producción de semilla y certificación de áreas libres de la enfermedad. Según el origen geográfico de las muestras, el patógeno mostró ser genéticamente heterogéneo, dificultando su control en regiones colombianas con problemas fitosanitarios con R. solanacearum en cultivo de papa.
Palabras clave: marchitez bacteriana, moko, PCR-16S ADNr, ELISA-NCM, PCR-RAPD.
ABSTRACT
A polymerase chain reaction-based diagnostic test (PCR) has been developed for amplifying a región and obtaining a 292 bp product by using specific 16S rDNA primers for the rapid and precise identification of the causative agent (Ralstonia solanacearum) of bacterial withering of potato in asymptomatic tubers. The bacteria was isolated from potato tubers and banana fruit using culturing techniques and immunological and molecular ELISA-NCM and PCR tests, respectively. PCR detected the presence of R. solanacearum on asymptomatic tubers by contrast with ELISA-NCM which did not detect this pathogen. Analysing random amplified polymorphic DNA (RAPD) led to differentiating and grouping R. solanacearum by geographical región and bacterial strain, suggesting that differences exist amongst existing collections according to their place of origin, presenting high genetic variability. The results showed that PCR is a sensitive and specific test for detecting R. solanacearum and can therefore be implemented as a method for controlling this pathogen in seed production and certification programmes in áreas free of the disease. The pathogen has been shown to be genetically heterogeneous according to the samples' geographical área thereby hampering control in áreas of Colombia experiencing phytosanitary problems with R. solanacearum in potato crops
Key words: bacterial withered, moko, PCR-16S rADN, ELISA-NCM, PCR-RAPD. Recibido: agosto 10 de 2004. Aceptado: noviembre 20 de 2005
|
|
* Biólogo. Investigador. Laboratorio Nacional de Análisis Molecular, Instituto Colombiano Agropecuario (ICA). Correo electrónico: bioedicha@gmail.com
** Ph.D. Coordinador. Laboratorio Nacional de Análisis Molecular, Instituto Colombiano Agropecuario (ICA). Correo electrónico: jorgecol@gmail.com
|
|
|
|
14
|
| ________________________________ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum | |
|
|
|
|
INTRODUCCIÓN
El cultivo de la papa (Solanum tuberosum L.) tiene gran impacto dentro de los sistemas agrícolas en los cuales se desarrolla su producción, principalmente en la región andina, debido a que posee condiciones agroecológicas propicias para su desarrollo además de su potencial alimenticio, que en términos de calorías es elevado; siendo una alternativa para contribuir a solucionar problemas de escasez de alimento a corto plazo con mayor ventaja competitiva sobre otros productos tradicionales. La bacteria ataca más de 44 familias de plantas en regiones tropicales y subtropicales, y 200 especies de plantas dentro de las cuales las de mayor importancia económica incluyen: tabaco, musáceas, tomate, papa, ají, berenjena, maní y varias plantas ornamentales (Hayward, 1991). Por lo menos tres razas de Ralstonia solanacearum son las que producen enfermedades en varios hospedantes; una de ellas ataca a todas las solanáceas y a muchos otros cultivos que no pertenecen a este grupo de plantas, así como algunos plátanos; otras sólo atacan a plantas de la familia del plátano y una tercera ataca a la papa y en ocasiones al tabaco (Agrios, 2001). En el plátano y banano causa moko, y en papa la enfermedad se conoce como dormidera, pudrición parda o marchitez bacteriana (Pérez, 1993). Debido a su difícil identificación se recurre a técnicas que presentan mayor especificidad, las técnicas de diagnóstico molecular como la reacción en cadena de la polimerasa (PCR) permiten la detección de la bacteria mediante el uso de iniciadores específicos y confirman los resultados dados por ensayos
|
inmunoenzimáticos. Entre los diferentes marcadores moleculares conocidos, los RAPD (ADN polimórfico amplificado al azar) son útiles para el desarrollo de huellas digitales de ADN cuando se requiere identificar variedades dentro de una especie, determinar parentesco de un material y caracterizar genéticamente el mismo. Esta metodología RAPD es una técnica molecular basada en la amplificación enzimática (PCR) de fragmentos de ADN definidos en el genoma, usando como œnico iniciador (primer) un decanucleótido de secuencia arbitraria.
MATERIALES Y MÉTODOS
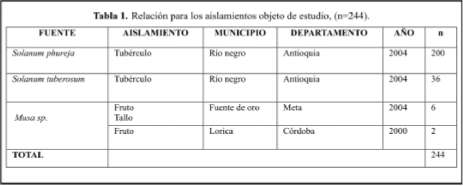
Población de estudio. El análisis del material biológico se efectuó a partir de tejido vegetal con síntomas característicos de la enfermedad, recolectado directamente en campo y enviado al Laboratorio Nacional de Análisis Molecular-ICA (LANAM). Se estudiaron 244 aislamientos provenientes de cultivares de papa (Solanum phureja y Solanum tuberosum) y plátano (Musa sp), los cuales provenían de regiones colombianas que presentan habitualmente focos de infección bacteriana y donde es importante realizar el análisis genético y diagnóstico de la bacteria. La fuente, lugar de origen, fecha y tipo de aislamiento se describe en la tabla 1.
Protocolo para la evaluación de semilla de papa. El tamaño de la muestra fue de 200 tubérculos, tomados en la cosecha o en el almacenamiento para analizarse en 8 submuestras de 25 tubércu-
|
|
|
|
 |
|
|
|
|
|
15
|
|
|
|
|
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31_______________________________________________________________
|
|||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||
|
los. Los fragmentos del anillo vascular tomados de 25 tubérculos de S. phureja previamente desinfectados con hipoclorito al 1 % se maceran en una bolsa individual después de añadir el volumen de la solución tampón de extracción, buffer citrato al 1%. Para cada submuestra se mezcla 500 mL del extracto del tubérculo con el mismo volumen del caldo SMSA (Priou, 2001) e incuban durante 48 h a 30 "C con agitación constante. Se colocan sobre la membrana por duplicado por cada extracto enriquecido para la prueba serológica (Priou, 2001), el ensayo de PCR se le realizó a cada extracto enriquecido.
ELISA-NCM® (prueba inmunoenzimática en membrana de nitrocelulosa). Los extractos vegetales fueron sembrados en medio SMSA para la realización del ensayo inmunoenzimático con el kit suministrado por el Centro Internacional de la Papa (CIP) ELISA-NCM®, siguiendo las instrucciones del fabricante. Como controles negativos se utilizaron: Pseudomonas fluorecens, Erwinia caratovora, Pseudomonas chicoríi y Xanthomonas albilineans.
Extracción de ADN bacteriano. Para la obtención del ADN bacteriano se utilizaron tres protocolos de extracción. La metodología propuesta por Boucher et al. (1985), el método descrito por Seal et al. (1992) para realizar PCR a partir de cultivos líquidos sometidos a calentamiento y la metodología recomendada por Chen and Kuo,
|
(1993) para una extracción rápida de ADN de bacterias Gram negativas. Los 3 protocolos fueron probados por triplicado y en 4 ocasiones diferentes la concentración de ADN fue cuantificada utilizando un Biophotometer Eppendorf 6131; los datos fueron tomados en el mismo momento de terminado el protocolo de extracción. Después de consignados los datos de concentración de ADN por protocolo se obtuvieron los siguientes datos utilizando el software Microsoft Excel 2000: concentración promedio por aplicación, concentración promedio total por protocolo, valores de concentración máxima y mínima, varianza y desviación estándar. El ADN extraído fue corrido en gel de agarosa al 0.8% preteñido con bromuro de etidio al 0.5 u.g/mL y fotografiado bajo luz ultravioleta en el analizador de imágenes Syn Gene®.
Reacción en cadena de la polimerasa (PCR). La prueba de PCR se realizó con las siguientes condiciones para cada reacción y la posterior amplificación en un termociclador MJ Research PTC-200, las concentraciones finales de los compuestos para un volumen de reacción de 25 u.L destinados a la amplificación de ADN de R. solanacearum, a partir de caldo enriquecido SMSA, fueron: 10 mM Tris-HCl (pH 8.5), 50 mM KCl, 1.5 MgCI2, 0.2 mM dNTP, 0.5 mM de cada oligonucleó-tido, 1.25 unidades de Taq (Promega®) y 1 u.L de ADN molde. El programa de termociclado utilizado
|
||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||
|
Tabla 2. Ciclos de tiempos y temperaturas utilizadas en la prueba PCR
|
|||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||
|
|||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||
|
16
|
|||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||
|
________ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
|
|
|
|
 |
|
|
|
|
|
se ilustra en la tabla 2. Los oligonucleótidos usados para la amplificación específica de un fragmento de 292 pares de bases fueron OLI1 y Y2, cuyas regiones blanco son secuencias específicas 16S ADNr (Seal et al., 1999). Para visualizar los productos de amplificación de la PCR se realizaron electroforesis en gel de agarosa al 1.5% preteñidos con bromuro de etidio al 0.5 u.g/mL, utilizando un marcador de peso molecular de 100 pb (PCR Marker-Promega™) y fotografiados bajo luz ultravioleta en el analizador de imágenes Syn Gene®.
Especificidad y sensibilidad de la prueba.
La capacidad diagnóstica de un método viene determinada por el cálculo de sensibilidad y especificidad de ese determinado método, frente a la técnica considerada de referencia. Se determinó la especificidad, sensibilidad y valor predictivo de la prueba mediante el uso de tablas de contingencia (Saunders, 1999).
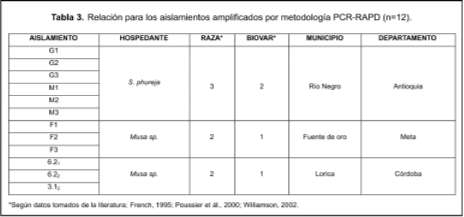
Condiciones para la amplificación PCR-RAPD. EL ADN genómico de 12 aislamientos de R solanacearum, tabla 3, previamente confirmados y que se encontraban disponibles en el banco de cepas del Laboratorio Nacional de Análisis Molecular-ICA, se utilizó para generar perfiles de RAPD con 25 decanucleótidos cuya secuencia corresponde a los iniciadores establecidos por Operon Technologies Inc (www.operon.com), de los cuales se tomaron 5 iniciadores reproducibles (OPA 13,
|
OPC 6, OPC 8, OPC 11 y OPD 7), tabla 4, para cada ensayo planteado por duplicado de reproducibilidad de la prueba de PCR-RAPD. El criterio de selección de los 25 iniciadores corresponde a los resultados obtenidos por Thwaites et al. (1999), quienes probaron un nœmero total de 100 iniciadores RAPD y encontraron 20 iniciadores reproducibles, dentro de los cuales se hallaban 4 que generaron un buen patrón de bandas RAPD en el presente estudio.
Las condiciones fueron optimizadas a partir de las concentraciones descritas por Thwaites et al. (1999) para permitir alta reproducibilidad de los fragmentos generados por la metodología. Los parámetros de reacción tomados en cuenta para la optimización fueron principalmente la concentración de MgCI2, la concentración de oligonucleótido, concentración de templado y la concentración de Taq polimerasa. Las concentraciones finales de los compuestos para un volumen de reacción de 25 u.L destinados a la amplificación de ADN de R. solanacearum por metodología de PCR-RAPD fueron: Buffer (10 mM Tris-HCl (pH8.5), 50 mM KCl, 0,1 % de tritón 100X), 3.0 mM MgCI2 , 0.2 mM dNTP, 1 mM de cada iniciador RAPD, 2.0 unidades de Taq (Promega®), 0.04% Seroalbœmina bovina y 1 u.L de ADN molde, 50 hg/mL. Las amplificaciones fueron realizadas en un termociclador MJ Research PTC-200, luego del análisis de las pruebas de
|
|
|
|
|
17
|
|
|
|
|
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31__________________________________________________________
|
|||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||
|
Tabla 4. Secuencia de los oligonucleótidos utilizados en las pruebas de
PCR-RAPD
|
|||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||
|
optimización y reproducibilidad. Los ciclos de tiempos y temperaturas utilizados están descritos en la tabla 5 (Williams et al., 1990; Raba, 2003).
Los fragmentos de ADN amplificados se hicieron migrar en un gel de agarosa al 1.5% (p/v), mediante electroforesis (por 3 horas, con una corriente de 100V en buffer TBE) (Maniatis et al., 1982). Lavisualización se realizó mediante tinción del ADN con bromuro de etidio (0.5 u.g/mL) en un digitalizador de imágenes Syn Geneá utilizando el programa GeneSnapá. Para el cálculo aproximado de los pesos moleculares de las bandas obtenidas se utilizó el programa GeneToolsá, el
|
cual toma como patrón de comparación el marcador de peso molecular (1 kb DNA Ladder Gibco ™) corrido en el gel. El criterio utilizado para identificar una banda como polimórfica fue su presencia o ausencia en forma consistente en dos amplificaciones independientes. Estos resultados permitieron construir una matriz binaria que combinó todos los polimorfismos detectados en los aislamientos de R. solanacearum en estudio, con los 5 iniciadores anteriormente descritos. Esta matriz fue analizada utilizando el software NTSYS (Thwaites et al., 1999). Para cada par de aislamientos se determinó el grado de similitud utilizando el coeficiente de similaridad de Dice
|
||||||||||||||||||||||||||||||||||||||||
|
|
|||||||||||||||||||||||||||||||||||||||||
|
18
|
|||||||||||||||||||||||||||||||||||||||||
|
_______ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
|
|
|
|
|
(Sneath y Sokal, 1973), y luego construir un dendograma mediante el algoritmo UPGMA (Unweighted pair Group Method with Arithmatic Mean). Para evaluar la consistencia de los agrupamientos representados en el dendograma se determinó el valor de la correlación entre la matriz de los valores cofenéticos de los datos de similitud y la misma matriz de similitud. Adicional-mente, los datos RAPD fueron sometidos a un análisis de coordenadas principales (PCA). Estos dos œltimos análisis se realizaron utilizando las subrutinas adecuadas del programa NTSYS.
RESULTADOS
Extracción de ADN bacteriano. Los dos mejores protocolos evaluados son los citados por Seal et al. (1992) y Chen and Kuo (1993) con promedios totales de 108.4 mg/ul, y 191.7 mg/u.L respectivamente, con el protocolo de Boucher et ál. (1985) se obtuvo la menor concentración promedio de ADN 91.8 mg/ul. Los valores de varianza y desviación estándar entre metodologías que presentaron mayor valor son los de las metodologías de Boucher et al. (1985) y Seal et al., (1992); estas medidas de dispersión sugieren poca reproducibilidad y amplios rangos en las concentraciones de ADN obtenidas. Los valores de varianza y desviación estándar sugieren que el protocolo de Chen and Kuo (1993) es el mejor de
|
los tres protocolos debido a que posee un valor de 10.3 de desviación estándar contra valores de 27.8 y 30.4 de los protocolos de Boucher et al. (1985) y Seal et al. (1992), respectivamente, esto indica la homogeneidad de las concentraciones del ADN obtenido con la metodología descrita por Chen and Kuo (1993), la cual debido a esto se considera en este trabajo la más reproducible y confiable (tabla 6).
Evaluación por hectárea de semilla de papa de Solanum phureja
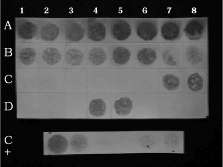
Prueba NCM-ELISA. Las ocho submuestras de S. phureja analizadas por duplicado, tal como lo recomienda Priou (2001) para el análisis de lotes de semilla de papa resultaron positivas, evidenciándose la coloración púrpura (figura 1) para las submuestras y una concentración bacteriana de 108 a 107 bact/mL, confirmando el grave estado de los tubérculos.
Reactividad cruzada. Ésta ocurre cuando la prueba NCM-ELISA arroja un falso positivo si esta misma es realizada simultáneamente con bacterias diferentes a R. solanacearum. En el ensayo se probaron 4 bacterias de diferente especie a R. solanacearum, encontrándose reactividad cruzada con Pseudomonas chicorii (figura 1, C7-8). Puede ocurrir que se comparta la misma conformación
|
|
|
|
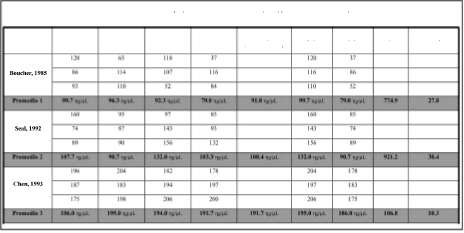 Tabla 6. Datos estadísticos para la estandarización de ADN bacteriano
Aplicación 1 Aplicación 2 Aplicación 3 Aplicación 4 Promedio total Máximo Minimo Varianza Desv. Estándar
|
|
|
|
|
|
19
|
|
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31__________________________________________________
|
||
|
|
||
 |
proteica de los epitopes de la bacteria, que son reconocidos por los anticuerpos específicos para R. solanacearum. Con los datos dados por el NCM-ELISA se realizó el análisis de tablas de contingencia para encontrar la sensibilidad y especificidad de la prueba.
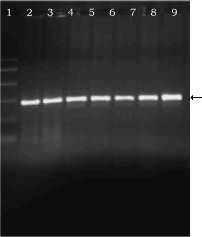
Reacción en cadena de la polimerasa (PCR). A las submuestras de S. phureja se les realizó la prueba de PCR con los iniciadores específicos OLI1 y Y2 evidenciándose producto de amplificación de 292 pb. Se logró detectar la presencia de la bacteria en 6 submuestras (figura 2). De esta forma se confirman los resultados dados por el ensayo inmunoenzimático. Respecto a la reactividad cruzada, la PCR no encontró producto de amplificación para la bacteria Pseudomonas chicorii.
Detección de R. solanacearum en tubérculo de Solanum tuberosum
Diagnóstico fitopatológico. De los 36 tubérculos analizados 21 de ellos, que corresponden al 59%, eran tubérculos asintomáticos, el 41% restante corresponde a 15 tubérculos sintomáticos. Es importante realizar las pruebas diagnósticas a los tubérculos asintomáticos para detectar la presencia de la bacteria en estos mismos.
|
|
|
Figura 1. Análisis NCM-ELISA de 8 submuestras obtenidas por hectárea de semilla de Solanum phureja. El orden de siembra en la membrana corresponde: pozos A-1 a A-8; submuestras 1 a la 8, cada submuestra conformada por 25 tubérculos de Solanum phureja y sembradas por duplicado sobre la membrana. Pozos C-1 a C-6, controles blancos de reacción. Pozos C-7 y C-8; Pseudomonas chicorii. Pozos D-1 a D-3, controles blancos de reacción. Pozos D-4 y D-5; cepa 6.2. Pozos D-6 a D-8, controles blancos de reacción. Centro inferior, tira control positivo de reacción; coloración púrpura: reacción positiva; ausencia de color: reacción negativa.
|
||
 |
292 pb
|
|
 |
||
|
Figura 3. Análisis NCM-ELISA de 36 tubérculos de Solanum tuberosum. El orden de siembra en la membrana corresponde a: pozos A-1 a E-5; aislamientos 1 al 36 a partir de tubérculos de Solanum tuberosum, respectivamente, sembrados individualmente. Pozos E-5, 6, 7 y 8; controles blancos. Centro inferior, tira control de reacción; coloración púrpura: reacción positiva; ausencia de color: reacción negativa.
|
||
|
Figura 2. Productos de amplificación. Los carriles 1 al 9 corresponden a las siguientes muestras: carril 1 marcador de 100 pb; carriles 2 al 7 submuestras 1, 2, 3, 4, 5 y 6 de tubérculos sintomáticos de S. phureja; carriles 8 y 9 control positivo y negativo, respectivamente.
|
||
|
|
||
|
20
|
||
|
|
||||||||||||||||||||||||||||||||||||
|
ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
|
Prueba NCM-ELISA. El 59% es decir 21 de los tubérculos fueron positivos a la prueba ELISA-NCM, el 41% restante corresponde a 15 tubérculos que son negativos al ensayo de ELISA-NCM, detectándose una concentración bacteriana de 108a 107 bact/mL (figura 3).
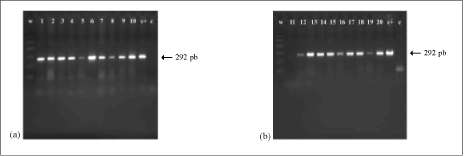
Reacción en Cadena de la Polimerasa (PCR). EL 97% equivalente a 35 tubérculos, dieron productos de PCR positivos con los iniciadores OLI1 yY2, observándose bandas de amplificación de 292 pb, para cada aislamiento, con estos resultados se detecta la presencia de R. solanacearum en tubérculos de S. tuberosum sintomáticos y asintomáticos. El 3% restante corresponde al aislamiento 11 que es negativo a la
|
prueba ELISA-NCM y al ensayo de PCR (figura 4 y 5).
Tubérculos sintomáticos y asintomáticos analizados por la prueba de PCR y la ELISA-NCM. La PCR detectó en un 98% de tubérculos asintomáticos de Solanum tuberosum en comparación con la prueba ELISA-NCM, que no detectó en su totalidad los tubérculos asintomáticos, 47% fue el porcentaje de tubérculos asintomáticos que logró detectar la prueba inmunoenzimática (figura 6). Para el caso de tubérculos sintomáticos, la prueba de PCR detectó la totalidad de los tubérculos que manifestaban la patología característica, la ELISA-NCM logró detectar el 73% de tubérculos sintomáticos (figura 6).
|
|||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
 |
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
|
Figura 4. Productos de amplificación de 292 pb con los oligonucleotidos OLI1 y Y2 para la detección molecular de R. solanacearum en tubérculo de S. tuberosum. w; marcador de 100 pb. (a) Aislamientos 1 al 10. (b) Aislamientos 11 al 20.
|
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
|
Figura 5. Productos de amplificación de 292 pb con los oligonucleotidos OLI1 y Y2 para la detección molecular de R. solanacearum en tubérculo de S. tuberosum. w; marcador de 100 pb. (a) aislamientos 21 al 30. (b) aislamientos 31 al 36.
|
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
|
21
|
||||||||||||||||||||||||||||||||||||
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31
|
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
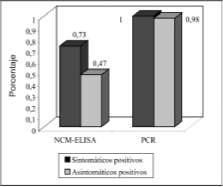 |
 Tabla 7. Aislamiento de ADN de microorganismos fitopátogenos distintos a R. solanacearum
|
|||||||||||||||||||||||||||||||||||
|
Figura 6. Porcentaje de tubérculos de Solanum tuberosum sintomáticos y asintomáticos que logra detectar las pruebas de PCR y NCM-ELISA.
|
||||||||||||||||||||||||||||||||||||
|
Análisis de especificidad y sensibilidad mediante el uso de tablas de contingencia. Los
resultados obtenidos en la prueba PCR se evaluaron como positivos y negativos de acuerdo con la presencia/ausencia de producto de amplificación. Se evaluó la reactividad cruzada con nueve aislamientos de ADN distintos al de R. solanacearum (tabla 7). Estos resultados fueron sometidos al análisis de tablas de contingencia 2x2 (Saunders D., 1999) para determinar sensibilidad y especificidad de la prueba, tomando como prueba estándar el análisis NCM-ELISA ofrecido por el Centro Internacional de la Papa (CIP) como kit diagnóstico de R. solanacearum en tubérculo de papa.
Para el análisis de contingencia se captó al azar una submuestra de 25 aislamientos compuestos de muestras positivas y negativas de los 244 aislamientos estudiados. Se obtuvo un total de 11 muestras positivas y 14 muestras negativas, de las cuales, en el análisis de contingencia, se estableció que 0 aislamientos de las 11 muestras positivas son falsos positivos, es decir, son valores negativos reales que la PCR reporta como positivos. El 21 % (3 muestras) de las 14 muestras negativas son falsos negativos o valores positivos reales que la PCR reporta como negativos. La sensibilidad de esta prueba fue del 78% y la especificidad del 100% (tabla 8).
|
||||||||||||||||||||||||||||||||||||
|
Tabla 8. Tabla de contingencia PCR vs. NCM-ELISA ELISA - NCM
|
||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
|
22
|
||||||||||||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||||||
|
______ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
|
|
|
|
|
Amplificación aleatoria de polimorfismos de ADN (PCR-RAPD). Para la evaluación de la diversidad genética de R. solanacearum se probaron 25 iniciadores, de los cuales se tomaron 5 iniciadores RAPD, teniendo como parámetro de selección la alta reproducibilidad que generó cada iniciador. En el análisis se tomaron bandas mayores de 200 pb (Lo Presti etál., 1998; López, 2004), ya que bandas de menor tamaño poseen baja reproducibilidad. Se logró amplificar un total de 394 bandas, de las cuales 380 fueron RAPD, es decir, bandas polimórficas, en un rango de tamaño de 418-4072 pb (tabla 9).
Frecuencia de variación molecular por genotipos. Se calculó contando el número total de bandas polimórficas (Marulanda et ál., 2002) encontradas por cada una de las tres regiones geográficas objeto de estudio, teniendo en cuenta la fuente de cada aislamiento que correspondía a S. phureja y Musa sp. La frecuencia de variación molecular por genotipos fue 0.34 para Río Negro, departamento de Antioquia, y cuya fuente de los aislamientos era S. phureja; para Lorica, departamento de Córdoba, la frecuencia fue de 0.07 y los aislamientos provenían de Musa sp; Fuente de Oro, departamento del Meta, presentó una frecuencia de 0.01 y la fuente de los aislamientos fue Musa sp.
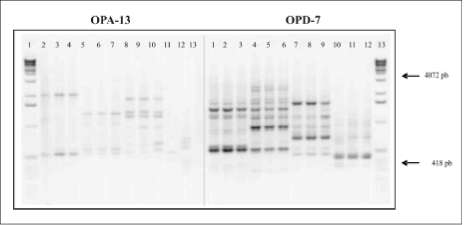
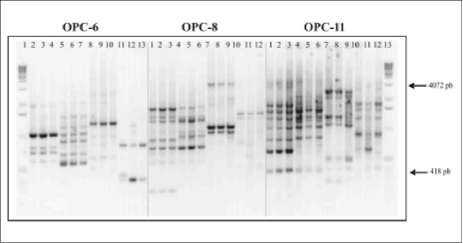
Perfiles electroforéticos para los 5 iniciadores seleccionados con los aislamientos de R. solanacearum. Se presentan los perfiles electroforéticos obtenidos para 12 aislamientos de R. solanacearum con los iniciadores RAPD; OPA 13, OPD 7, OPC 6, OPC 8 y OPC 11; se puede observar gran similitud o patrón monomórfico exis-
|
tente entre los aislamientos F4V F42 y F43 al igual que para el patrón de bandeo de los aislamientos 6.2.,, 6.22 y 3.1r Respecto de los aislamientos Gv G2, G3, M1, M2 y M3 presentaron mayor polimorfismo al momento de la visualización del perfil electroforético (figura 7 y 8).
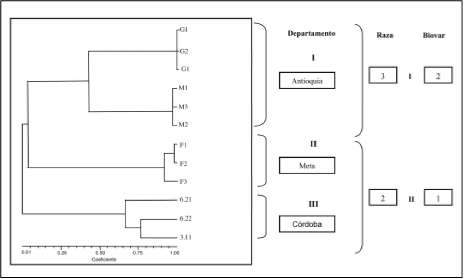
Estimación del coeficiente de correlación cofenético. Se determinó el valor de la correlación entre la matriz de los valores cofenéticos de los datos de similitud y la misma matriz de similitud. Se calculó el coeficiente de correlación cofenético, el cual arroja un valor de r = 0.998, valor muy cercano a 1, lo que permite un grado de confiabilidad muy alto, es decir, que el árbol (dendrograma) representa la matriz de similitud original de una manera confiable y que el método UPGMA es uno de los que crea menor distorsión del árbol respecto de la matriz original de similitud.
Se logra visualizar en el dendrograma obtenido, utilizando las matrices de distancia genética, que el análisis intraespecífico con marcadores RAPD diferenció las procedencias de cada uno de los aislamientos y las agrupó por regiones geográficas. Se muestra tres grupos claramente diferenciados, correspondientes a las tres regiones de donde se colectó el material vegetal, éstas fueron; Antioquia, Meta y Córdoba (figura 9). Además existe la formación de dos grandes grupos conformados por razas de la bacteria, para los ais-lamientos de S. phureja provenientes del departamento de Antioquia su agrupación corresponde a la raza 3 biovar 2, respecto de los aislamientos de Musa sp. que provenían de los
|
|
|
|
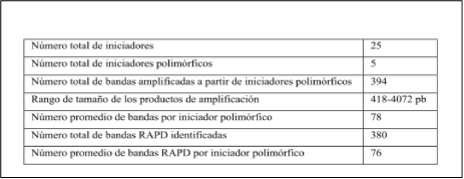 Tabla 9. Resumen de marcadores RAPD en R. solanacearum
|
|
|
|
|
|
23
|
|
|
|
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31
|
|
|
 |
|
|
|
Figura 7. Productos de amplificación PCR-RAPD con los iniciadores OPA 13 y OPD 7. Izquierda: Línea 1: marcador de peso molecular de 1 kb; líneas 2 a la 13: aislamientos Gv G2, G3, Mv M2, M3 F4V F42, F43, 6.2r 6.22y 3.1V respectivamente. Derecha: líneas 1 a la 12: aislamientos Gv G2, G3, Mv M2, M3, F4V F42, F43,6.2.,, 6.22 y 3.1.,, respectivamente. Línea 13: marcador de peso molecular de 1 kb (la relación, origen y fuente de aislamiento, ver tabla 3).
|
|
|
 |
|
|
|
Figura 8. Productos de amplificación PCR-RAPD con los iniciadores OPC 6, OPC 8 y OPC 11. Izquierda: línea 1: marcador de peso molecular de 1 kb; líneas 2 a la 13: aislamientos Gv G2, G3, Mv M2, M3, F4r F42, F43, 6.2.,, 6.22 y 3.1.,, respectivamente. Centro: líneas 1 a la 12: aislamientos Gv G2, G3, Mv M2, M3, F4r F42, F43, 6.2V 6.22 y 3.1V respectivamente. Derecha: líneas 1 a la 12: aislamientos Gv G2, G3, Mv M2, M3 F4V F42, F43, 6.2^ 6.22y 3.1r respectivamente. Línea 13: marcador de peso molecular de 1 kb (la relación, origen y fuente de aislamiento, ver tabla 3).
|
|
|
|
24
|
|
|
|
|
|||
|
ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
|||
|
|
|||
|
departamentos de Meta y de Córdoba la agrupación se da para la raza 2 biovar 1, esto quiere decir que el análisis RAPD logró distinguir los aislamientos por razas segœn el hospedero de la bacteria (figura 9).
El grupo más divergente resulta el del departamento de Antioquia, soportado por el 45%, donde se evidencia la formación de dos subgrupos; un primer grupo correspondiente a los aislamientos Gv G2, G3, que presentan un perfil de bandas similar en la mayoría de los iniciadores probados, también es posible observar en el dendrograma una alta relación entre estos aislamientos. En un segundo grupo están los aislamientos M1, M2, y M3, con valores de similitud de 0.97; este departamento presentó el valor más alto cuando se calculó la frecuencia de variación molecular, soportando de esta forma la divergencia del grupo. Los grupos de Meta y Córdoba, aislamientos: Fv F2, F3 y 6.2.,, 6.22, 3.1.,, respectivamente, son los menos divergentes, presentado valores de similitud altos en un rango de 0.68-0.98. Para el cálculo de la frecuencia de varia-
|
ción molecular estos grupos presentaron valores relativamente bajos.
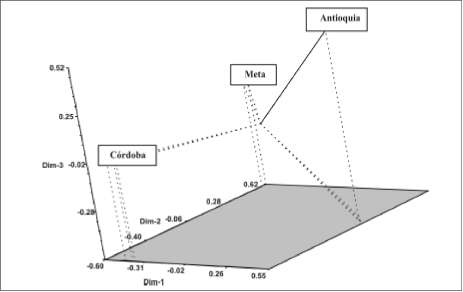
En el gráfico obtenido se muestra la posición tridimensional de tres grupos claramente diferenciados que corresponden a los tres departamentos: Antioquia, Meta y Córdoba; sin embargo, la región de Antioquia de donde provenían los aislamientos de S. phureja resulta la más divergente, evidenciándose la formación de un grupo disperso en la coordenada del departamento. Esto confirma la agrupación por departamentos que arrojó el dedrograma, queriendo decir que los marcadores RAPD discriminaron los aislamientos por fuente geográfica. Respecto de la agrupación por razas, el análisis de coordenadas arroja la ubicación de la raza 2 biovar 1, que son los aislamientos de Musa sp., departamentos de Meta y de Córdoba, posesionados en una misma coordenada, distante a la raza 3 biovar 2, que se encuentra en una coordenada independiente y son aislamientos de S. phureja, cuya fuente es Antioquia, apoyando lo encontrado en el dendrograma (figura 10).
|
||
|
|
|||
 |
|||
|
|
|||
|
Figura 9. Dendrograma construido a partir de la matriz de distancia genética basada en los datos RAPD entre 12 aislamientos de R. solanacearum.
|
|||
|
|
|||
|
25
|
|||
|
|
|||
|
|
|||
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31
|
|||
|
|
|||
|
DISCUSIÓN
Extracción de ADN bacteriano. El método de extracción a partir de cultivos líquidos descrito por Chen and Kuo (1993) permite una buena recuperación de ADN bacteriano en todos los aislamientos estudiados, es un método que requiere de 30 minutos para la extracción y otros 30 minutos para el secado del ADN. Puede ser utilizado para diagnóstico molecular, ya que es un protocolo relativamente corto en comparación con otros más largos que requieren la utilización de lisosima y proteinasa K, que aumentan el tiempo de extracción por más de dos horas. Este método está estandarizado para la obtención rápida de ADN a partir de bacterias gram negativas y se obtiene un ADN de alta calidad.
Diagnóstico de R. solanacearum en semilla de Solanum phureja. La ELISA-NCM es una prueba inmunoenzimática para la que se usan membranas de nitrocelulosa en lugar de placas de microtitulación. Para la lectura de los resultados, la reacción enzimática produce una coloración púr-
|
pura en las muestras positivas, las cuales varían de claro a oscuro según la concentración de R. solanacearum en las muestras.
Es importante advertir que la intensidad de la señal es cualitativa y depende de la agudeza visual del observador (Marinkelle et al., 2000). Ni la coloración morada clara (semejante o más clara a la concentración 107 bact/mL), ni la coloración marrón o verde son consideradas como positivas (Priou, 2001).
Para el análisis de lote de semilla de papa de S. phureja se encontró que las 8 submuestras dispuestas por duplicado sobre la membrana son positivas, observándose la coloración púrpura para cada una de ellas. En el ensayo NCM-ELISA se probaron 4 bacterias de diferente especie a R. solanacearum, encontrándose reactividad cruzada con Pseudomonas chicorii.
La PCR amplificó un producto de 292 pb para 6 submuestras probadas con los iniciadores OLI1 y Y2. De esta manera se logró detectar la bacteria en
|
||
|
|
|||
 |
|||
|
|
|||
|
Figura 10. Análisis de coordenadas principales (PCA). Diagrama en tres dimensiones que muestra las agrupaciones formadas por los aislamientos.
|
|||
|
|
|||
|
26
|
|||
|
|
|||
|
|
|||
|
ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
|||
|
|
|||
|
semilla de papa de S. phureja, la PCR se presenta como una prueba confirmatoria para ensayos inmunoenzimáticos. Respecto de la reactividad cruzada, la PCR no encontró producto de amplificación para la bacteria Pseudomonas chicorii. Los resultados son reproducibles manifestándose la banda patrón para todos los ensayos de PCR.
Diagnóstico de R. solanacearum en semilla de S. tuberosum. En el ensayo de ELISA-NCM se logró detectar 21 tubérculos de S. tuberosum, el equivalente al 59% entre material asintomático y sintomático, y el 41% que corresponde a 15 tubérculos no son detectados por la prueba inmunoenzimática. En comparación con la prueba de PCR que logra detectar un 97%, 35 tubérculos entre asintomáticos y sintomáticos, el tubérculo número 11 resultó negativo a ambas pruebas diagnósticas y corresponde al 3% restante que no es detectado por los ensayos inmunoenzimático y molecular.
Cuando se compararon los resultados de detección para ambas pruebas, enzimática y molecular, se halló que la prueba de PCR logra detectar la bacteria en un 98% de los tubérculos asintomáticos, comparando con el ensayo de ELISA-NCM que detecta el 47% de tubérculos asintomáticos; de esta manera se tiene que la prueba de PCR resulta óptima en la detección de tubérculos ausentes de manifestaciones patológicas, actuando esta prueba como confirmatoria a ensayos inmunoenzimáticos. También puede ser implementada en programas de certificación de semilla de papa.
Sensibilidad y especificidad de la prueba implementada. La prueba de PCR demandó poco tiempo, obteniéndose resultados en un promedio de 12 horas segœn el nœmero de muestras por analizar. Es un ensayo confirmatorio de los resultados dados por inmunoensayos, la especificidad es del 100% segœn el análisis de contingencia, presenta una sensibilidad del 78%. Los datos arrojados por el ensayo de contingencia dejan ver la PCR como una prueba confirmatoria, estandarizada y sustentada con datos numéricos de sensibilidad y especificidad para el diagnóstico rutinario de R. solanacearum a partir de tubérculos semilla en estado asintomático o sintomático. El análisis de PCR parece ser el método más adecuado para una
|
tipificación y para una detección específica, porque a diferencia de lo que sucede con las proteínas y con algunos antígenos, como los lipopolisacáridos utilizados en inmunoensayos, el ADN de una bacteria no sufre ningún cambio en función del estado o crecimiento bacteriano. Por ello, los resultados son mucho más reproducibles y se pueden seleccionar genes que caractericen a cada especie fitopatógena; además presenta niveles de sensibilidad y especificidad de gran interés práctico. No obstante es recomendable trabajar simultáneamente con los ensayos inmunoenzimáticos que presentan buena sensibilidad y un rango de detección de 104 ufc/mL (Robinson, 1993).
Reproducibilidad de la prueba diagnóstica de PCR. Cualquier tipo de prueba diagnóstica debe cumplir con ciertos requisitos que aseguren la efectividad de la misma, como son; especificidad, sensibilidad y, uno muy importante, reproducibilidad. Para la PCR se logró amplificar ADN bacteriano a partir de distintos aislamientos como lo eran papa y plátano, identificándose la raza 3 y 2 con los iniciadores OLI1 y Y2. Se encontró una banda patrón de 292 pb. La importancia de esto es que se cuenta con una prueba altamente específica diseñada y estandarizada para el diagnóstico molecular de R. solanacearum. Estos resultados son semejantes a los obtenidos por Seal et al., (1992, 1993) y Pradhanang et al. (2000).
Amplificación aleatoria de polimorfismos de ADN (PCR-RAPD). Un total de 394 bandas RAPD fueron amplificadas para los aislamientos de R. solanacearum, de las cuales 380 fueron polimórficas. No se evidenció ninguna banda monomórfica entre los 12 aislamientos de la bacteria, es decir, la presencia de alguna banda marcadora género-específica, pero al contrario se observa alto polimorfismo entre aquellos aislamientos provenientes de las tres distintas regiones colombianas (Antioquia, Meta y Córdoba). Los perfiles de las bandas son característicos para cada uno de ellos; si se observa los patrones de las bandas para cada una de las razas de la bacteria: raza 3 biovar 2 y raza 2 biovar 1, representadas por los aislamientos provenientes de S. phureja y Musa sp. respectivamente, se muestran marcadores RAPD raza-específicos para aislamientos de S. phureja, raza 3 biovar 2, que pueden ser tomados como patrones moleculares; sin embargo, no es posible
|
||
|
|
|||
|
27
|
|||
|
|
|||
|
|
|||
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31
|
|||
|
|
|||
|
proponerla aœn como marcador de raza, debido a las posibles diferencias de secuencias que pueden haber entre las amplificaciones, lo que atiende a uno de los problemas que presenta la técnica; habría que realizar estudios fingerprinting (huella digital por PCR) u otras caracterizaciones moleculares con distintas metodologías para confirmar el anterior resultado.
Respecto a la raza 2 biovar 1 no se evidencia ningún patrón molecular consistente raza-específico; no obstante, las agrupaciones por razas se logran ver en el árbol (dendrograma) conformado por dos grandes grupos, de los cuales hace parte cada una de las razas señaladas. Esto evidencia la elevada variabilidad intraespecífica, tanto a nivel bioquímico-molecular como de patogenicidad, en función de las distintas especies de vegetales que actœan como hospedadoras de la bacteria (Hayward, 1994). Los coeficientes de similaridad demuestran la estrecha similitud entre las cepas de R. solanacearum, cuyos valores oscilan 0.68 y 1.00 (68%-100% de similaridad). En el dendrograma se pudo observar la formación de tres grupos, reuniendo las tres regiones geográficas colombianas, donde se colectaron los aislamientos de la bacteria.
El departamento de Antioquia se identificó como el más divergente, soportado por el 40%, pero con una alta similitud entre los dos subgrupos conformados por seis aislamientos de la bacteria y que fueron obtenidos de S. phureja. Para los aislamientos Gv G2 y G3, el índice resultó de 100%, el resto de aislamientos Mv M2 y M3 presentaron el 97% de similaridad, esto soporta el patrón molecular observado, donde se visualiza 4 bandas marcadoras que corresponden a; 1569, 1477, 1100 y 517 pares de bases. En el departamento del Meta, el análisis resultó con índices que oscilan entre 0.94 y 0.97 para los aislamientos Fv F2 y F3 obtenidos de Musa sp., esto sugiere una estrecha relación genética entre estos aislamientos. Córdoba representada por tres aislamientos cuya fuente era Musa sp. tiene valores de similaridad entre los rangos de 0.68 y 0.76, las cepas 6.2V 6.22y 3.12son las de más bajo índice, pero sus valores no dejan de ser significativos y demuestran una cercanía genética. Estos aislamientos han sido sometidos a procesos de criopreservación y utilizados como aislamientos con-
|
trol al inicio de la investigación, queda ver si el mantenimiento y reactivación de las cepas provenientes de criopreservación influye en la integridad del genoma bacteriano. Es importante señalar que el análisis RAPD también discriminó los aislamientos de R. solanaceraum por razas, lográndose detectar dos grandes grupos que correspondían a la raza 3 biovar 2, representada por aislamientos a partir de S. phureja y raza 2 biovar 1, aislamientos realizados a partir de Musa sp.
Para el análisis de frecuencia de variación molecular, los datos arrojados son similares a los encontrados por el dendrograma. Las colectas o genotipos traídos de Antioquia presentaron un valor de 0.34, de esta forma se confirma como el grupo de mayor variación respecto al nodo de la formación de los subgrupos del dendrograma, que se encuentra en una valor de similaridad del 40%, lo que puede deberse principalmente a la persistencia de la bacteria por varios años en esta región y al manejo que se le ha dado a la enfermedad, generándose presión de selección y de esta manera alta variabilidad genética, dificultándose algunas veces el control de R. solanacearum en cultivos de papa. Es importante relacionar esta diversidad con la agresividad de la bacteria; estos aislamientos resultaron patológicamente muy activos, aumentado la severidad de la infección en cuestión de días, esto se observó al momento de la recepción y manejo del material en el laboratorio. Los aislamientos que tienen menor frecuencia de variación molecular son los de Musa sp. departamentos del Meta y de Córdoba con valores de 0.01 y 0.07 respectivamente, confirmado los datos del dendrograma, no obstante el índice 0.07 correspondiente a Córdoba presenta una diferencia significativa de 0.06 en contra del departamento de Meta si consideramos que la fuente de los aislamientos era Musa sp.
El análisis de coordenadas principales (APC) muestra 3 grupos diferenciados, construidos por las tres regiones geográficas en estudio. El análisis en tres dimensiones logró discriminar en dos coordenadas las respectivas razas de la bacteria, raza 3 y 2; esto soporta la elevada variabilidad intraespecífica encontrada entre las razas y los departamentos, evidenciándose en el dendrograma grupos independientes.
|
||
|
|
|||
|
28
|
|||
|
|
|||
|
|
|||
|
ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
|||
|
|
|||
|
Varios métodos de caracterización para la identificación de la variabilidad genética de R. solanacearum han sido utilizados: PCR-RFLP, Rep-PCR y RAPD (Jaunet and Wang, 1999; Louws et al., 1994 y Poussier et ál., 1999), y más recientemente AFLP (Poussier et al., 2000). La técnica SDS-PAGE también ha sido œtil para la caracterización de la diversidad genética de R. solanaceraum. Dristig y Dianese (1990) trabajaron en la caracterización de proteínas de membrana de la bacteria en distintos biovares, encontrando agrupaciones individuales para cada biovar. Smith et ál. (1995) aplicaron la metodología de electroforesis de campo pulsado para fragmentos deADN genómico digeridos porendonucleasasde restricción (RC-PFGE) en aislamientos de R. solanacearum que pertenecían a la raza 3. Es importante resaltar que este grupo de investigación encontró la raza 3 muy homogénea, resultados similares a los obtenidos en este estudio.
En trabajos recientes realizados por Thwaites et ál. (1999) se demostró el poder de discriminación de los marcadores RAPD para agrupar los aislamientos por posición geográfica y razas de la bacteria, resultados consistentes con el presente estudio. Otra herramienta de caracterización han sido los AFLP. Recientemente Poussier et al. (2000) logró caracterizar un buen número de asilamientos de varias partes del mundo, demostrando que la bacteria presenta una alta diversidad intraespecífica, permitiendo generar agrupaciones por análisis AFLP por origen geográfico, razas y biovares. Son consistente los resultados obtenidos por varios grupos de investigación alrededor del mundo, cuando en la mayoría de análisis por índices de símilaridad y su respectiva agrupación en árboles o dedrog ramas las agrupaciones siguen siendo por posición geográfica y razas, notándose la elevada variabilidad del patógeno en distintas partes del mundo, presentándose como una bacteria muy variable y de amplia distribución en el mundo. Cabe resaltar que esta variabilidad puede estar asociada con genes que confieren tolerancia a condiciones ambientales adversas o incluso con resistencia a bactericidas, lo que dificulta el control de la bacteria en ciertas regiones. La existencia de variación genética para un patógeno es importante porque ésta puede traducirse en diferencias en la virulencia o respuestas para su control químico, siendo de gran
|
relevancia para conocer la estructura de la población de un microorganismo (Tibayrenc, 1996; Sánchez, 2004).
CONCLUSIONES
El protocolo descrito por Chen and Kuo (1993) permite una buena recuperación de ADN bacteriano en los aislamientos estudiados y puede ser implementado en el diagnóstico molecular de R. solanacearum.
La PCR presenta una alta especificidad y sensibilidad comparada con la prueba estándar NCM-ELISA, el 100% y 78%, respectivamente; estos rangos resultan óptimos para implementar el ensayo de PCR como prueba rutinaria en el diagnóstico de la bacteria, siendo reproducible, e identifica razas 1, 2 y 3.
La PCR detectó tubérculos asintomáticos de S. tuberosum previo prenriquecimiento de la muestra en comparación con la prueba ELISA-NCM, que no detectó en su totalidad los tubérculos asintomáticos.
Es posible utilizar la PCR en programas de evaluación epidemiológica que impliquen el análisis de poblaciones y el movimiento del material de siembra, para el caso de importaciones y exportaciones, estudio de poblaciones y áreas libres o con presencia de la bacteria para el control fitosanitario de la bacteria.
El análisis intraespecífico con marcadores moleculares RAPD diferenció las procedencias de R. solanacearum y las agrupó por regiones geográficas y razas bacterianas, indicando que hay diferencias entre las colectas de acuerdo con su región geográfica. Lo que hace suponer la alta variabilidad genética de la bacteria.
La raza 3 biovar 2 presenta un patrón molecular que puede ser usado como marcador raza-específico para poblaciones bacterianas colombianas; no obstante la diferencia de pesos moleculares y variación de secuencias, se presenta como un inconveniente de la técnica de PCR-RAPD para este fin.
|
||
|
|
|||
|
29
|
|||
|
|
|||
|
|
|||
|
Rev. Colomb. Biotecnol. Vol. VIII N° 1 Julio 2006 14-31
|
|||
|
|
|||
|
La variabilidad genética encontrada y los análisis de agrupamiento por dendrogramas y análisis de coordenadas principales (PCA), construidos por metodologías moleculares, no permiten considerar a R. solanacearum como un grupo homogéneo genéticamente, esto implica que el control de la bacteria en ciertas zonas del país productoras de papa y plátano se dificulte, lo que constituye una relación genética asociada con genes de resistencia a ciertos bactericidas.
AGRADECIMIENTOS
Los autores agradecen al Instituto Colombiano Agropecuario-ICA, Congelados Agrícolas-CONGELAGRO S.A., Corporación Colombiana de Investigación Agropecuaria-CORPOICA por la financiación de la presente investigación. A los doctores María del Rosario Silva (UNILLANO) y José Luis Zapata (CORPOICA-La Selva). A Luisa Fernanda Páez (CORPOICA-Plan Maíz) y Julián Martínez H. (ICA-LANAM).
BIBLIOGRAFÍA
Agrios, G. 2001. Fitopatología. México: Editorial Limusa. 571-572.
Boucher, C; Barberis, A.; Trigales, A.; Démery, D. 1985. Transposon mutagenesis of Pseudomo-nas solanacearum: isolation of Tn5-induced avirulent mutants. Journal of General Microbiol-ogy. 131: 2449-2457.
Chen W.; Kuo, T. 1993. A simple and rapid method for the preparation of gram negative bacterial genomic DNA. Nucleic Acid Research. 21: 2260.
Dristig, M.G.C.; Dianese, J.C. 1990. Characterization of Pseudomonas solanacearum biovars based on membrane protein patterns. The American Phytopathological Society. 80: 641-646.
Daniel, W. 1995. Bioestadística. 5 ed. México: Edit. Uteha.
French, E.; L. Gutarra. 1995. Culture media for Ralstonia solanacearum isolation, identification and maintenance. Phytopathology. 30(3): 126-130.
García, R. 1999. Distribución, incidencia y variabilidad de Ralstonia solanacearum, agente causal de la marchitez bacteriana de la papa en el estado de Mérida. Bioagro. 11(1): 12-23.
Hayward, A. 1991. Biology and epidemiology of bacterial wilt caused by Pseudomonas
|
solanacearum. Annu. Rev. Phytopathology. 29: 65-87
Hayward, A.; Hartman, G. 1994. Bacterial wilt the disease and it's causative agent. Pseudomonas solanacearum. Ed CAB Intemational Guilford.
Jaunet, T.X.; J.F. Wang. 1999. Variation in genotype and aggressiveness of R. solanacearum race 1 isolated from tomato in Taiwan. Phytopathology. 89:320-327.
López, J.. 2004. Determinación de variabilidad genética entre aislamientos de Rosellinia Sp., Rosellinia bunodes y Rosellinia pepo mediante la técnica de amplificación aleatoria de polimorfismos de DNA (RAPD) y análisis de espaciadores de transcritos internos (ITSs). Trabajo de grado para optar al titulo de Biólogo. Bogotá: Universidad Nacional de Colombia.
Lozada, R. 2001. Detección Ralstonia solanacearum en un suelo de uso potencial para la multiplicación de semilla de papa utilizando la técnica molecular de PCR. Trabajo de grado para optar el titulo de Magíster en Microbiología. Bogotá: Universidad Javeriana.
Louws, F.J.; Fulbright; D.W.; Stephens, C; Bruijn, F.J. 1994. Specific genomic fingerprints of phytopathogenic Xanthomonas and Pseudomonas pathovars and strains generated with repetitive sequences and PCR. Appl. Environ. Microbiol. 60: 2286-2295.
Maniatis, T.; Sambrook, J. 1982. Molecular Cloning. A Laboratory Manual. New York: Cold Spring Harbor.
Marinkelle, C.J.; Piragauta, A.; Giraldo, F.; Castañeda, H. 2000. Estandarización de la técnica Dot-Elisa para el serodiagnóstico del cisticerco vesicular Taenia solium en porcinos. Revista Científica UNINCCA. 6(1): 35-50.
Marulanda, M.L.; Márquez, M. 2002. Evaluación de la estabilidad genética de vitroplantas de Rubus glaucus mediante marcadores moleculares RAPD. Actualidades Biológicas. 24(76): 31-36.
Nei, M.; S. Kumar. 2000. Molecular evolution and phylogenetics. New York: Oxford University Press. 285-288.
Pérez, L. 1993. Enfermedades de las plantas. Medellín: Edit. Lealon. 102-106.
Poussier, S.; Demery-Trigalet, D. 2000. Genetic diversity of R. solanacearum as assessed by PCR-RFLP of the gene región, AFLP and 16S rRNA sequence analysis, and identification of an
|
||
|
|
|||
|
30
|
|||
|
|
|||
|
|
|||
|
ESTABLECIMIENTO DE UN SISTEMA DIAGNÓSTICO PARA LA DETECCIÓN DE Ralstonia solanacearum
|
|||
|
|
|||
|
African subdivisión. Microbiology. 146: 1679-1692.
Poussier, S. 1999. Genetic diversity of African and worldwide strains of R. solanacearum as determined by PCR-RFLP analysis of the hrp gene region. Appl. Environ. Microbiol. 65: 2184-2194.
Pradhanang, P.; Elphinstone, J.; Fox, R. 2000. Identification of crop and weed hosts of Ralstonia solanacearum biovar 2 in the hills of Nepal. Plant Pathology. 49: 403-413.
Pradhanang, P.; Elphinstone, J.; Fox, R. 2000. Sensitive detection of Ralstonia solanacearum in soil: a comparasion of different detection techniques. Plant Pathology. 49: 414-422.
Priou, S. 2001. Kit ELISA-NCM para la detección de R. solanacearum en papa. Centro Internacional de la Papa (CIP).
Robinson, A. 1993. Serological detection of Pseudomonas solanacearum by ELISA. Proceedings of the international bacterial wilt. Symposium, Kaohsiung, Taiwan, 28-31 de octubre, 1992. ACIAR, Camberra, Australia, 54-61.
Raba, M. Andrés.; Ángel, E.; González, F. 2003. Caracterización genética de poblaciones de la mosca suramericana de las frutas Anastrepha fraterculus (Wiedemann) en Colombia. Revista ICA Informa. 30: 8-14.
Sánchez, Q.; A. Zilpa. 2004. Evaluación de la variabilidad genética de aislamientos ambientales de Cryptococcus neoformans recuperados en Bogotá D.C. mediante la técnica de huella digital por PCR. Trabajo de grado para optar al titulo de Bióloga. Bogotá: Universidad INCCA de Colombia (UNINCCA).
Saunders, D. 1999. Bioestadística Médica. México: Edit. Manual Moderno. 275-292.
Seal, S.; Jackson, L; Daniels, M. 1992. Isolation of a Pseudomonas solanacearum specific DNA
|
probé by subtraction hybridization and construction of species specific oligonucleotide pimers for sensitive detection by the polymerase chain reaction. Appl. And Environ. Microbial. 58: 3751-3758.
Seal, S.; Taghavi, M.; Fegan, N.; Hayward, A.; Fegan, M. 1999. Determination of Ralstonia solanacearum rDNA subgroups by PCR test. Plant Pathology. 48: 115-120.
Smith, J.; Offord, C; Holdemess, M.; Saddler, S. 1995. Genetic diversity of Burkholderia solanacearum race 3 in Kenya. Appl. Environ. Microbiol. 61: 4263-4628.
Sneath, P.; Sokal, R. 1987. Principles of numerical taxonomy. London: Freeman.
Tybayrenc, M. 1996. Towards a unifled evolutionary genetics of microorganisms. Annu. Rev. Microbiol. 50: 401-429.
Thwaites, R.; Mansfield, J.; Edén - Green, S.; Seal, S. 1999. RAPD and rep PCR-based fingerprinting of vascular bacterial pathogens of Musa spp. Plant Pathology. 48: 121-128.
Williams, J.G.K.; Kubelik, K.J.; Rafalski, J.A.; Tingey, S.V. 1990. DNA polymorphis amplified by arbitrary primers are useful as genetic markers. Nucleic Acid Research. 18: 6531-6535.
Williamson, L; Nakaho, K.; Hudelson, B.; Allen, C. 2002. Ralstonia solanacearum race 3, biovar 2 strains isolated from geranium are pathogenic on potato. American Phytopathology Society. 86: 987-991.
Yabuuchi, E.; Kosako, Y; Yano, I.; Hotta, H.; Nishiuchi, Y. 1995. Transfer of two burkholdeira and an Alcaligenes species to Ralstonia Gen. Nov: Proposal of Ralstonia pickettii (Ralston, Palleroni and Duodoroff, 1973) Comb. Nov., Ralstonia solanacearum (Smith, 1896) Comb. And Ralstonia eutrpha (Davis, 1969) Comb. Nov. Microbiol. Immunol. 39: 897-904.
|
||
|
|
|||
|
31
|
|||
|
|
|||
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2006 Revista Colombiana de Biotecnología

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
Esta es una revista de acceso abierto distribuida bajo los términos de la Licencia Creative Commons Atribución 4.0 Internacional (CC BY). Se permite el uso, distribución o reproducción en otros medios, siempre que se citen el autor(es) original y la revista, de conformidad con la práctica académica aceptada. El uso, distribución o reproducción está permitido desde que cumpla con estos términos.
Todo artículo sometido a la Revista debe estar acompañado de la carta de originalidad. DESCARGAR AQUI (español) (inglés).


















