ANÁLISIS COMPARATIVO DEL GENOMA MITOCONDRIAL EN GASTRÓPODOS
Palabras clave:
Gastrópoda, genoma mitocondrial, reorganizaciones génicas, tasas de evolución molecular (es)Los genomas mitocondriales en el grupo Gastrópoda muestran altas tasas de variación entre ellos en cuanto a la organización de sus genomas lo que lo convierte en un excelente grupo modelo para el estudio de la evolución del genoma mitocondrial. En este trabajo presentamos un análisis comparativo de los genomas mitocondriales en gastrópodos. Se calculó la composición de nucleótidos y de aminoácidos de todas las secuencias y se hizo un análisis visual comparativo de los codones de inicio y de parada. La organización del genoma se comparó calculando el número de secuencias intergénicas, la ubicación de los genes y el número de reorganizaciones génicas (“breakpoints”) en comparación con la secuencia que se presume ancestral para el grupo. Para calcular si existen variaciones en las tasas de evolución molecular en el grupo, estas últimas se calcularon utilizando el “relative rate test”. A pesar de las diferencias en el tamaño de los genomas, el número de amino acidos es mas conservado. La composicion nucleotidica y aminoacidica es similar entre los Vetigastropoda, Ceanogastropoda y Neritimorpha en comparacion con Heterobranchia y Patellogastropoda. Los genomas mitocondriales para el grupo son muy compactos con pocas secuencias intergenicas, la unica excepción es el genoma de Patellogastropoda con 26.828 pb. Existe una alta variabilidad en cuanto a codones de inicio para los grupos Heterobranchia y Patellogastropoda y un aumento en el numero de genes reorganizados con respecto a la secuencia de O. vulgaris también para estos dos grupos. En general se rechaza la hipotesis de tasas de evolución molecular constante entre los grupos, excepto cuando se comparan los genomas de Neritimorpha y Vetigastropoda.
ANÁLISIS COMPARATIVO DEL GENOMA MITOCONDRIAL EN GASTRÓPODOS
Comparative Analysis of the Mitochondrial Genomes in Gastropods
MOISES ARQUEZ1, Biólogo; JUAN ESTEBAN URIBE1, Biólogo; LYDA RAQUEL CASTRO1,2, Ph. D.
1Grupo de Investigación en Evolución, Sistemática y Ecología Molecular. INTROPIC. Santa Marta, Colombia. moarquezm@hotmail.com, juanes@mncn.csic.es 2 Programa Biología. Universidad del Magdalena. Laboratorio 2. Carrera 32 n.° 22-08. Sector San Pedro Alejandrino. Santa Marta, Colombia. Tel.: 57-5-430 12 92, ext. 273. lydaraquelcastro@hotmail.com
Presentado el 2 de marzo de 2012, aceptado el 10 de abril de 2012, correcciones el 16 de mayo de 2012.
RESUMEN
En este trabajo presentamos un análisis comparativo de los genomas mitocondriales en gastrópodos. Se calculó la composición de nucleótidos y de aminoácidos de todas las secuencias y se hizo un análisis visual comparativo de los codones de inicio y de parada. La organización del genoma se comparó calculando el número de secuencias intergénicas, la ubicación de los genes y el número de reorganizaciones génicas (breakpoints) en comparación con la secuencia que se presume ancestral para el grupo. Para calcular si existen variaciones en las tasas de evolución molecular en el grupo, estas últimas se calcularon utilizando el relative rate test. A pesar de las diferencias en el tamaño de los genomas, el número de aminoácidos es más conservado. La composicion nucleotídica y aminoacídica es similar entre los Vetigastropoda, Ceanogastropoda y Neritimorpha en comparacion con Heterobranchia y Patellogastropoda. Los genomas mitocondriales para el grupo son muy compactos con pocas secuencias intergenicas, la unica excepción es el genoma de Patellogastropoda con 26.828 pb. Existe una alta variabilidad en cuanto a codones de inicio para los grupos Heterobranchia y Patellogastropoda y un aumento en el número de genes reorganizados con respecto a la secuencia de O. vulgaris también para estos dos grupos. En general, se rechaza la hipótesis de tasas de evolución molecular constante entre los grupos, excepto cuando se comparan los genomas de Caenogastropoda y Vetigastropoda.
Palabras clave: gastrópoda, genoma mitocondrial, reorganizaciones génicas, tasas de evolución molecular.
ABSTRACT
In this work we presented a comparative analysis of the mitochondrial genomes in gastropods. Nucleotide and amino acids composition was calculated and a comparative visual analysis of the start and termination codons was performed. The organization of the genome was compared calculating the number of intergenic sequences, the location of the genes and the number of reorganized genes (breakpoints) in comparison with the sequence that is presumed to be ancestral for the group. In order to calculate variations in the rates of molecular evolution within the group, the relative rate test was performed. In spite of the differences in the size of the genomes, the amino acids number is conserved. The nucleotide and amino acid composition is similar between Vetigastropoda, Ceanogastropoda and Neritimorpha in comparison to Heterobranchia and Patellogastropoda. The mitochondrial genomes of the group are very compact with few intergenic sequences, the only exception is the genome of Patellogastropoda with 26,828 bp. Start codons of the Heterobranchia and Patellogastropoda are very variable and there is also an increase in genome rearrangements for these two groups. Generally, the hypothesis of constant rates of molecular evolution between the groups is rejected, except when the genomes of Caenogastropoda and Vetigastropoda are compared.
Key words: Gastropoda, mitochondrial genome, genome reorganizations, rates of molecular evolution.
INTRODUCCIÓN
El genoma mitocondrial (mt), al igual que el nuclear, está constituido por una doble cadena de nucleótidos, cada una de las cuales se denomina ligera (L) o pesada (H) debido a la densidad diferencial que muestran cuando se centrifugan empleando cloruro de cesio, pero a diferencia del genoma nuclear, es una molécula pequeña en cuanto a su extensión pese a la importancia de la información que codifica. En mamíferos esta molécula puede alcanzar los 20 Kb de longitud, aunque dependiendo del tipo de organismo del que se haga referencia puede ser menor o mayor. Este genoma generalmente no presenta intrones excepto por las secuencias regulatorias de unas cuantas bases (Taanman, 1999) y en mamíferos codifica 37 genes, la mayoría de los cuales se localizan en la hebra pesada; la hebra ligera únicamente codifica ocho ARN de transferencias (ARNt) y un solo ARN mensajero (ARNm; Attardi y Schatz, 1988). En adición, los genes se encuentran separados por secuencias de nucleótidos cortas, que no codifican productos (Burger et al., 2003).
El genoma mitocondrial codifica menos de 40 genes y los productos de estos genes (2 ARNr, 22 ARNt y 13 proteínas, en animales) están relacionados en su totalidad con la cadena respiratoria de la célula, transporte de electrones y fosforilación oxidativa (Stuart y Brown, 2006). A pesar de la función conservada del ADN mitocondrial, porque todos los productos de su codificación están relacionados con la producción de energía, es posible observar una gran diversidad de genomas mitocondriales entre los eucariotas, incluso entre organismos cercanamente relacionados. Esta diversidad aparentemente no presenta variaciones funcionales drásticas y se limita a las características estructurales de la molécula (Burger et al., 2003).
En cuanto a la estructura, es aceptado de forma general que el genoma mitocondrial es circular, sin embargo, existen organismos que poseen genomas mt lineales, este es el caso de Amoebidium parasiticum, Plasmodium, Chlamydomonas (Burger et al., 2003) y muchos otros organismos entre los que además se registran especies de cnidarios (Bridge et al., 1992). Por otra parte puede presentarse que, durante los análisis con enzimas de restricción un genoma mitocondrial de característica lineal pueda ciclarse o viceversa, y por ende darse una falsa interpretación de su estructura. Estudios de genomas mitocondriales, que emplean electroforesis de campo pulsado en gel, han demostrado que existen organismos con muchas fracciones de ADN mitocondrial de estructura lineal, cuyo genoma ha sido reportado como circular, este el caso del protozoario Tetrahymena pyriformis y el ciliado Paramecium aurelia (Nosek y Tomáska, 2003).
Así mismo, los genomas mitocondriales presentan grandes variaciones en cuanto a su extensión, para dar un ejemplo se pueden citar los casos más extremos que corresponden a los genomas de Apicomplexa y Oriza sativa, con 6 y 400kb de extensión respectivamente (Nosek y Tomáska, 2003). Además de las diferencias en tamaño y forma, con el advenimiento de las técnicas avanzadas de investigación en biología molecular, y la secuenciación de cada vez más genomas mitocondriales, se han podido determinar diferencias más profundas que aumentan la diversidad génica mitocondrial. Tales diferencias hacen referencia a la organización de los genes dentro del genoma.
Las reorganizaciones génicas, son cambios en la posición de los genes dentro del genoma, que tiene como resultado un nuevo arreglo u organización de la molécula. Este tipo de fenómenos es por lo regular mediado por enzimas (topoisomerasas) y altera la estructura normal del genoma por los cambios que se producen generando, en conclusión, un nuevo genoma (Grande et al., 2008). Las reorganizaciones génicas presentan también características que los proponen como excelentes marcadores moleculares, pero se han sugerido estudios comparativos de grupos sujetos a altas tasas de reorganización génica con el fin de incrementar el conocimiento de la evolución de estos cambios (Boore y Brown, 1998; Dowton et al., 2002).
De los genomas mitocondriales completos de gastrópodos secuenciados y disponibles en GenBank, la mayoría pertenecen al grupo Heterogastropoda y Caenogastropoda, con pocos genomas disponibles para los grupos Vetigastropoda, Patellogastropoda y Neritimorpha. Estos genomas muestran altas tasas de variación entre ellos en cuanto a su organización, lo que convierte a los gastrópodos en un excelente grupo modelo para el estudio de la evolución del genoma mitocondrial. En este trabajo se hace un análisis comparativo de la organización, el contenido y la dinámica de los genomas mitocondriales en gastrópodos.
MATERIALES Y MÉTODOS
Se obtuvieron los genomas mitocondrial de 48 gastrópodos, disponibles en GenBank (http://www.ncbi.nlm.nih.gov/genbank/). Debido al gran número de genomas mt secuenciados para el grupo Heterobranchia y Caenogastropoda, cuatro para Vetigastropoda, y un solo representante para los grupos Patellogastropoda y Neritimorpha, en algunos análisis se utilizó una submuestra más homogénea de 16 genomas de gastrópodos que incluyó cinco Caenogastropoda, seis Heterobranchia, los cuatro Vetigastropa disponibles y el único representante de los demás grupos.
Las secuencias fueron alineadas utilizando CLUSTAL X (Thompson et al., 1997), en el programa de libre distribución MEGA (v. 4) (Tamura et al., 2007). MEGA permitió hacer el alineamiento de las secuencias de aminoácidos con base en las secuencias de nucleótidos respectivas. Este programa también fue utilizado para calcular la composición de nucleótidos y de aminoácidos de todas las secuencias. Los alineamientos se usaron a su vez, para hacer un análisis visual comparativo de los codones de inicio y de parada. En este caso, se hizo el muestreo de todos los codones de inicio y de parada de todos los genes que codifican para proteína exceptuando los codones de parada del gen NAD6 para el taxón Cepaea nemoralis y el codón de parada para el gen NAD2 del taxón Piramidela dolabrata, pues su genoma estaba incompleto.
La organización del genoma se comparó calculando el número de secuencias intergénicas, la ubicación de los genes (cadena ligera o pesada) y el número de reorganizaciones génicas en comparación con la secuencia que se presume ancestral para el grupo, la del cephalopodo Octopus vulgaris (Grande et al., 2008; Ki et al., 2010). La medida más simple para calcular el número de reorganizaciones génicas entre dos genomas es el break point number (BP) (Blanchette et al., 1999). Consideramos este método como el más adecuado porque es independiente para el tipo de reorganización (inversión, transposición, transposición+ inversión) (Xu et al., 2006), el cual no conocemos. El BP se calculó utilizando el programa SPRING (Lin et al., 2006). Para grupos con más de un genoma disponible, como es el caso de Heterobranchia y Caenogastropoda, se calculó el BP promedio.
Para calcular si existen diferencias en las tasas de evolución molecular del genoma mt entre los diferentes grupos de gastrópodos, las secuencias alineadas y concatenadas de todos los genes que codifican para proteína, se importaron a MEGA versión 4.0 (Tamura et al., 2007) para realizar el relative rate test (RRT) (Tajima, 1993). El test de Tajima considera tres secuencias, 1, 2, y 3, la secuencia 3 como grupo externo. Esta prueba examina si E(nijk) = E(njik), en donde i, j, y k representan diferentes nucleótidos en las secuencias 1, 2 y 3, respectivamente. Bajo la hipótesis de reloj molecular, E(nijk) = E(njik), independientemente del modelo de sustitución e independientemente de si las tasas varían en cada sitio. Si la hipótesis es rechazada, la hipótesis de reloj molecular puede ser rechazada para ese set de secuencias. Para nuestros análisis, utilizamos la secuencia de O. vulgaris como grupo externo (secuencia 3) y se hicieron comparaciones entre cada uno de los grupos principales de gastrópodos.
RESULTADOS Y DISCUSION
COLECCIÓN DE SECUENCIAS
El muestreo total fue de 48 gastrópodos disponibles en GenBank: Heterobranchia con 26 integrantes; Caenogastropoda con 16 integrantes; Vetigastropoda con 4 integrantes; Patellogastropoda con 1 integrante y Neritimorpha con 1 integrante (Tabla 1).
COMPOSICIÓN AMINOACÍDICA Y NUCLEOTÍDICA
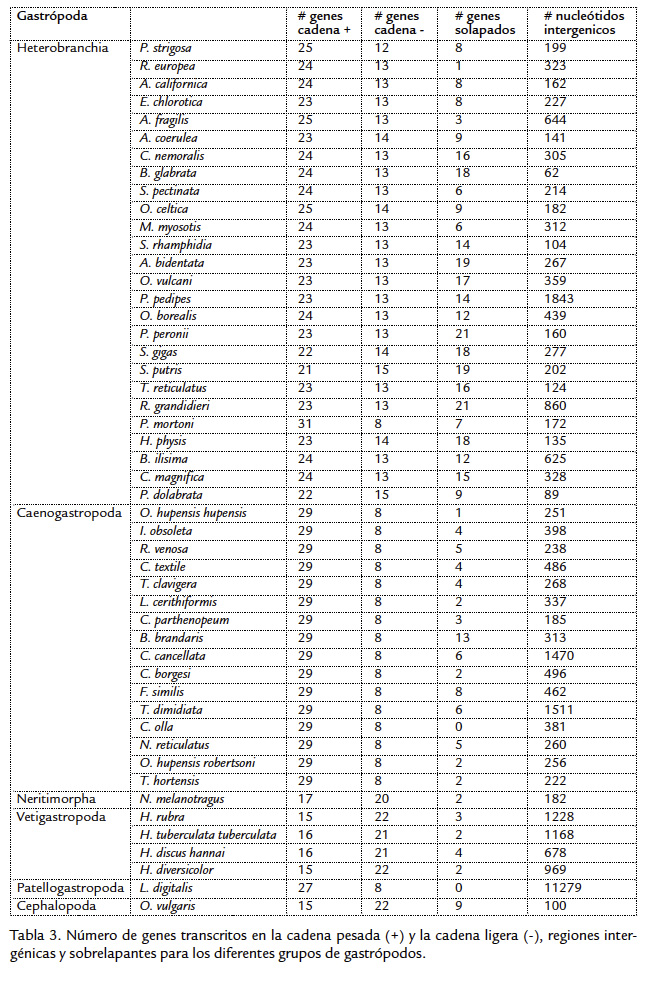
Comparando el número de aminoácidos totales nos damos cuenta que se forman dos grupos, por un lado (Neritimorpha, Caenogastropoda y Vetigastropoda) y por el otro (Patellogastropoda y Heterobranchia (Opistobranchia, Pulmonata y Piramidelloidea)). El primer grupo presenta mayor número de aminoácidos que el segundo grupo (Fig. 1). Al comparar la extensión del genoma (número total de nucleótidos), se observa que solo una especie perteneciente al grupo Patellogastropoda (Lottia digitalis) presenta una diferencia considerable a todos los demás grupos, con un valor total de 26.828 pb comparado con el resto de los genomas con valores promedio de 15.204 pb. En cuanto a la comparacion de nucleótidos se observa que el número de adenina, timina, guanina y citocina es similar en los Neritimorpha y los Caenogastropoda. Heterobranchia es el grupo con genomas más compactos, ninguno supera las 15.000 pb (Tabla 2; Fig. 2). Aunque los genomas mt en vertebrados no varían mucho en tamaño (entre 16 y 18 kb aproximadamente), el tamaño de los genomas mt en invertebrados es mucho más variable (15 a 78 kb; Gray, 1989). Estas variaciones pueden deberse a diferencias pequeñas en el tamaño de los genes, a la carencia de algún gen, como se ha visto en Nematodos (Okimoto et al., 1992), a la presencia de pseudogenes o a diferencias en el número de nucleótidos intergénicos. En el caso de Patellogastropoda, el único grupo de gastrópodos con un genoma mt considerablemente más grande, este tamaño se debe principalmente a la presencia de secuencias intergénicas largas.
El porcentaje A-T es muy similar en todos los grupos y al igual que en otros invertebrados, los gastrópodos presentan un % A-T más alto que el % G-C, con valores que oscilan entre 58,19 % y 70,65 % A-T. A pesar de las diferencias en el tamaño de los genomas, el número de aminoácidos es más conservado. Se puede ver, por ejemplo, que el genoma de Lottia digitalis cuya longitud sobrepasa enormemente la de los otros, codifica para un número similar de aminoácidos (Tabla 2).ANÁLISIS DE CODONES DE INICIO Y PARADA
El codón ATG es el más frecuente, en especial para los grupos Patellogastropoda, Caenogastropoda, Neritimorpha y Vetigastropoda. En los Vetigastropoda y Neritimorpha solo se presentaron dos tipos de codones de inicio (ATA y ATG), en los Caenogastropoda tres tipos de codones de inicio (ATG, ATA y ATT), en los Patellogastropoda se observan los dos tradicionales (ATG y ATT) y uno novedoso que solo se presenta en ese grupo (CTG). En el grupo Heterobranchia se observaron todos los codones anteriores más dos nuevos (GTG y ATC). En el genoma mitocondrial ATG suele ser el codón de iniciación más común para los genes que codifican para proteínas, sin embargo, se han publicado varias excepciones en donde GTT, GTG, ATG, ATC y ATT han sido también propuestos para ser usados como codones de inicio (Tzeng et al., 1992). Los codones de parada no son tan diversos y solo se presentan dos tipos TAG y TAA, estos no mostraron diferencias entre los grupos.
ORGANIZACIÓN DE GENOMA
Regiones intergénicas y sobrelapantes. Los genomas mt en gastrópodos son bastante compactos y con excepción del Patellogastropoda, en donde el número de nucleótidos intergénicos es de 11.279, el número total de nucleótidos intergénicos para los otros grupos va desde 89 en Pyramidalla dolabrata hasta 1.228 en Haliotis rubra (Tabla 3). El número promedio de nucleótidos entre genes es menor a 10 para todo el grupo (sin contar Patellogastropoda). Dentro de los gastrópodos, los genomas más compactos están en el grupo Heterobranchia con genomas de tamaños inferiores a 15.000 pb.
Al igual que en otros genomas mt de invertebrados, en gastrópodos hay genes que se sobrelapan (comparten nucleótidos entre ellos). Realizando una inspección detallada encontramos que en general no siempre son los mismos genes que se sobrelapan entre los grupos y el sobrelapamiento siempre es de unos cuantos nucleótidos. El número de genes que se sobrelapan también varía entre grupos con un rango entre cero (para Patellogastropoda) hasta 18 (para Biomphalaria glabrata) (Tabla 3).
Posición de los genes. Los genes que se codifican en la cadena ligera y la pesada varían entre los grupos, siendo más o menos similar el número de genes que se codifican en cada cadena en Octopus vulgaris, Haliotis rubra y Nerita melanotragus. Por el contrario, para Caenogastropoda, Patellogastropoda y Heterobranchia en número de genes que se codifica en la cadena ligera es más alto (Tabla 3).
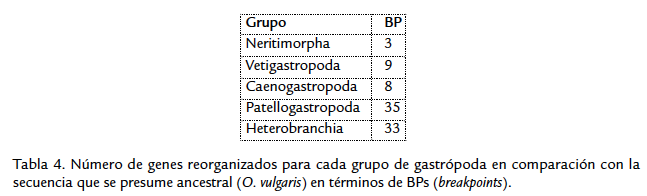
Número de reorganizaciones génicas. La tabla 4 muestra el número de BP para cada grupo en comparación con el genoma de Octopus vulgaris. Los genomas tanto de Patellogastropoda como de Heterobranchia presentan genomas muy reorganizados, con casi todos los genes en posición diferente en comparación con O. vulgaris. Los grupos Neritimorpha y Vetigastropoda son los menos reorganizados, por lo que se podría pensar que son grupos basales dentro de los gastrópodos. Los genomas mt en invertebrados parecen estar sujetos a frecuencias altas de transposición génica (Wolstenholme et al., 1987) y es posible que los altos niveles de transposición estén relacionados con altos eventos de especiación durante la evolución (Tzeng et al., 1992). Dentro de los gastrópodos, el grupo Heterobranchia es el que contiene el mayor número de especies (Ponder y Lindberg, 2008; Dinapoli y Klussmann-Kolb, 2010), además de ser evolutivamente muy diverso con representantes en hábitats marinos, dulceacuícolas y terrestres. Este hecho podría explicar el por qué de los genomas altamente reorganizados dentro de este grupo. Es importante resaltar que los otros grupos han sido muestreados pobremente, y no podemos saber si existen reorganizaciones génicas dentro del grupo Neritimorpha, Vetigastropoda o Patellogastropoda, pues tienen pocos representantes secuenciados. Aunque para Vetigastropoda hay cuatro genomas disponibles en genbank, todos pertenecen a la misma especie.
Aunque las relaciones filogenéticas entre los grupos de gastrópodos todavía no están bien definidas, un análisis reciente utilizando las secuencias tanto de nucleótidos como de proteínas de los genomas mt completos sugiere que Neritimorpha, Vetigastropoda y Caenogastropoda están cercanamente relacionados entre ellos (Castro y Colgan, 2010). La organización génica de los genomas de Vetigastropoda, Neritimorpha y Caenogastropoda, es muy parecida. Aunque el número de BP entre el genoma de O. vulgaris y Caenogastropoda es alto, haciendo una observación visual de estos genomas se puede ver que la diferencia implica solo dos eventos de reorganización génica entre ellos, cada evento con varios genes que se mueven en bloque (Ki et al., 2010).
CÁLCULO DE LAS TASAS DE EVOLUCIÓN MOLECULAR DEL GENOMA MT EN EL GRUPO
Los genomas mt de metazoos han sido de gran utilidad como marcadores filogenéticos porque contienen un set de genes bien caracterizados que varía poco entre especies. No obstante, puede haber muchas fuentes de error potencial en filogenética molecular. Por ejemplo, la señal de las ramas más ancestrales del árbol puede perderse por la saturación mutacional, o en casos en donde hay gran heterogeneidad en las tasas de evolución entre especies se pueden generar problemas por la atracción de ramas largas entre especies con rápidas tasas de evolución (Felsenstein, 1978). Algunos autores han sugerido el uso de métodos como máxima verosimilitud y métodos bayesianos para solucionar este problema (Huelsenbeck et al. 2001). Sin embargo, incluso en estos casos, es importante tener en cuenta la heterogeneidad de las secuencias para aplicar modelos de evolución adecuados que se ajusten a los datos (Chang, 1999). Adicionalmente, algunos estudios han sugerido que las tasas de evolución molecular se correlacionan con las tasas de reorganización génica (Shao et al., 2003; Xu et al., 2006). En este estudio se evaluó si existen diferencias en las tasas de evolución molecular en los genomas mt de los gastrópodos utilizando el test de Tajima, 1993. Los cálculos indicaron que en casi todos los casos las tasas de evolución molecular son diferentes entre los grupos. Hay algunos casos en los cuales la hipótesis nula (tasas de evolución iguales) no se rechaza, estos se dan en la mayoría de las comparaciones entre los genomas mitocondriales de Vetigastropoda con Caenogastropoda, y cuando se comparan el genoma de N. melanotragus (Neritimorpha) con el de H. rubra (Vetigastropoda) (Tabla 5).
CONCLUSIONES
El análisis comparativo de la diversidad estructural del genoma mitocondrial es necesario para entender su evolución. En gastrópodos vemos que aunque existen pocas diferencias en cuanto a los contenidos génicos (2 RNAs, 13 genes que codifican para proteínas y 22 RNAs de trasferencia) y los contenidos nucleotídicos y aminoacídicos de los genomas, existen diferencias entre las tasas de evolución molecular y las tasas de reorganización génica entre los grupos. Estas diferencias hacen que los genomas mt de gastrópodos sean interesantes para estudiar evolución y mecanismos de reorganización génica. Adicionalmente, estas diferencias pueden dificultar los análisis filogenéticos y deben tenerse en cuenta al momento de seleccionar modelos de evolución adecuados. A su vez, pueden servir como marcadores que corroboren hipótesis filogenéticas generadas con este marcador pero con otro tipo de aproximación, o con otros marcadores. La similitud en los genomas de los grupos Neritimorpha, Vetigastropoda y Caenogastropoda en cuanto a codones, a longitud y a número de aminoácidos, además de organización génica y tasas de evolución molecular, soportan lo encontrado por Castro y Colgan,2010, quienes sugieren una estrecha relación filogenética entre estos grupos. Se recomienda aumentar el número de genomas especialmente dentro del grupo Neritimorpha en donde hay solo un genoma mt secuenciado, con el fin de dilucidar más claramente las relaciones filogenéticas entre estos tres grupos.
AGRADECIMIENTOS
Este trabajo hace parte de los productos generados en el proyecto código 1117-489-25505 financiado por COLCIENCIAS y la Universidad del Magdalena.
BIBLIOGRAFÍA
ATTARDI G, SCHATZ G. Biogenesis of mitochondria. Annu Rev Cell Biol. 1988;4:289-333.
BANDYOPADHYAY PK, STEVENSON BJ, CADY MT, OLIVERA BM. The complete mitochondrial sequence and gene order of the cone snail Conus textile. [http://www.ncbi.nlm.nih.gov/protein/YP_001004258.1] website Direct submission to the Genome Project NCBI record NC_008797.1-2007
BANDYOPADHYAY P, STEVENSON B, CADY M, OLIVERA B, WOLSTENHOLME D. Complete mitochondrial DNA sequence of a Conoidean gastropod, Lophiotoma (Xenuroturris) cerithiformis: Gene order and gastropod phylogeny. Toxicon. 2006;48:29-43.
BLANCHETTE MT, KUNISAWA YD, SANKOFF. Gene order breakpoint evidence in animal mitochondrial phylogeny. J Mol Evol. 1999;49:193-203.
BOORE JL, BROWN WM. Big trees from little genomes: mitochondrial gene order as a phylogenetic tool. Curr Opin Genet Dev. 1998;8:668-674.
BRIDGE D, CUNNINGHAM CW, SCHIERWATER B, DESALLE R, BUSS LW. Class-level relationships in the phylum Cnidaria: Evidence from mitochondrial genome structure. Proc Natl Acad Sci U S A. 1992;89:8750-8753.
BURGER G, GRAY MW, LANG BF. Mitochondrial genomes: anything goes. Trends Genet. 2003;19(12):709-716.
CASTRO LR, COLGAN D. The phylogenetic position of Neritimorpha based on the mitochondrial genome of Nerita melanotragus (Mollusca: Gastropoda). Mol Phylogenet Evol. 2010;57:918-923.
CHANDLER EA, MCDOWELL JR, GRAVES JE. The mitochondrial genome of the rapa whelk (Rapana venosa) and the genetic variability of the mitochondrial coding regions in the neogastropods. [http://www.genome.jp/dbget-bin/www_bget?refseq:NC_011193] website Direct submission to the Genome Project NCBI record NC_011193-2008 2008.
CHANG JT. Inconsistency of evolutionary tree topology reconstruction methods when substitution rates vary across characters. Math Biosci. 1999;134:189-215.
CUNHA R, GRANDE C, ZARDOYA R. Neogastropod phylogenetic relationships based on entire mitochondrial genomes. BMC Evol Biol. 2009;9:210.
DEJONG R, EMERY A, ADEMA C. The mitochondrial genome of Biomphalaria glabrata (Gastropoda: Basommatophora), intermediate host of Schistosoma mansoni J Parasitol. 2004;90:991-997.
DINAPOLI A, KLUSSMANN KOLB A. The long way to diversity - Phylogeny and evolution of the Heterobranchia (Mollusca: Gastropoda). Mol Phylogenet Evol. 2010;55:60-76.
DOWTON M, CASTRO LR, AUSTIN AD. Mitochondrial gene rearrangements as phylogenetic characters in the invertebrates: Examination of genome ‘morphology’. Invertebr Syst. 2002;16:345-356.
FELSENSTEIN J. Cases in which parsimony or compatibility methods will be positively misleading. Syst Zool. 1978;27:401-410.
GRANDE C, TEMPLADO J, CERVERA J, ZARDOYA R. The complete mitochondrial genome of the nudibranch Roboastra europaea (Mollusca: Gastropoda) supports the monophyly of opisthobranchs. Mol Biol Evol. 2002;19(10):1672-1685.
GRANDE C, TEMPLADO J, ZARDOYA R. Evolution of gastropod mitocondrial genome arrangements. BMC Evol Biol. 2008;8:61.
GRAY MW. Origin and evolution of mitochondrial DNA. Annu Rev Cell Biol. 1989;5:25-50.
HATZOGLOU E, RODAKIS G, LECANIDOU R. Complete sequence and gene organization of the mitochondrial genome of the land snail Albinaria coerulea. Genetics. 1995;140:1353-1366.
HUELSENBECK JP, RONQUIST F, NIELSEN R, BOLLBACK JP. Bayesian inference of phylogeny and its impact on evolutionary biology. Science. 2001;294:2310-2314.
JUNG S, LEE Y, TOSHIHIRO H, CHO H, LEE J. Reishia clavigera mitochondrion, complete genome. [http://www.ncbi.nlm.nih.gov/nuccore/NC_010090] website Direct submission to the Genome Project NCBI record NC_010090-2005
KI JS, LEE YM, YUNG SO, HORIGUSHI T, CHO HS, LEE JS. Mitochondrial genome of Thais clavigera (Mollusca: Gastropoda): Affirmation of the conserved, ancestral gene pattern within the mollusks. Mol Phylogenet Evol. 2010;54:1016-1020.
KNUDSEN B, KOHN A, NAHIR B, MCFADDEN C, MOROZ L. Complete DNA sequence of the mitochondrial genome of the sea-slug, Aplysia californica: Conservation of the gene order in Euthyneura. Mol. Phylogenet. Evol. 2006;38:459-469.
KURABAYASHI A, UESHIMA R. Complete sequence of the mitochondrial DNA of the primitive opisthobranch gastropod Pupa strigosa: Systematic implication of the genome organization. Mol. Biol. Evol. 2000;17(2):266-277.
LIN YC, LU CL, LIU YI-C TANG CY. Spring: A tool for the analysis of genome rearrangement using reversals and block-interchanges. Nucleic Acids Res. 2006;34:696-699.
MAYNARD B, KERR L, MCKIERNAN J, JANSEN E, HANNA P. Mitochondrial DNA sequence and gene organization in Australian backup abalone Haliotis rubra (Leach). Mar Bio Technol. 2005;7:645-658.
MEDINA M, LAL S, VALLES Y, TAKAOKA T, DAYRAT B, BOORE J, et al. Crawling through time: Transition of snails to slugs dating back to the Paleozoic, based on mitochondrial phylogenomics. Mar Genomics. 2011;4:51-59.
NOSEK J, TOMÁSKA L. Mitochondrial genome diversity: evolution of the molecular architecture and replication strategy. Curr Genet. 2003;44:73-84.
OKIMOTO R, MACFARLANE JL, CLAY DO, WOLSTENHOLME DR. The mitochondrial genomes of two nematodes, Caenorhabditis elegans and Ascaris suum. Genetics. 1992;130:471-498.
PONDER WF, LINDBERG DR. Phylogeny and evolution of the Mollusca. Berkeley: University of California Press; 2008.
REN J, SHEN X, LIU X. Sequence analysis in the mitochondrial genome of the Pacific abalone Haliotis discus hannai (Gastropoda, Haliotidae) and exploitation of its application. [http://www.ncbi.nlm.nih.gov/nuccore/EU595789] website Direct submission to the Genome Project NCBI record EU595789-2009.
RUMPHO M, WORFUL J, LEE J, KANNAN K, TYLER M, BHATTACHARYA D, et al. Horizontal gene transfer of the algal nuclear gene psbO to the photosynthetic sea slug Elysia chlorotica. Proc Natl Acad Sci USA. 2008;105:17867-17871.
SHAO R, DOWTON M, MURRELL A, BARKER SC. Rates of genome rearrangement and nucleotide substitution are correlated in the mitochondrial genomes of insects. Mol Biol Evol. 2003;20:1612-1610.
SIMISON W, LINDBERG D, BOORE J. Rolling circle amplification of metazoan mitochondrial genomes. Mol Phylogenet Evol. 2006;39:562-567.
STUART JA, BROWN MF. Mitochodrial DNA maintanance and bioenergetics. Biochim Biophys Acta. 2006;1757:79-89.
TAANMAN JW. The mitochondrial genome: structure, transcription, translation and replication. Biochim Biophys Acta. 1999;1410:103-123.
TAJIMA F. Simple methods for testing the molecular clock hypothesis. Genetics. 1993;125:599-607.
TAMURA K, DUDLEY J, NEI M, KUMAR S. MEGA 4: Molecular evolutionary genetics analysis (MEGA) software versión 4. Mol Biol Evol. 2007;24:1596-1599.
THOMPSON JD, GIBSON TJ, PLEWNIAK F, JEANMOUGIN F, HIGGINS DG. The CLUSTAL X Windows interface. Flexible strategies for multiple sequence aligment aided by quality analysis tools. Nucl. Acid Res. 1997;25:4876-4882.
TZENG CS, HUI CF, SHEN SC, HUANG PC. The complete nucleotide sequence of the Crossostomalacustre mitochondrial genome: conservation and variations among vertebrates. Nucleic Acids Res. 1992;20:4853-4858.
VANWORMHOUDT A, LEBRAS Y, HUCHETTE S. Haliotis marmorata from Senegal; a sister species of Haliotis tuberculata: Morphological and molecular evidence Biochem. Syst Ecol. 2009;37:747-755.
WEI L, SHEN H, CHEN C. Analysis of complete mitochondrial genome of one kind of Onchidiidae (Mollusca: Pulmonata). [http://www.ncbi.nlm.nih.gov/nuccore/NC_013934.1] website Direct submission to the Genome Project NCBI record NC_013934.1-2012.
WHITE T, CONRAD M, TSENG R, BALAYAN S, GOLDING R, DE FRIAS MARTINS A, et al. Ten new complete mitochondrial genomes of pulmonates (Mollusca: Gastropoda) and their impact on phylogenetic relationships. BMC Evol Biol 2011;11:295.
WOLSTENHOLME DR, MACFARLANE JL, OKIMOTO R, CLARY DO, WAHLEITHNER JA. Bizarre tRNAs inferred from DNA sequences of mitochondrial genomes of nematode worms. Proc Natl Acad Sci U S A. 1987;84:1324-1328.
XIN Y, REN J, LIU X. Mitogenome of the small abalone Haliotis diversicolor Reeve and phylogenetic analysis within Gastropoda. Mar Genomics. 2011;4:253-262.
XU W, JAMESON D, TANG B, HIGGS PG. The Relationship between the rate of molecular evolution and the rate of genome rearrangement in animal mitochondrial genomes. Mol Evol. 2006;63:375-392.
YAMAZAKI N, UESHIMA R, TERRETT J, YOKOBORI S, KAIFU M, SEGAWA R, et al. Evolution of pulmonate gastropod mitochondrial genomes: comparisons of gene organizations of Euhadra, Cepaea and Albinaria and implications of unusual tRNA secondary structures. Genetics. 1997;145:749-758.
ZHAO Q, ZHANG S, DENG Z, JIANG M, NIE P. Conservation and variation in mitochondrial genomes of gastropods Oncomelania hupensis and Tricula hortensis, intermediate host snails of Schistosoma in China. Mol Phylogenet Evol. 2010;57(1):215-226.
Cómo citar
APA
ACM
ACS
ABNT
Chicago
Harvard
IEEE
MLA
Turabian
Vancouver
Descargar cita
Visitas a la página del resumen del artículo
Descargas
Licencia
Derechos de autor 2012 Acta Biológica Colombiana

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
1. La aceptación de manuscritos por parte de la revista implicará, además de su edición electrónica de acceso abierto bajo licencia Attribution-NonCommercial-ShareAlike 4.0 (CC BY NC SA), la inclusión y difusión del texto completo a través del repositorio institucional de la Universidad Nacional de Colombia y en todas aquellas bases de datos especializadas que el editor considere adecuadas para su indización con miras a incrementar la visibilidad de la revista.
2. Acta Biológica Colombiana permite a los autores archivar, descargar y compartir, la versión final publicada, así como las versiones pre-print y post-print incluyendo un encabezado con la referencia bibliográfica del articulo publicado.
3. Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
4. Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos institucionales, en su página web o en redes sociales cientificas como Academia, Researchgate; Mendelay) lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).